还在没日没夜跑定量?教你一招不做实验也能预测基因表达水平的小妙招!
丁香学术
RT-qPCR、转录组或者蛋白质组是实验室常用的表征基因的表达水平的方法,然而你知不知道其实不做实验也可以预测目标基因的表达水平呢?
测序技术的高速发展使数据库中可获得的 DNA 序列大量增长,基于这些海量的数据,科学家们进行了一些与 DNA 组成相关的统计分析,如密码子的使用情况。很快科学家就发现物种的基因之间存在相当大的密码子使用异质性,并且密码子偏好性的程度与基因的表达量成正相关。
基因在物种之间存在的密码子使用偏好差异可以用密码子适应指数 (Codon Adaptation Index,CAI) 进行表征量化,密码子偏好的程度呈正相关。CAI 由 Sharp 等人提出,它测量目标核酸序列和已知高度表达参考基因集之间的同义密码子使用偏好性的差异程度。
CAI 值的计算也十分简便,那接下来就一起跟着我来用 CAIcal 工具预测一下你感兴趣的基因表达水平如何吧。
CAIcal 是一款在线的用于评估密码子使用适应性的组合工具。
CAIcal 使用详解
1. 打开 CAIcal 服务器主页,点击 Enter 进入 CAI calculation 界面。

计算 CAI 要输入查询序列(Query sequence)、参考集及用于翻译的密码子规则。查询序列必须是 fasta 格式的 DNA 或 RNA 序列。
CAIcal 服务器会首先检查查询序列是否是编码蛋白质的 DNA 或 RNA 区域,以及序列是否包含内部终止密码子 (通常表示基因发生了移码突变)、是否存在非标准字符,以及序列是否编码氨基酸。
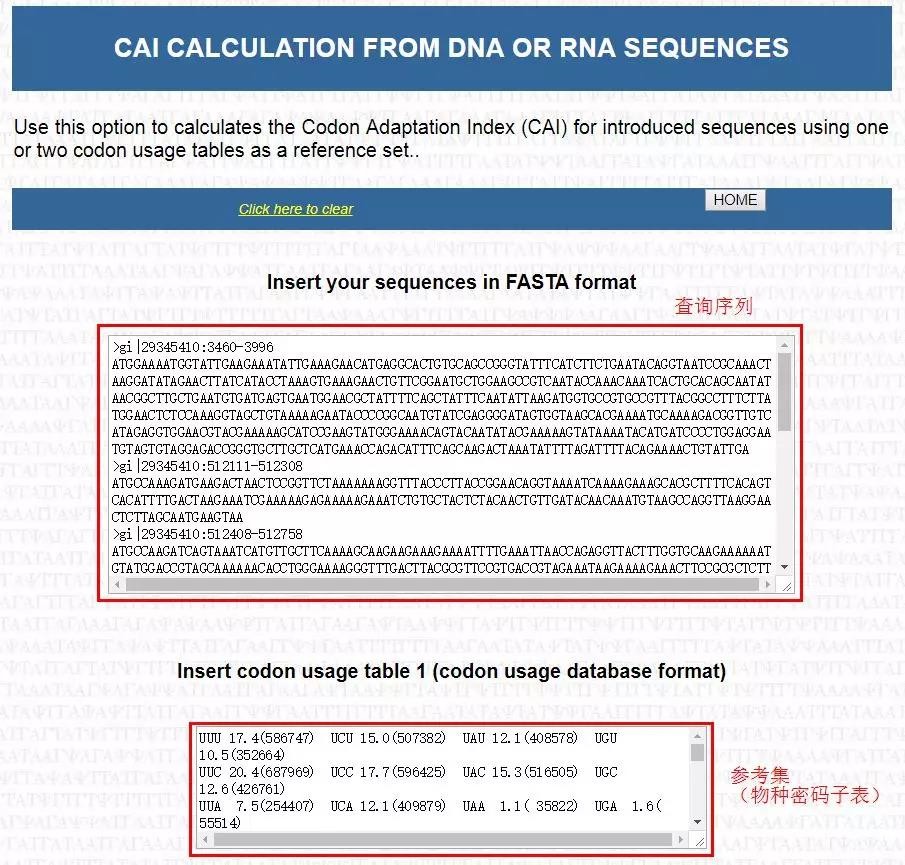
2. 计算 CAI 所需的参考集可以从密码子使用数据库中导入,可以同时使用两个参考集,比较关于两种不同生物的密码子使用,并检查它是否更适合其中一种。
CAIcal 界面中提供了此数据库的链接。该数据库包含从 GenBank 中获取并按物种的密码子使用表。

3. 选择翻译的遗传密码规则(默认为细菌的遗传密码)。然后提交任务。
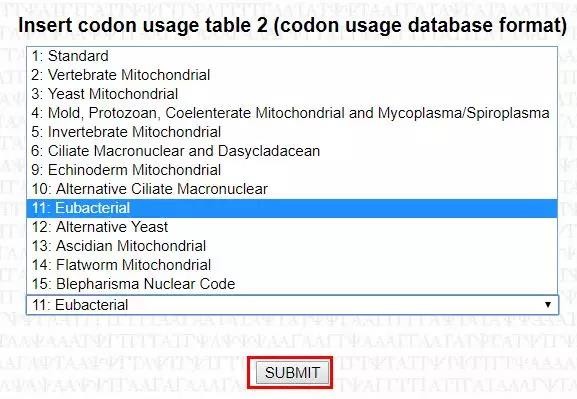
4. 运行结束后,该服务器会提供许多有用的计算结果。服务器会生成几个表格和图形以及文本框,其中包含以制表符分隔格式的结果,可以很容易地将它们复制并粘贴到电子表格中:
输出结果
1. DNA 序列或参考序列组的 CAI 值。该指数可测量由用户选择的同义密码子使用参考集基因序列集的适用性。
2. 输入 DNA 序列的同义密码子的使用情况,以及其他有用参数,如长度、总 G + C 含量、三个密码子位置的 G + C 含量,以及有效密码子的数量和密码子权重。

3. CAI 的预期值:通过计算查询序列的氨基酸组成和 G + C 含量从查询序列中随机产生 500 个序列来确定 CAI 的预期值(eCAI)。
因此,该预期 CAI 提供了,用于辨别 CAI 值的差异是否具有统计学意义的直接阈值,可以用来判断这种差异是源于密码子偏好或者仅仅是由 G + C 组合物和 / 或氨基酸组成的内部偏差引起的假象。

4. 计算出的每个密码子的权重,即密码子使用的频率与参考集中该氨基酸的最佳密码子的使用频率之比,可以使用滑动窗口沿着 DNA 序列直观可视化 CAI 的变化。

其他功能
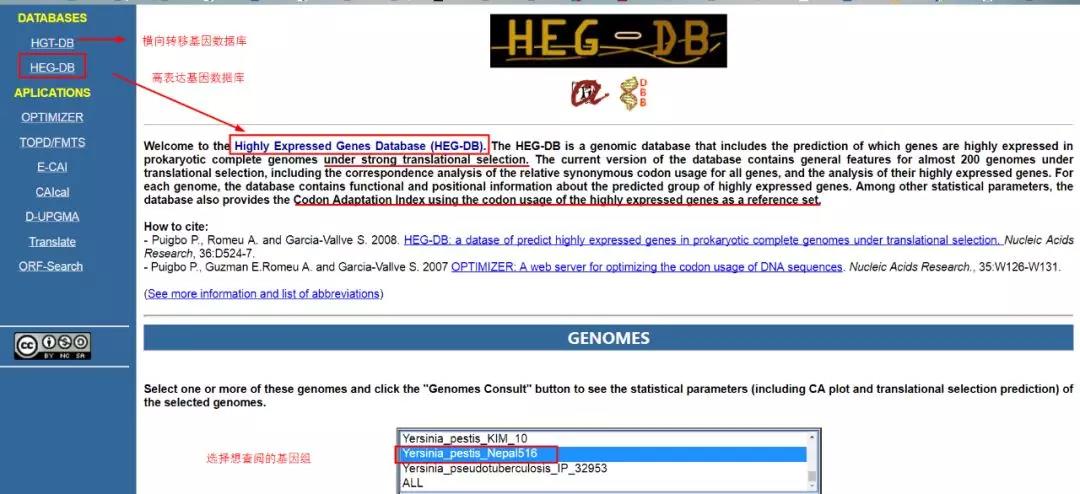
在 CAIcal 服务器主页面的左侧,还提供了几个访问其他相关程序或数据库的链接,包括 HEG-DB(高表达基因数据库)、HGT-DB(横向转移基因数据库),NCBI、Swiss-Prot 和 SGDB 数据库,基因预测、翻译、OPTIMIZER 密码子优化程序等,十分方便。
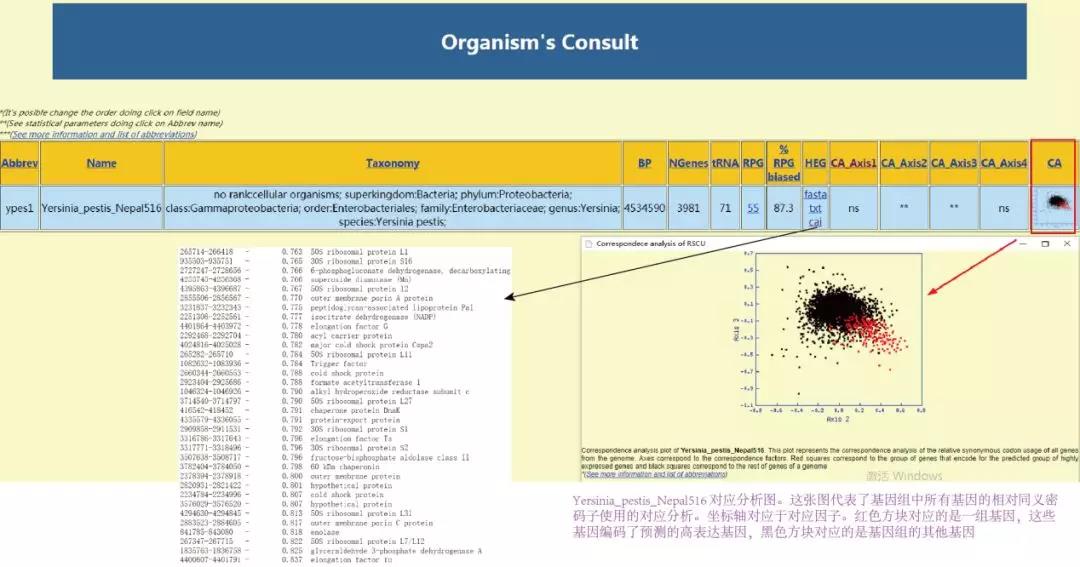
1. HEG-DB 数据库收集了模式微生物的基因组中的高表达基因,并计算了它们的 CAI 值。
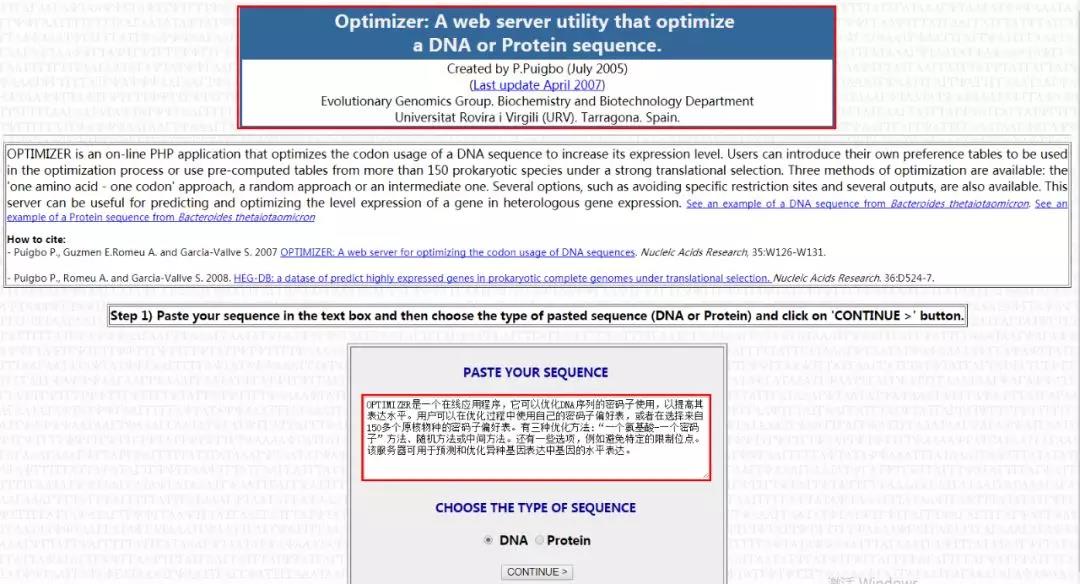
2. 在线程序 OPTIMIZER 可用于预测和优化异源基因表达中基因的水平表达。具体来说,它可以优化 DNA 序列的密码子使用,以提高其表达水平。用户可以在优化过程中使用自己的密码子偏好表,或者在选择来自 150 多个原核物种的密码子偏好表。
有三种优化方法,包括:氨基酸 - 密码子对应法、随机方法或中间法。还有一些选项,例如选择避开改变特定的限制位点等。
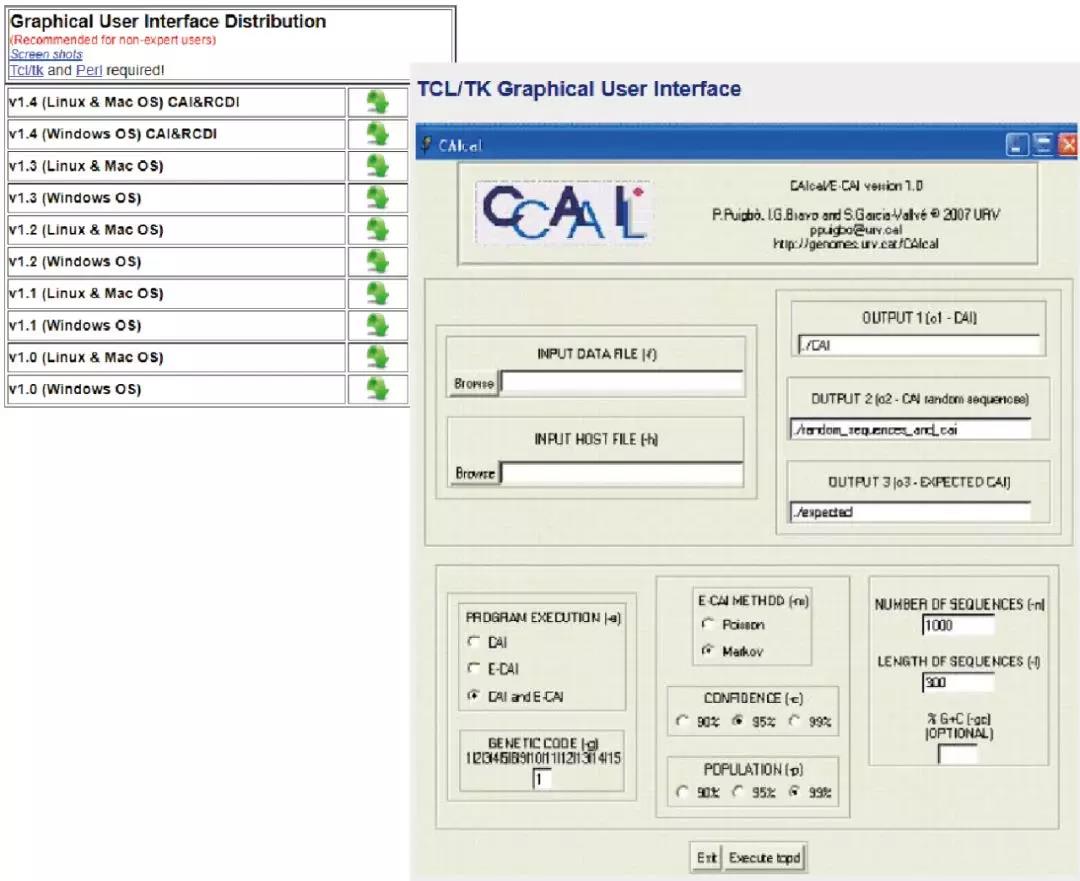
3. 此外,还可以将 CAIcal 中包含的工具(图形界面)下载并本地化,并且可以轻松地在全基因组范围内计算上千条序列的 CAI 和 eCAI 值。
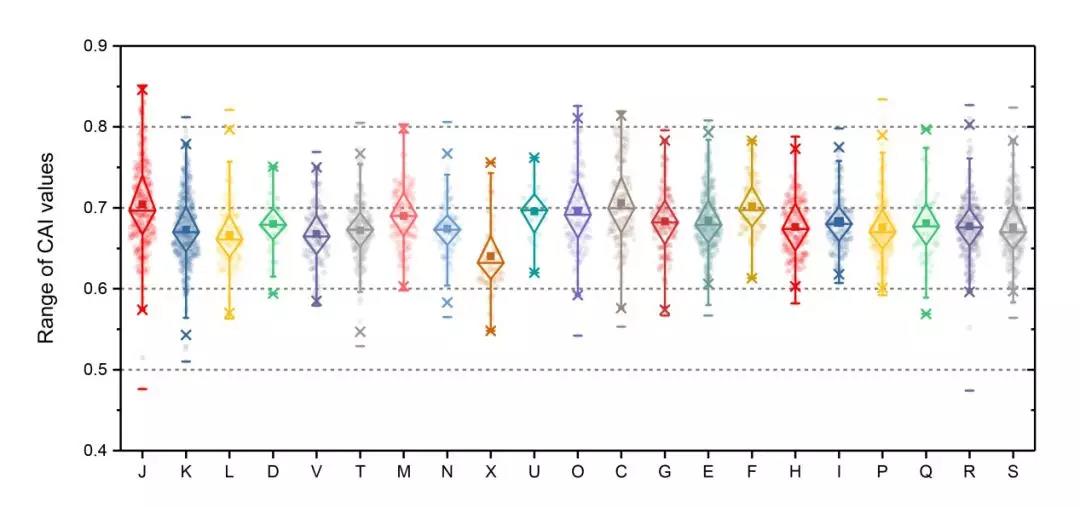
最后,放上自己整理 CAIcal 服务器的数据做的 CAI 值箱线图:某菌株全基因组范围内基因序列的 CAI 值范围的可视化(基于 COG 功能分类)。
参考文献:
[1] Puigbo P, Bravo IG and Garcia-Vallve S. (2008) CAIcal: a combined set of tools to assess codon usage adaptation. Biology Direct, 3:38.
[2] Puigbo P., Guzmen E.Romeu A. and Garcia-Vallve S. 2007 OPTIMIZER: A web server for optimizing the codon usage of DNA sequences. Nucleic Acids Research, 35:W126 -W131.
[3] Puigbo P., Romeu A. and Garcia-Vallve S. 2008. HEG-DB: a datase of predict highly expressed genes in prokaryotic complete genomes under translational selection. Nucleic Acids Research. 36:D524 - 7.
CAIcal 主页:http://genomes.urv.es/CAIcal/