零代码实现完整的GEO数据分析、多个 GEO 数据整合分析!
ZSCI
今天给大家推荐了一个专门针对 GEO 数据分析的综合在线工具--BART,这个工具最近发表在 BMC Bioinformatics 上,是一个从 CELL 或 expression 表达矩阵输入到可视化分析、富集分析的综合分析平台。
对于那些生信还没入门的同学来说,该工具的价值更加明显,可以帮助没有生信经验的同学们更完整的做 GEO 数据分析,甚至是多个 GEO 数据的整合分析。
一、简单介绍
1、BART 优劣分析:
① 功能全面:包括 GEO 数据分析的批次效应矫正、差异分析、火山图、聚类图、PCA 图、富集分析;
② 分析速度快,亲测过网页版分析速度,基本 10s 内出结果;
③ 操作简单,交互逻辑清楚,容易上手,非常适合新手;
④ 有可多种可视化结果图片,可以直接搜索差异基因,所有结果可下载;
⑤ 不足之处:火山图、PCA 图部分不能个性化调整参数,后续有需求可以从该平台下载的标准化表达矩阵或者差异分析结果用 R 进行作图分析。BART 只采用 LIMMA 作为差异分析工具,二代高通量测序的数据并不适合用 BART 进行分析。
2、BART 总流程图如下图所示,依次流程为:
数据输入→分组和批次→标准化→差异分析→富集分析。

(图片来源于生信草堂)
3、BART 有网络版和本地版两种:
①网络版:bart.salk.edu(本文举例是用的网页版)
②本地版:https://bitbucket.org/Luisa_amaral/bart
4、BART 输入数据有两大类:
①GSE ID,对应着 CEL 原始数据和 expression 表达数据两种输入数据类型,BART 会自动根据 GSE ID 搜索对应的数据并进行下载和使用;
②用户自定义的芯片表达数据,数据上传后,BART 会首先对表达矩阵进行 log2 转换并做标准化,生成一个可以直接搜索基因表达的表格。
二、GEO 数据分析
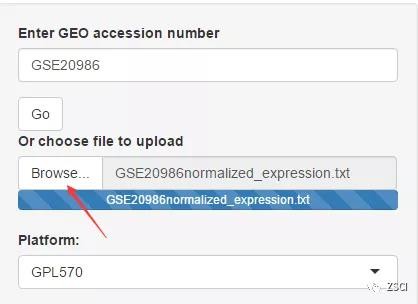
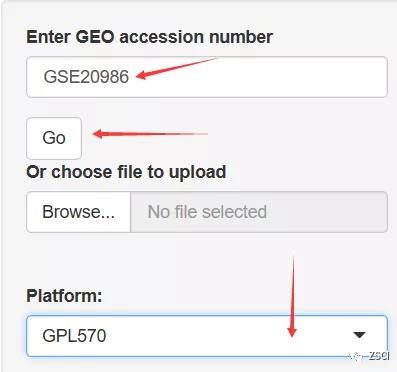
1、进入左侧面板输入 GSE 号,点击 Go 开始下载并分析该 GEO 数据,几分钟后弹出 Platform 信息。
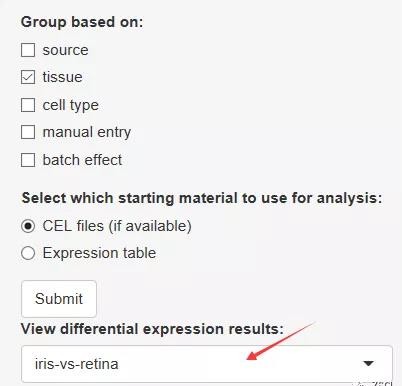
2、我们根据「Sample grouping」中的分组信息在「Group based on」中选择分组依据,接下来,选择原始 CEL 文件或表达数据(BART 会通知 CEL 文件是否可用),点击 Submit。
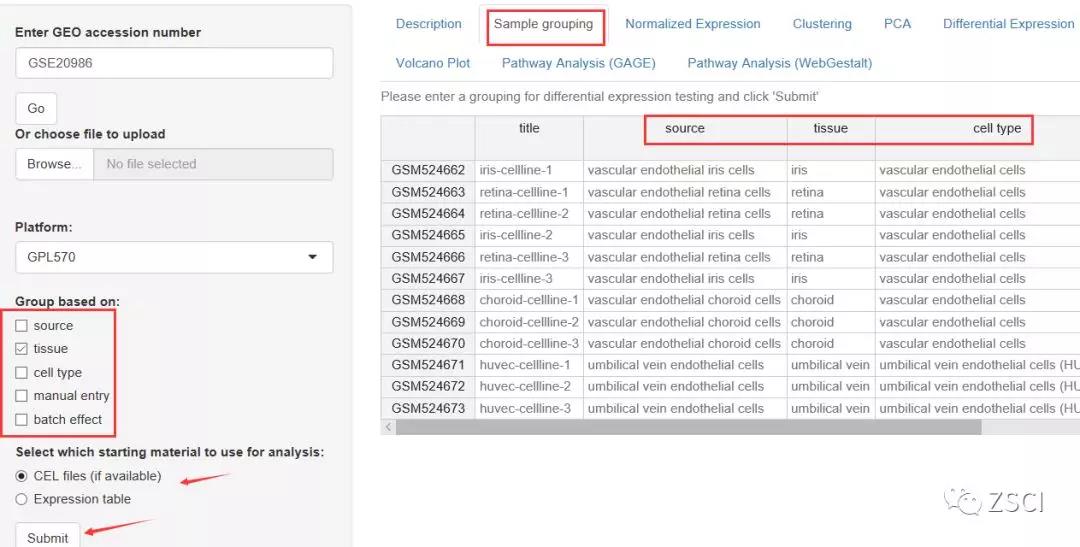
3、我们也可以选择手动输入和批次效应矫正分组。(观察 PCA 图和热图后再确定是否有必要进行批次效应矫正)
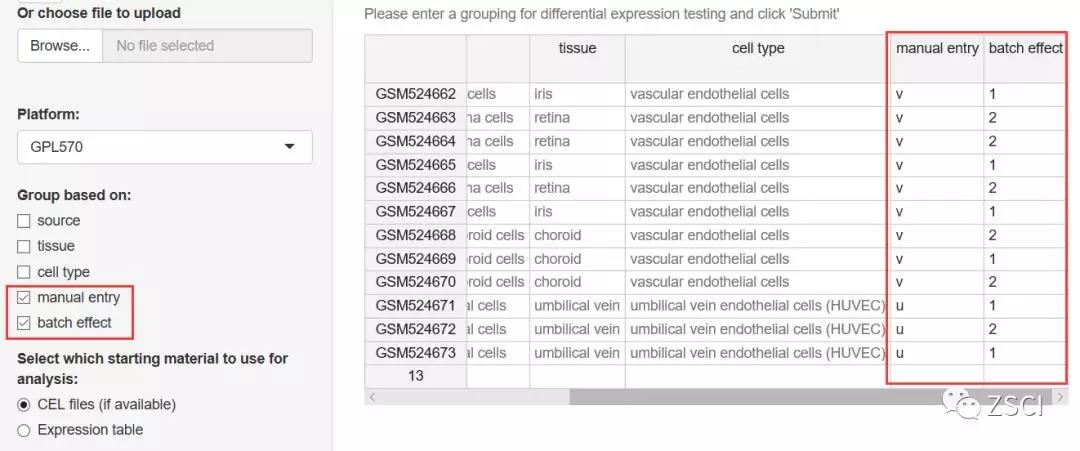
4、在 View differential expression results 部分可选择想要的差异分组。
三、GEO 数据分析结果
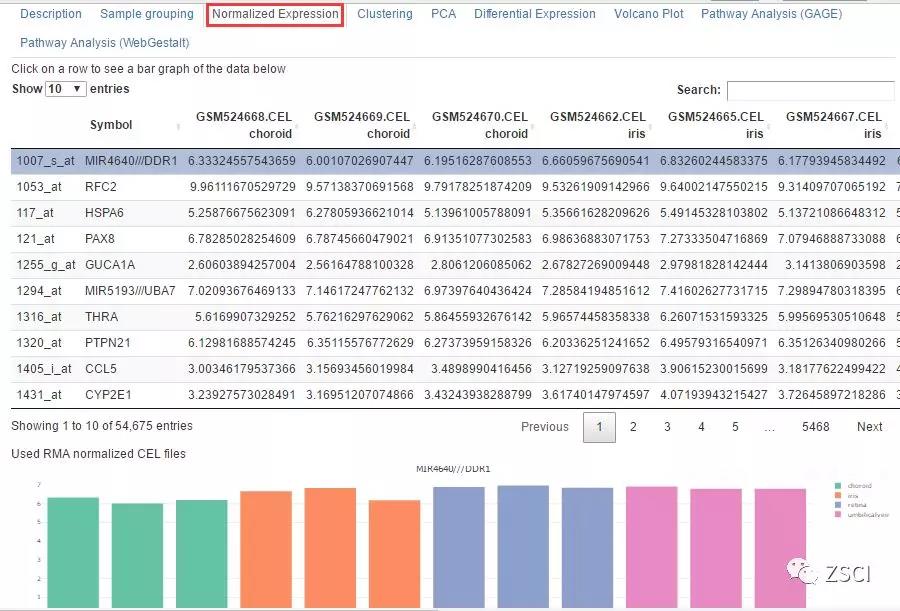
1、Normalized Expression 选项卡中,可以搜索感兴趣的「基因」或「探针 ID」。
单击表中的某一行,在表格下面可显示每个样本中标准化的基因表达。
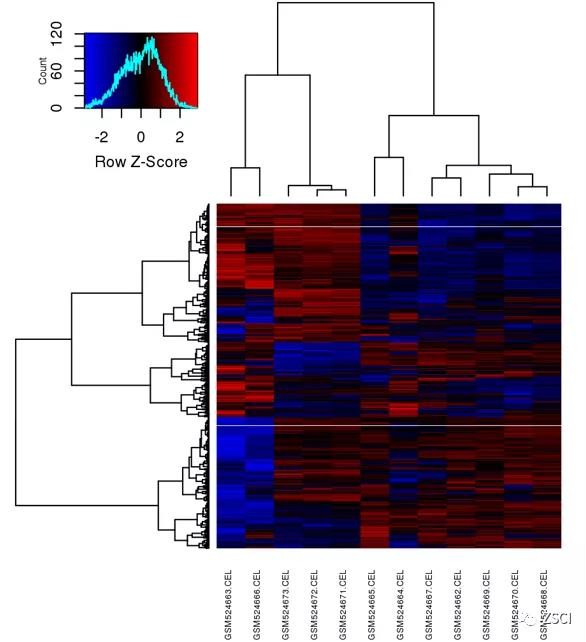
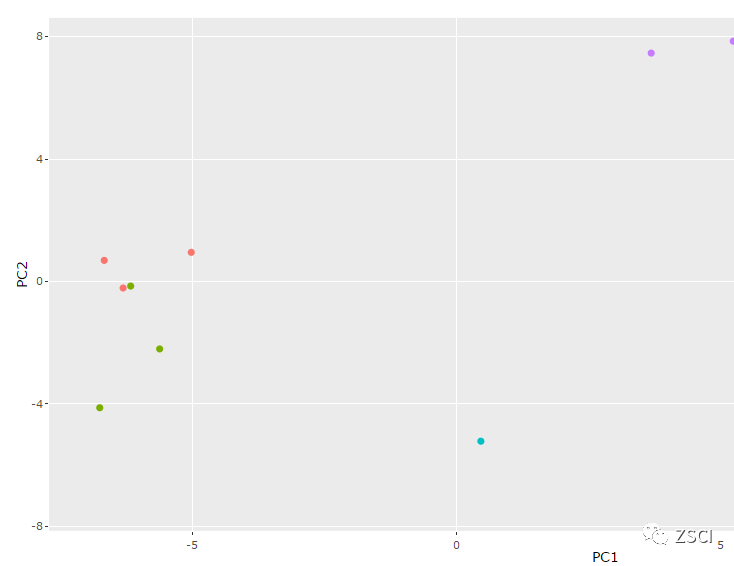
2、选择 Clustering、PCA 选项卡,可显示聚类图和 PCA 图。
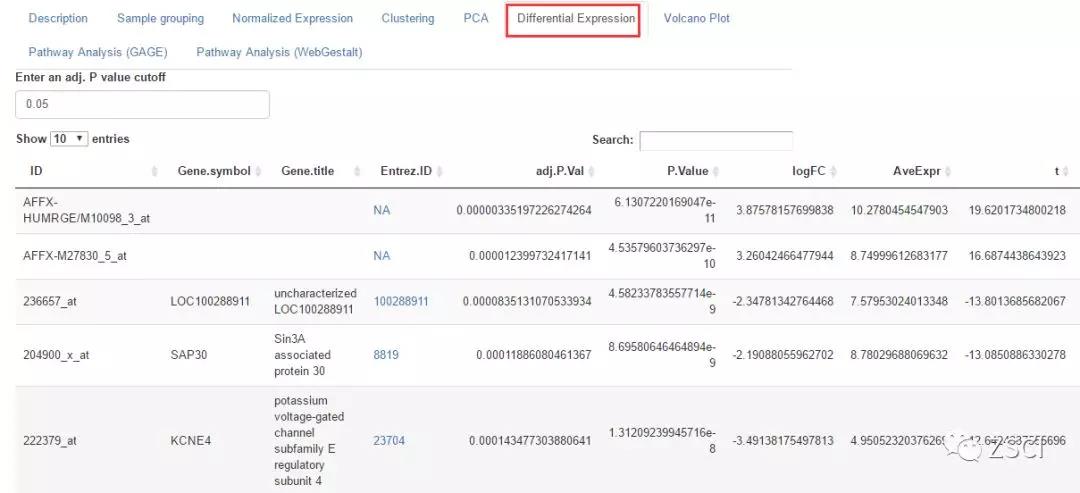
3、选择 Differential Expression 选项卡,可显示所有矫正后 P 值小于 0.05 的基因,在搜索框里可以搜索感兴趣的基因。
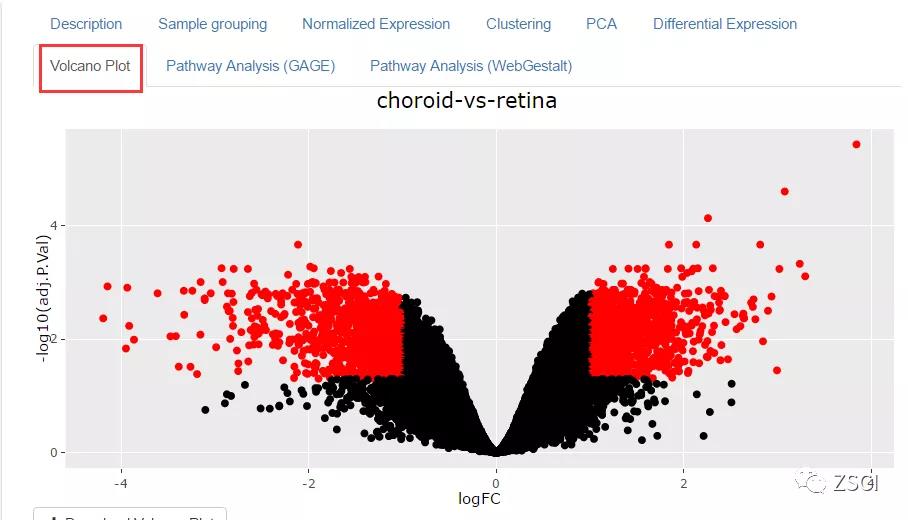
4、点击 Volcano Plot 选项卡,可显示差异基因火山图。
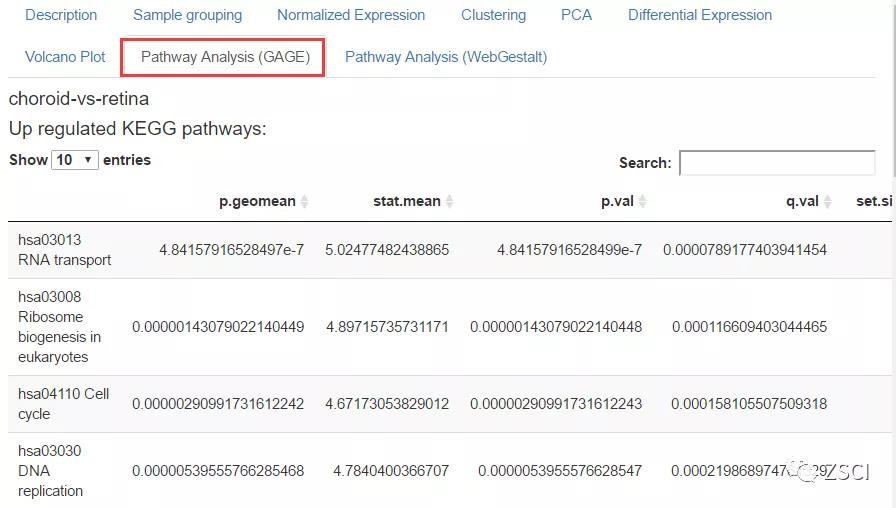
5、GAGE 富集分析
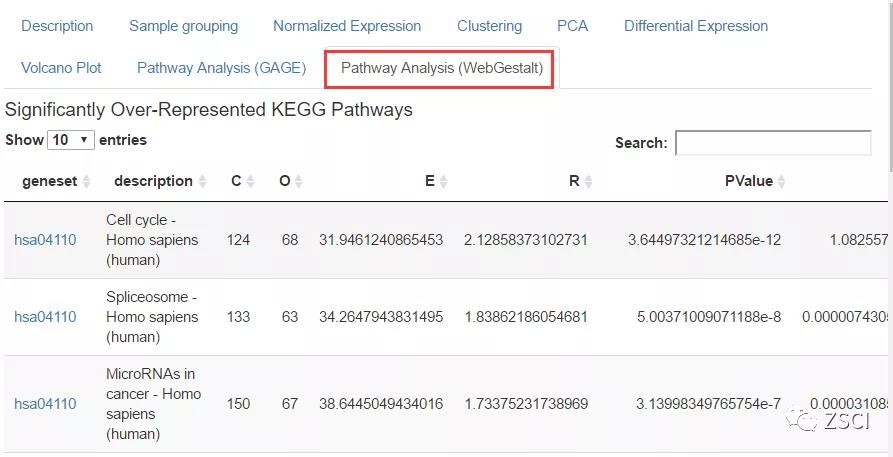
6、WebGestalt 富集分析
四、自定义芯片数据分析
1、按照 BART 示例上的格式处理要输入的文件。

2、在 Browse 选择本地的 txt 文件上传。