零代码下载TCGA数据库数据
ZSCI
TCGA 数据库目前是科研中最常用的数据库之一,其中储存着多种疾病的各组学的数据,借助该数据库,帮助了很多研究生们发表了自己的文章。
一、基于 TCGA 官网下载 RNAseq 数据
1、TCGA 数据库简介
TCGA 数据库全称为 The Cancer Genome Atlas,主要储存关于各类肿瘤的一个基本信息,包括 RNAseq,miRNAseq,DNA 甲基化,CNV,SNP 等信息,它是目前为止可以获得的公开数据库里面数据相对全面的一个,在各个领域得到了广泛的应用,为肿瘤基础医学和转化医学研究者提供了海量的基因组数据和与其关联的临床数据, 这为挖掘有意义的基因组变化和发现影响肿瘤起始、发展、分化、转移等生物学机制提供了海量数据基础。
2、下载数据
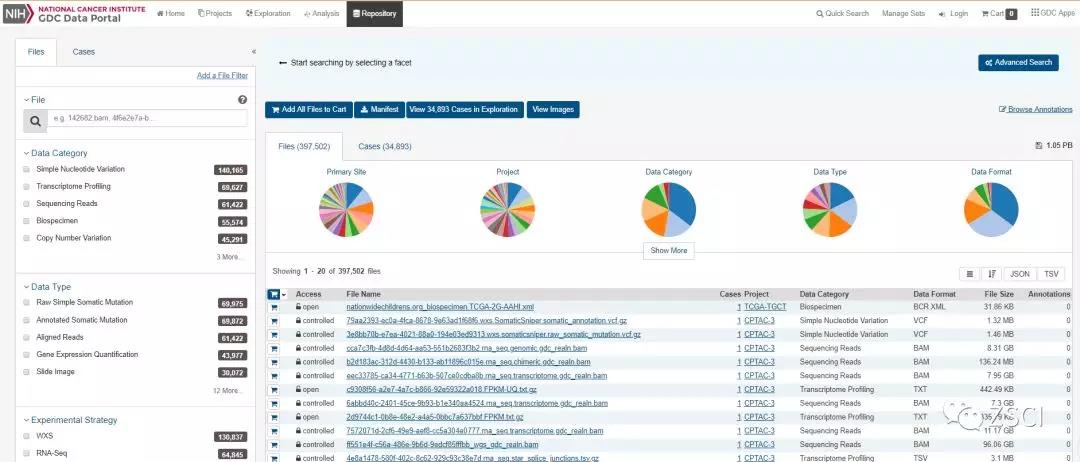
2.1、进入 TCGA 网址,点击 Repository,进入数据存储地。
网址:https://portal.gdc.cancer.gov/
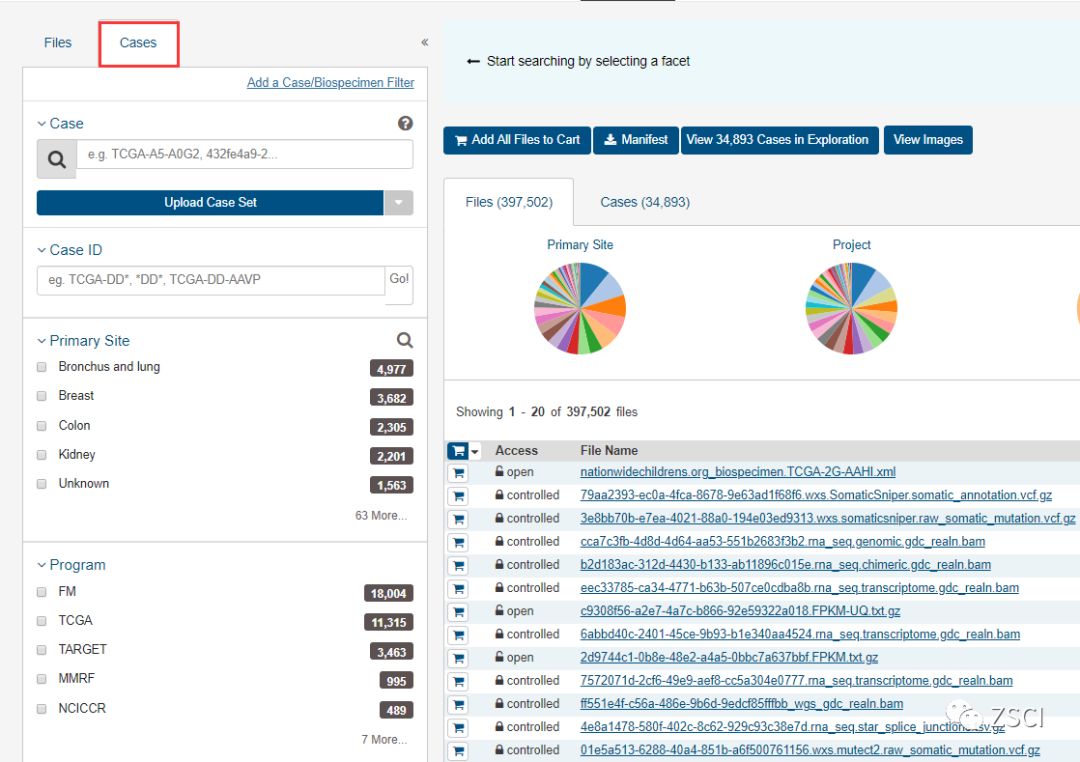
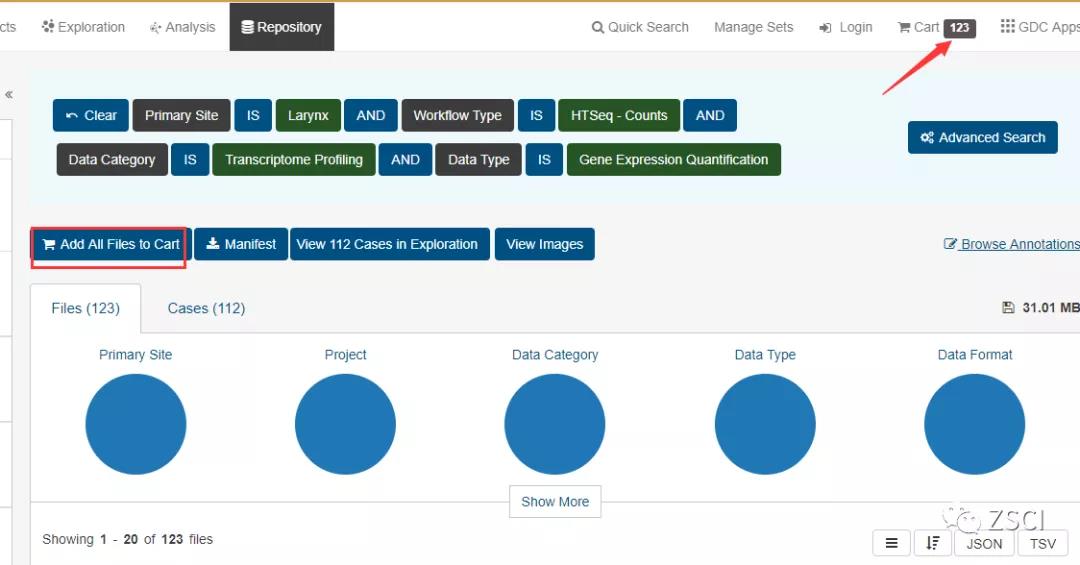
2.2、点击 Case,选择疾病类型,比如选择喉癌(Larynx),在前面打钩即可。

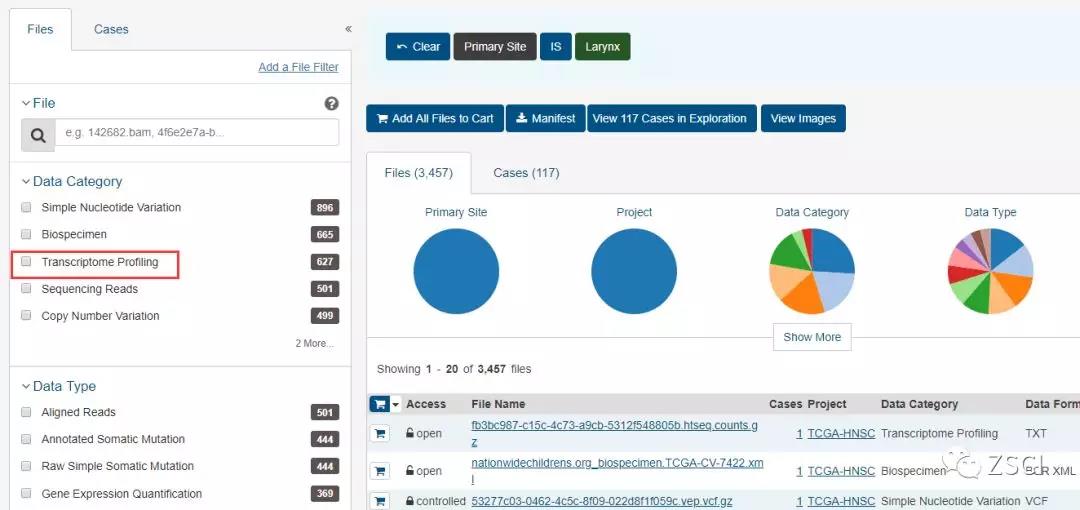
2.3、接着点击 File,选择需要下载文件的数据类型,因为需要下载的是 RNAseq 数据,所以在 Transcriptome Profiling 前面打钩。
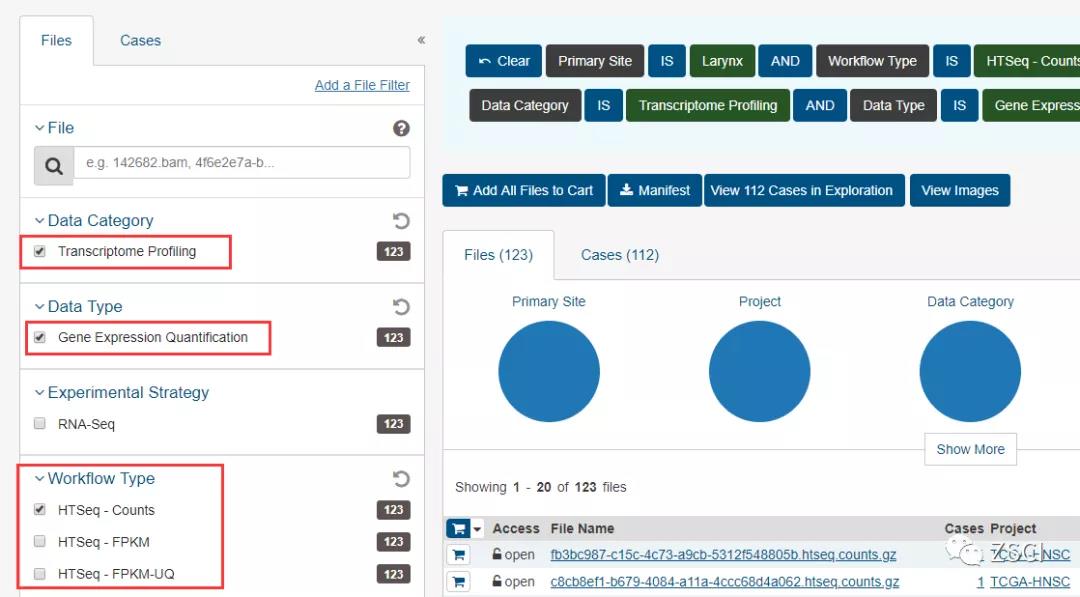
2.4、在 Data Type 选择 Gene Expression Quantification,在 Experimental Strategy 上由于只有一个选项,所以不用选择,主要看到有三种类型,分别是 counts,FPKM 和 FPKM-UQ,分别表示 counts 数和两种归一化后的值,推荐下载 counts 数据。
2.5、点击 counts 之后,可以看到每一步操作都会记录在里面,也可以看到数据大小是 31 兆。

2.6、点击 Add All Files to Cart,然后就会看到箭头所指的地方,变为相应的样本数,接着点击 cart,如下:
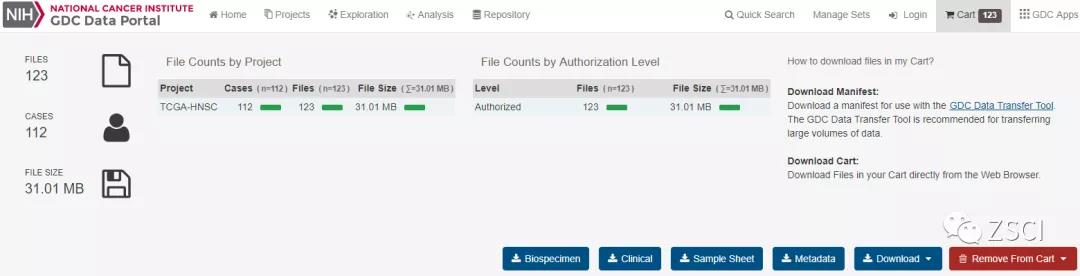
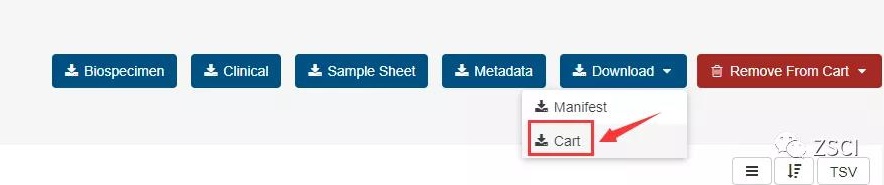
2.7、点击 download,下载 cart 即可。
二、基于 UCSC-XENA 下载 TCGA 的 RNAseq 数据
1、进入 UCSC-XENA
网站:https://xena.ucsc.edu/

注:黄色框框是官网给的如何使用该网址的教程,红色框框是需要点击进入的界面。
2、点击 launch Xena,出现下面的界面,那数据藏在哪里了呢?
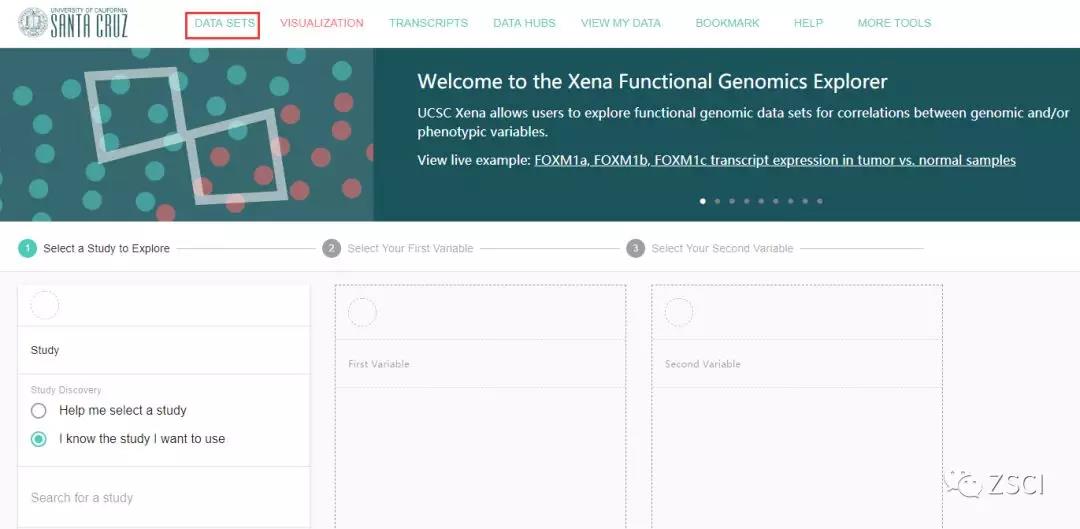
3、点击 DATA Sets 进入数据存储站。
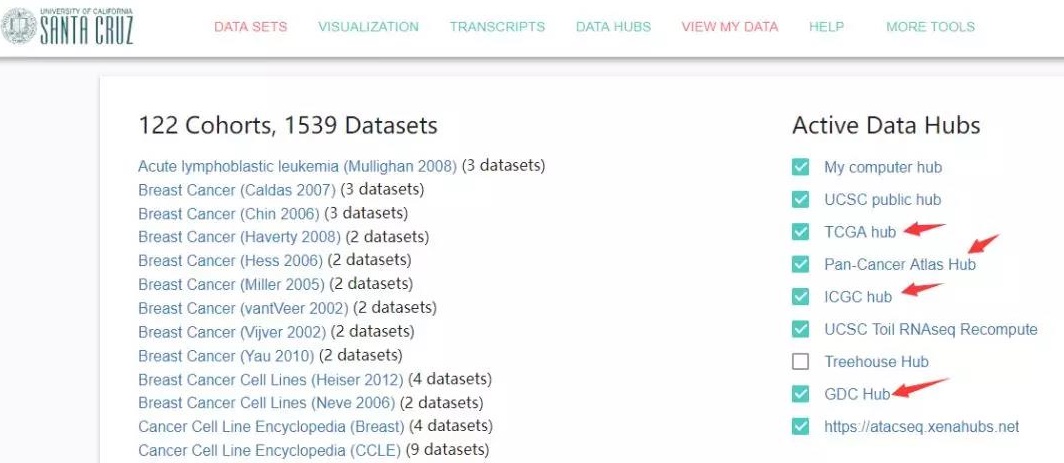
注:可以看到在右侧的 Active Data Hubs 包括了很多,不仅涵盖了 TCGA 还包括 ICGA,Pan-Cancer Atlas Hub 等数据节点。因为是研究 TCGA,只需要选中 TCGA 就可以了。
4、选中 TCGA,进入 TCGA 数据站,其包含的数据主要是下面 38 个。

5、随便选择一个癌症,比如选择第一个 AML,点击进去,出现下面的界面。
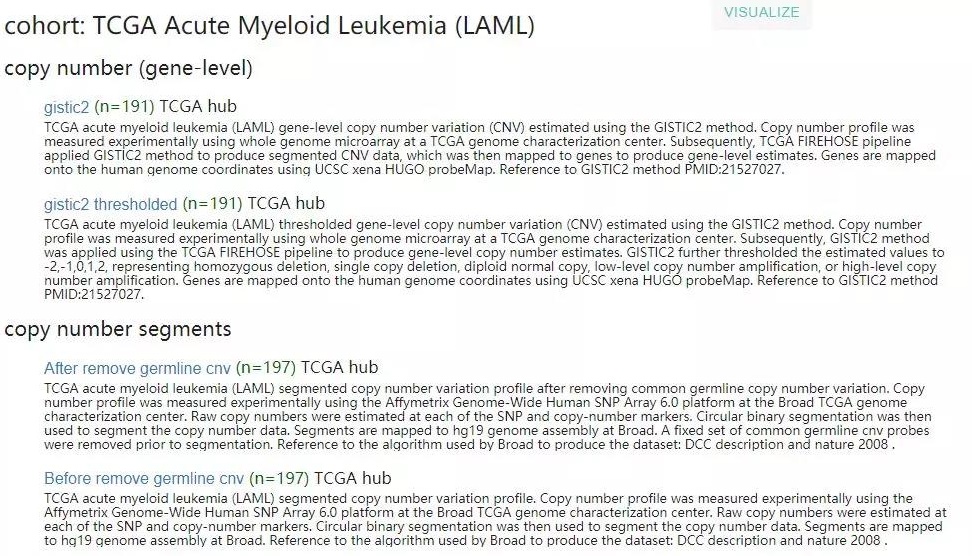
注:发现该网站已经整理好了各类数据,包括 CNV,DNA 甲基化等,方便用户下载。接着找到 RNAseq 数据,注意有两个,一个是 exon expression RNAseq,一个是 gene expression RNAseq。
6、选择 gene expression RNAseq,点击带有号的数据,进入。

7、点击之后,进入下面的界面,点击红色框内链接下载数据。
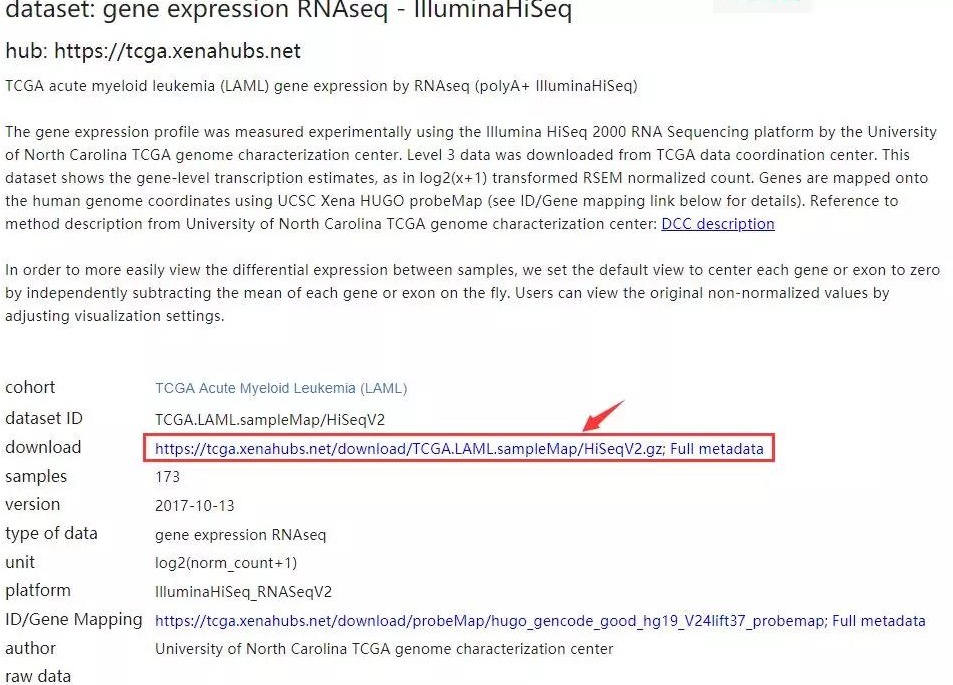
注:它的单位是 log2(count+ 1),说明这个网站是对 count 数进行了这种方式的处理来归一化的,不是 FPKM哦 。
三、通过 cBioportal 下载 TCGA 的 RNAseq 数据及临床信息
1、进入 cBioportal
网站:http://www.cbioportal.org/index.do
2、先点击 Query,进入该界面。
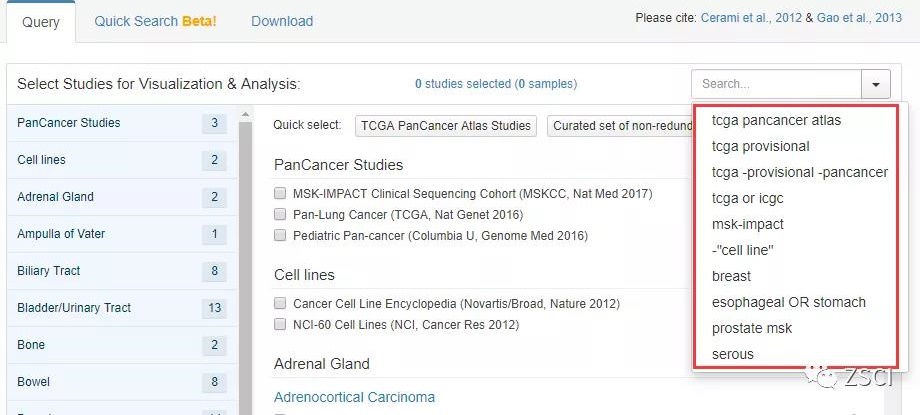
3、点击 TCGA provisional,进入 TCGA 数据站,会出现下面的界面。
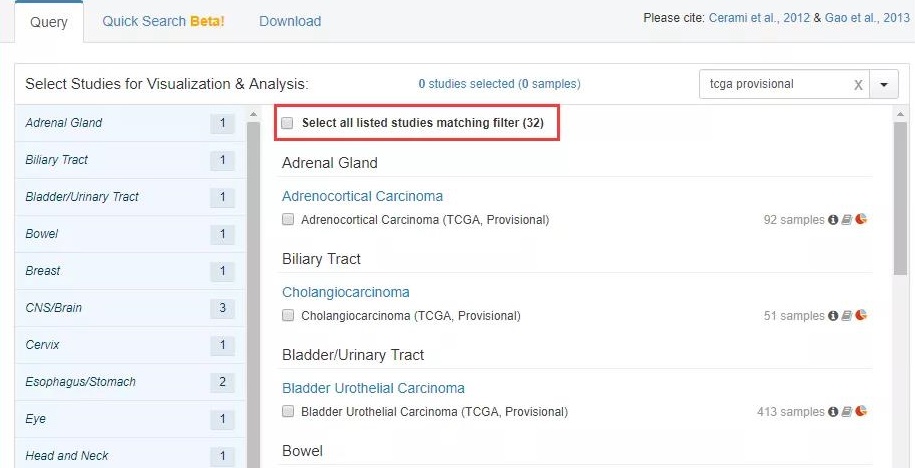
注:可以看到一共有 32 个 TCGA 数据集。
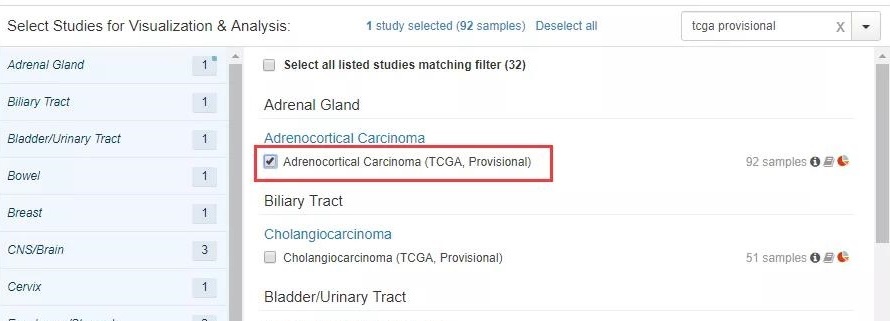
4、点击第一个肾上腺皮质癌,点击进去, 点击 view summary

5、出现下面的界面

注:可以看到主要是包括了 CNV 的信息和 Matution 信息,Mutation 按照突变频率进行了排序,可以看到在肾上腺皮质癌中 ERCC2 突变频率最高,CNV 主要涵盖了两类分别是 del 和 amp。
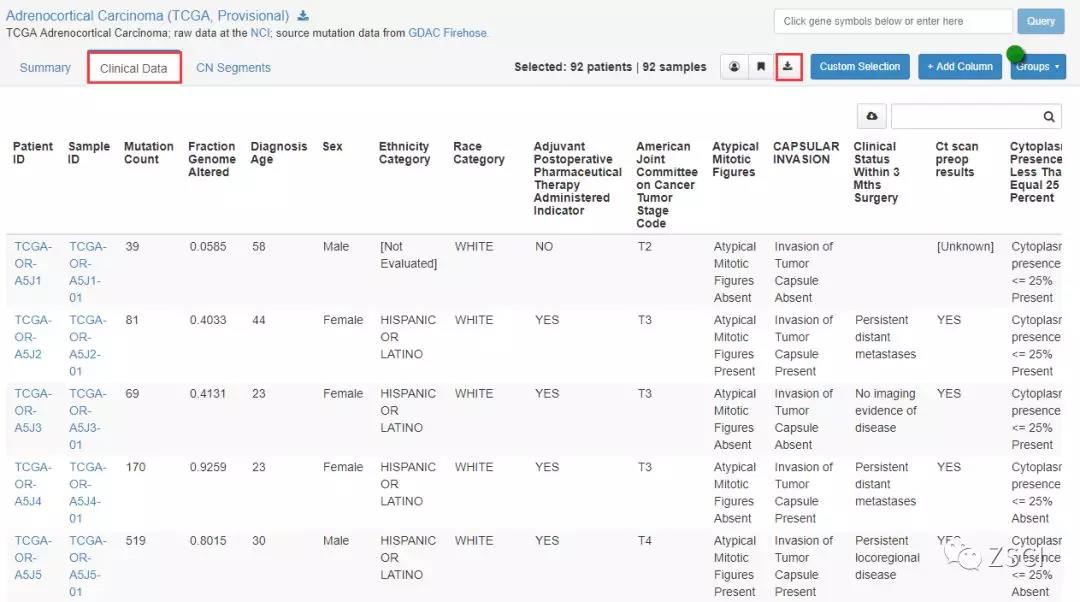
6、点击 Clinical Data 进入该疾病的临床数据存储站,点击下载按钮,便可下载该数据。
注:可以看到在右侧的 Active Data Hubs 包括了很多,不仅涵盖了 TCGA 还包括 ICGA,Pan-Cancer Atlas Hub 等数据节点。因为是研究 TCGA,只需选中 TCGA 就可以了。
7、前面说了这麽多,还是没有讲到 TCGA 的 RNAseq 数据的下载,那来看一下怎么下载,首先点击 data sets,进入下面的界面。

8、接着找到 Adrenocortical Carcinoma (TCGA, Provisional),进行数据下载。
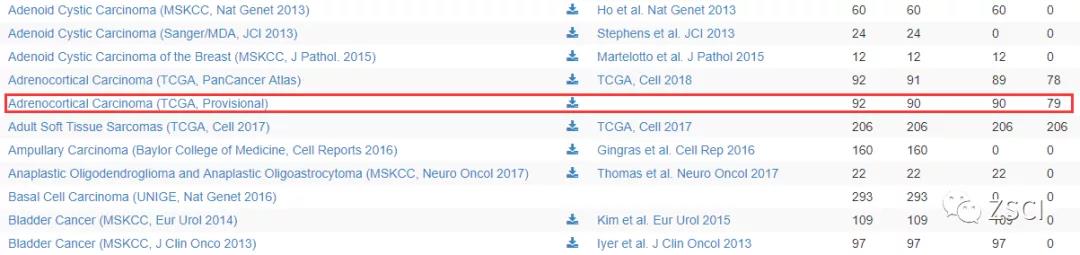
9、下载之后的数据如下,是一个压缩包,继续解压,发现包含的数据,很多,真的是一次下载多次使用。
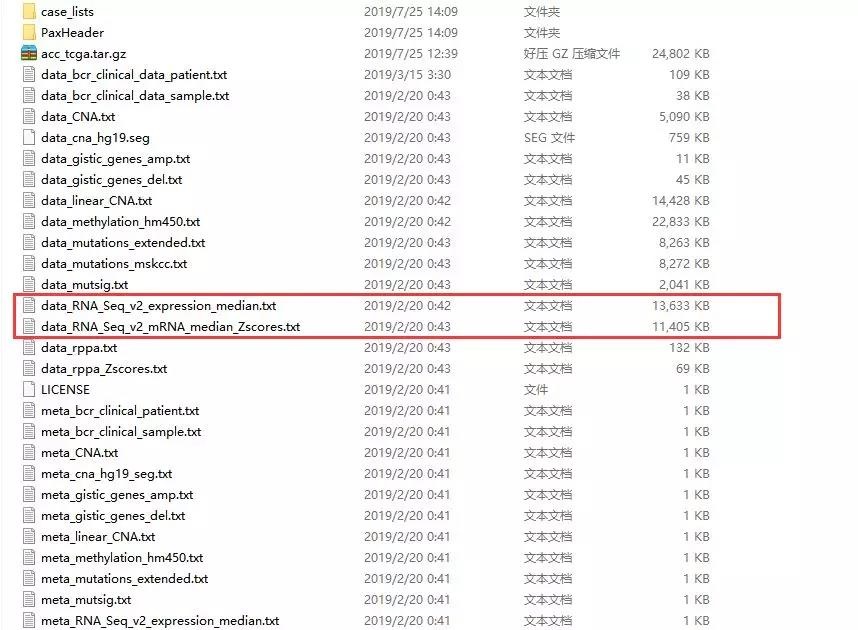
注:RNAseq 的数据类型有两种,选择一种即可。发现下载的数据不仅包括了 RNAseq 数据还包括其他各种数据,比如突变数据,450K 甲基化数据等,几乎就是把一种疾病的所有类型数据全部给下载了。
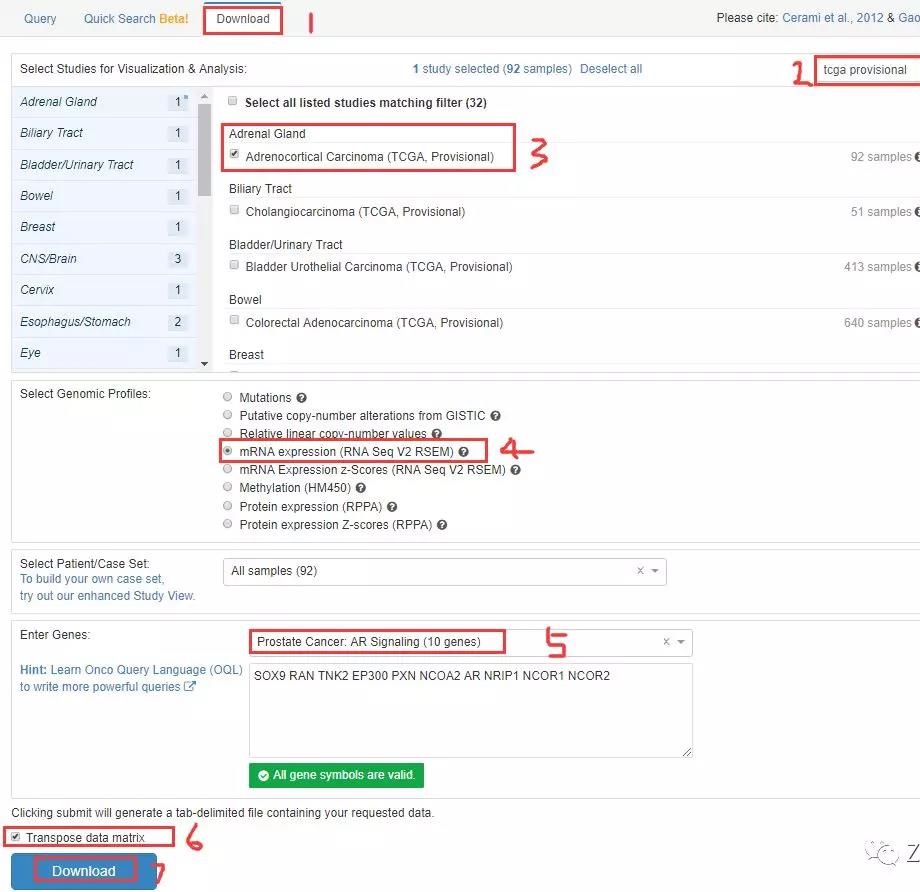
10、还可以下载关键通路上的基因表达信息,如下 1 - 7 个步骤,可以下载 AR 信号通路上 10 个基因的表达信息, 选中 Transpose data matrix, 便可使得下载的数据为表达矩阵格式
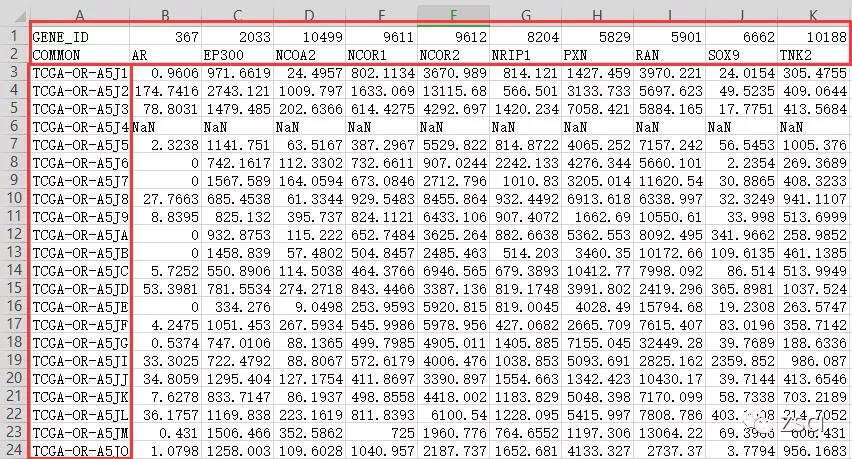
11、结果文件如下,行是样本名,列是基因名
四、通过 Fire Browse 下载 TCGA 的 RNAseq 数据
1、进入 Fire Browse
网站:http://firebrowse.org/

注:和 Fire Browse 非常相近的 FireHouse,这个和 Firebrowse 的关系,就是 Fire Browse 是 FireHouse 的浏览器,FireHouse 是数据的存储站
2、进入 FireHouse
网站:https://gdac.broadinstitute.org/
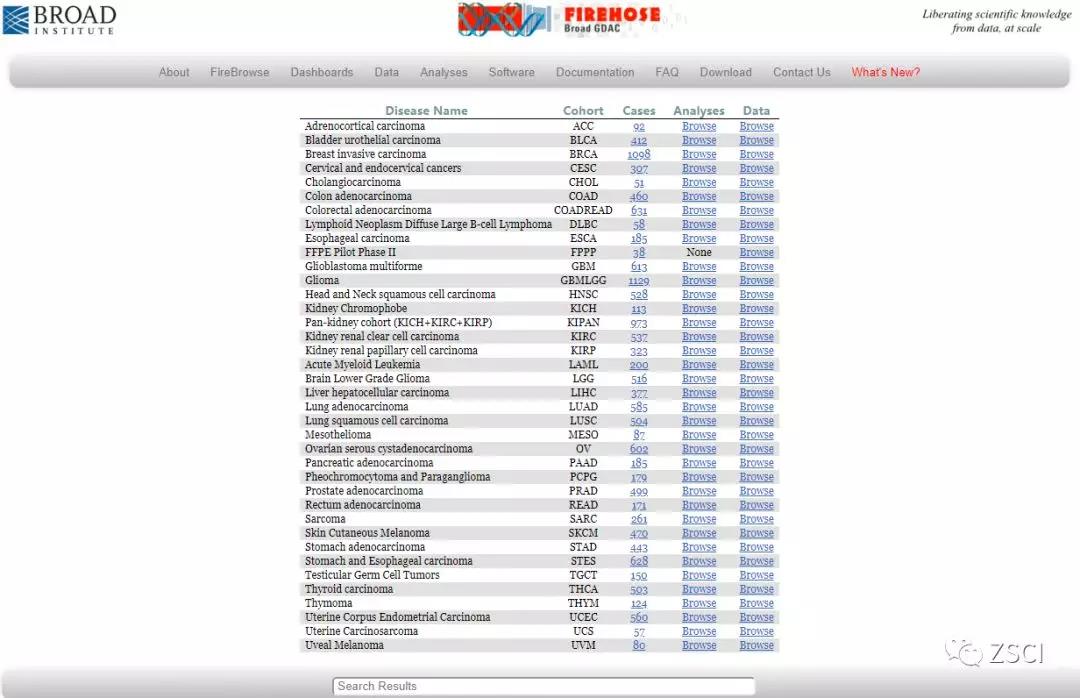
注:先来认识一下 FireHouse,主要关注 Software 和 Download
3、点击 software,界面如下:
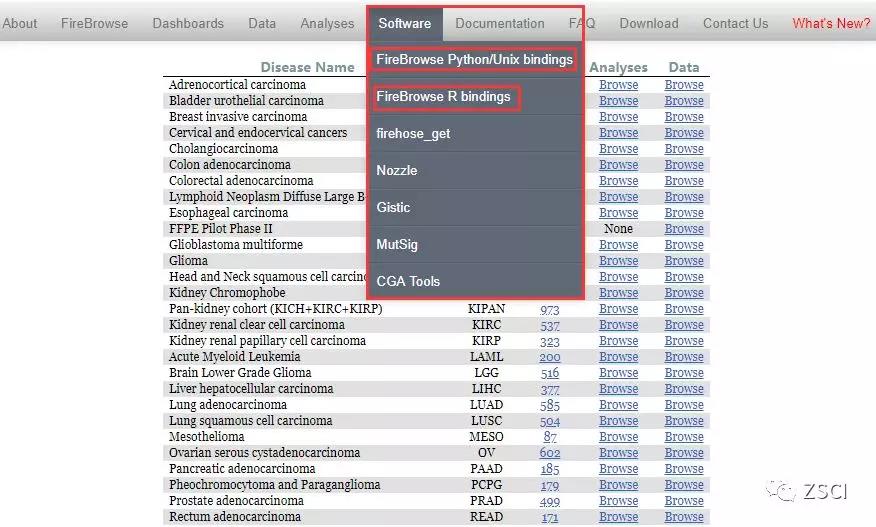
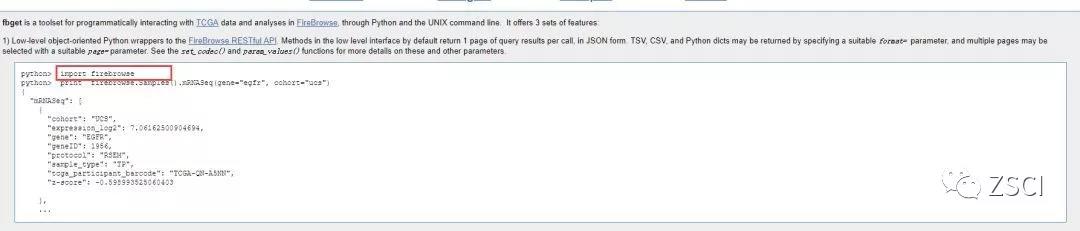
4、发现其提供了基于 python 和 R 的数据处理方式,比如点开 python,如下:
注:这个需要安装 python 环境下的 firehouse 库
5、接着点击 R 环境,如下:
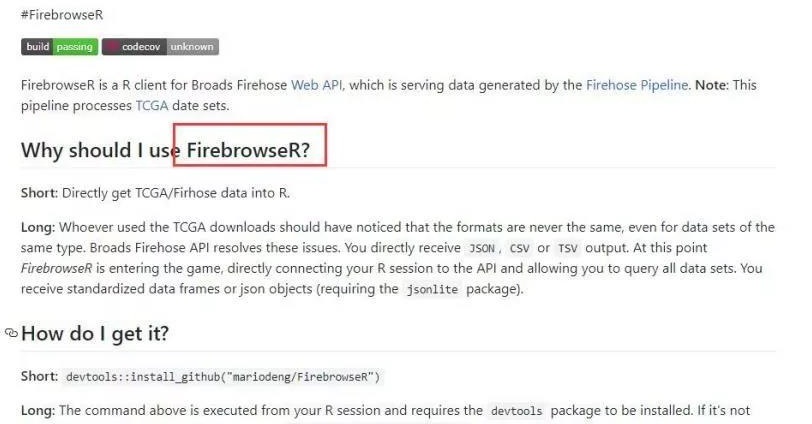
注:要求安装 FirebrowseR 这个包
6、接着点击 download,发现它提供了一个数据下载工具 firehose_get 工具,支持数据下载,并且提供了详细的软件使用说明。
7、用软件这么复杂的操作?那如何通过鼠标点击实现下载呢?首先进入 FireHouse 主界面,如下:

8、点击第一个 Data 下面的 Browse,便进入肾上腺皮质癌的数据展示界面,会出现下面的界面。
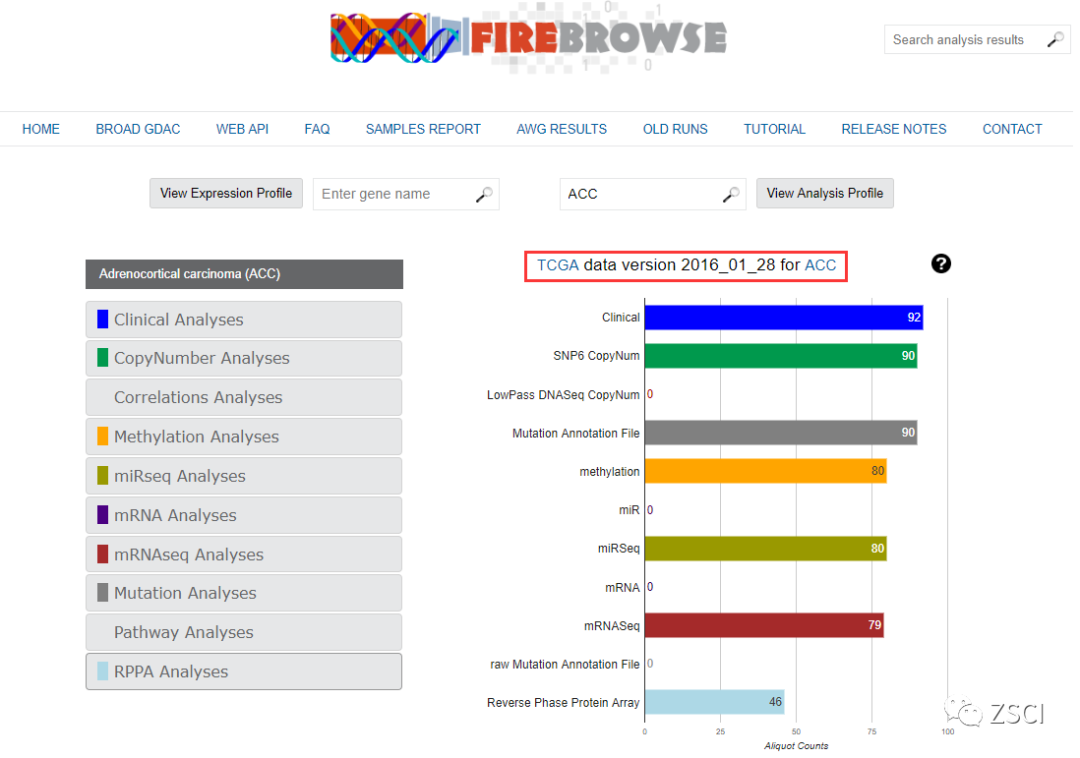
注:看到 FireBrowse 是不是有一种莫名的熟悉感?红色框框内部便是看到的 for ACC,ACC 是对肾上腺皮质癌的简写。
9、点击 mRNAseq
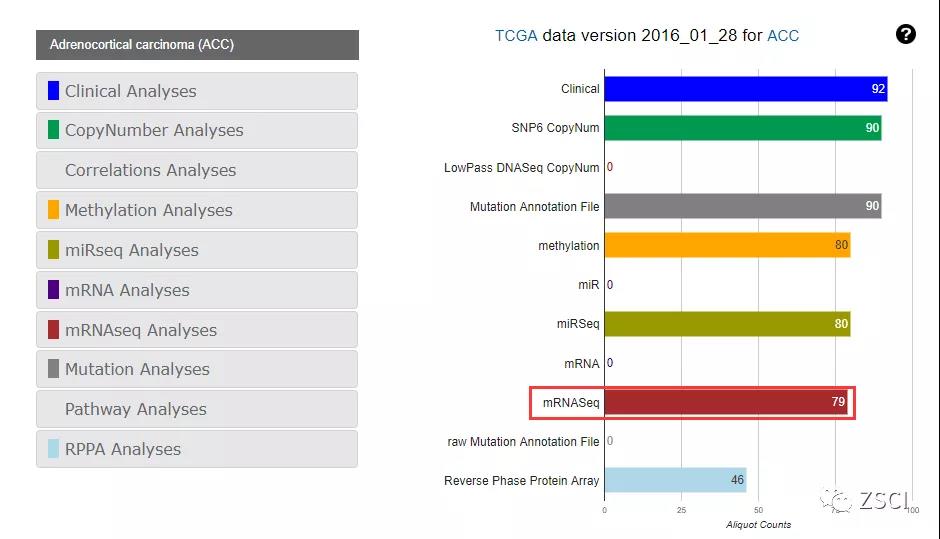
10、看到了有上述 5 类数据:
illuminahiseq_rnaseqv2 -RSEM_isoforms_normalized (MD5) 基于 RSEM 的软件基因的 isoforms 的归一化之后的表达数据
illuminahiseq_rnaseqv2 -exon_quantification (MD5) 基于 RSEM 的软件外显子定量的表达数据
illuminahiseq_rnaseqv2 -RSEM_genes (MD5) 基于 RSEM 的软件基因的表达数据
illuminahiseq_rnaseqv2 -RSEM_genes_normalized (MD5) 基于 RSEM 的软件基因的归一化之后的表达数据
mRNAseq_Preprocess (MD5) 基于 RSEM 的软件处理过程
illuminahiseq_rnaseqv2 -RSEM_isoforms (MD5) 基于 RSEM 的软件的 isoforms 的定量数据
illuminahiseq_rnaseqv2 -junction_quantification (MD5) 基于 RSEM 的软件融合基因定量数据
注:RNAseq 数据下载我们主要关注两个文件,分别是 illuminahiseq_rnaseqv2 -RSEM_genes (MD5),illuminahiseq_rnaseqv2 -RSEM_genes_normalized (MD5)
11、分别用 Excel 打开如下:
illuminahiseq_rnaseqv2 -RSEM_genes (MD5)

注:可以看到,该文件里面包含了 raw count 文件
illuminahiseq_rnaseqv2 -RSEM_genes_normalized (MD5)

注:可以看到,该文件里面包含了归一化之后的 count 文件
12、数据下载好之后,点击 Analysis

如下:
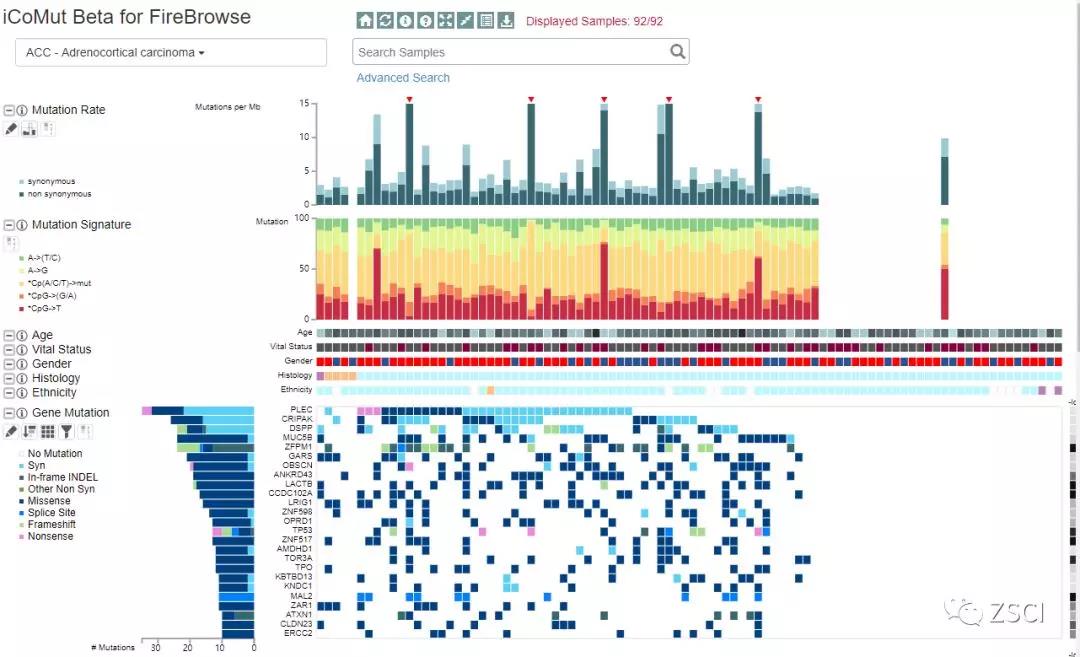
注:可以看到最显著的明显的突变基因列表。
除此之外,还可以 ACC 的 CNL 的信息,如下:
