献给初学者:如何用 Pubmed 设计 qPCR 引物
&丁香园
今天准备跟大家分享一下 qPCR 引物设计的具体流程。在进行引物设计之前,首先大家都应该了解引物设计需要注意的一些细节与原则,归纳如下:
引物设计原则
1. 引物应用核酸系列保守区内设计并具有特异性。
要设计引物首先要找到 DNA 序列的保守区。同时应预测将要扩增的片段单链是否形成二级结构。如这个区域单链能形成二级结构,就要避开它。如这一段不能形成二级结构,那就可以在这一区域设计引物。
2. 产物不能形成二级结构。
3. 引物长度一般在 15~30 碱基之间。G+C 含量在 40%~60% 之间。
4. 碱基要随机分布。
5. 引物自身不能有连续 4 个碱基的互补。引物之间不能有连续 4 个碱基的互补。
6. 引物 5' 端可以修饰。引物 3′ 端不可修饰。
7. 引物 3′ 端要避开密码子的第 3 位。
8. 引物所对应模板位置序列的 Tm 值在 72℃ 左右可使复性条件最佳。
Tm 值的计算有多种方法,如按公式 Tm=4(G+C)+2(A+T),在 Oligo 软件中使用的是最邻近法「the nearest neighbor method」。尽可能使用两条引物的 Tm 值相同「最好相差不要超过 5℃」,退火温度根据较低的 Tm 值选定,也可以通过改变引物的长度来平衡两条引物的退火温度。而对于较长的引物,Tm 值需要考虑动力学参数、从「最近邻位」的计算方式得到,现有的 PCR 引物设计软件大多数都采用这种方式。
对于 Tm 值的计算有争议的地方是附加序列应不应该计算在内,我觉得有值得商讨的地方。因为从理论上只有最开始的循环引物的附加序列是不与模板链结合的,而在后来的 PCR 反应中,引物的附加序列是和模板链结合了的。
9. ΔG 值是指 DNA 双链形成所需的自由能,该值反映了双链结构内部碱基对的相对稳定性。应当选用 3' 端 ΔG 值较低(绝对值不超过 9),而 5' 端和中间 ΔG 值相对较高的引物。引物的 3' 端的 ΔG 值过高,容易在错配位点形成双链结构并引发 DNA 聚合反应。
10. 引物二聚体及发夹结构的能值过高(超过 4.5 kcal/mol)易导致产生引物二聚体带,并且降低引物有效浓度而使 PCR 反应不能正常进行。
11. 引物的 3' 端要与模板严格配对,而 5' 端碱基没有严格的限制,只要与模板 DNA 结合的部分足够长,其 5' 端碱基可不与模板 DNA 配对而呈游离状态,这样,我们可以在引物的 5' 末端加上酶切位点(引入酶切位点时,要考虑到进行双酶切的共用缓冲液,否则给下游工作带来困难)、启动子,方便下游操作。
引物设计流程
了解了设计原则,接下来我们就看看具体的设计流程,本次讲解应用 pubmed 来设计。比如我们要设计小鼠源性白介素 4(IL-4)的引物。
1. 首先打开 pubmed, 输入如图,点击搜索:
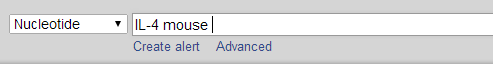
2. 在搜索结果中选择一个点击进入,得到如下信息:
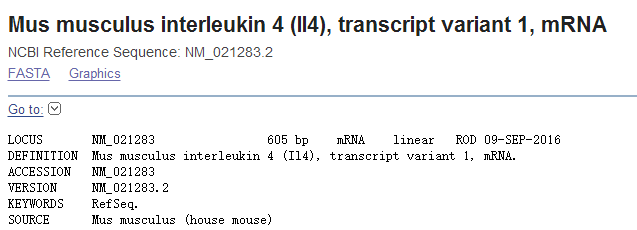
3. 点击上述界面右侧的 Pick Primers。
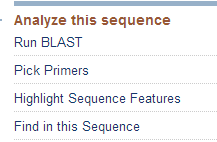
4. 点击之后进入如下界面,根据上述引物设计原则里面所述原则,填写好相关的产物长度,引物长度,Tm 值、FASTA sequence 等信息之后,点击 get primers。

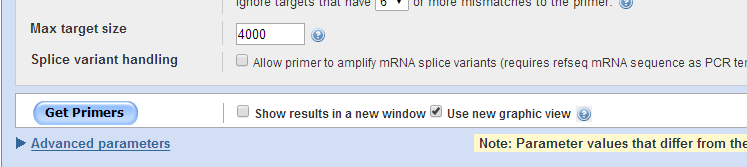
5. 点击之后就能得到很多条引物序列啦,根据需要及引物设计原则选择合适的引物序列即可。
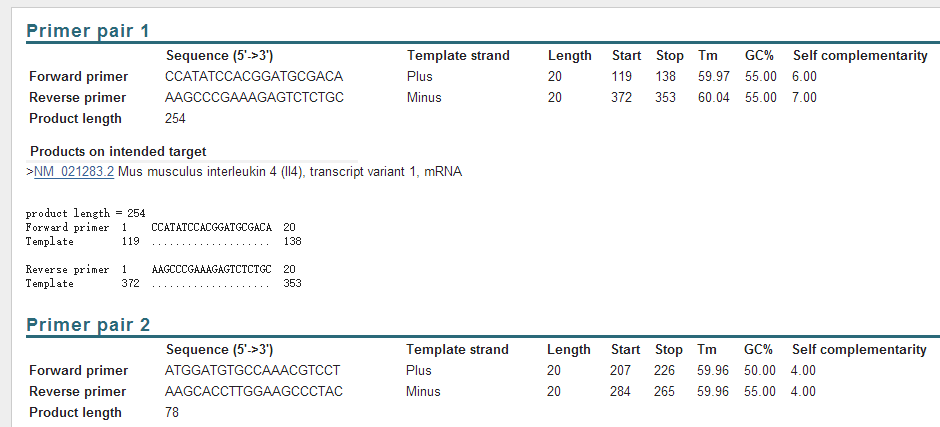
6. 引物设计好后,再应用 BLAST 检验一下。步骤如下,打开 pubmed,点击首页下方 BLAST。

将刚刚选择的引物序列输入:
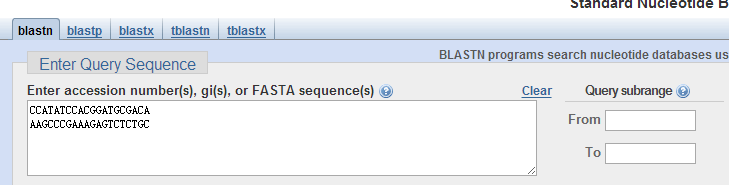
点击 blast,得到:

搜索结果中有我们刚刚查询的 NM-021283.2,说明此序列无误。
7. 另外,我们还可以应用 OligoCalc 来检测引物互补情况。进入界面输入刚刚选择的引物序列。

点击 Check 即可,结果显示 none,则为无互补情况,引物可行。
作者:魔生