简介
RNA 测序 (RNA Sequencing,简称 RNA-Seq) ,是基于高通量测序技术的转录组学研究方法,可以快速对基因组中的 RNA 种类和数量进行分析,揭示特定生物学或疾病发生过程中的分子机制。RNA 文库制备通常需要 100 ng 以上的起始 RNA,然而实际的科研实验中常会遇到样本本身较少或类型特殊,导致 RNA 起始量不足的情况,给建库实验带来极大的挑战。本章内容我们将为大家介绍微量 RNA(单细胞或低至 10 pg RNA)的特殊建库方案。
原理
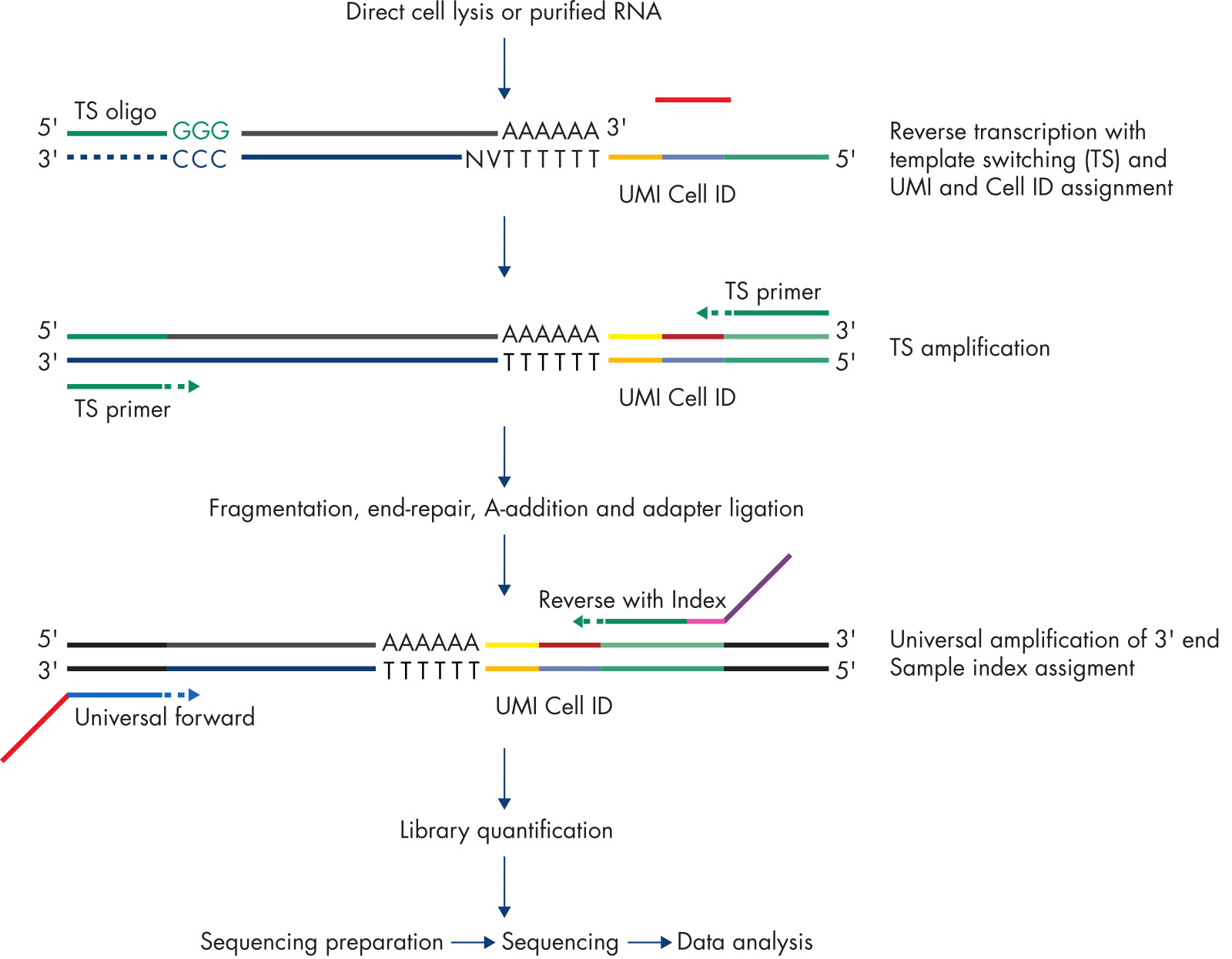
针对单细胞/微量细胞(1-1000 cells)转录组测序,目前使用的是 SMART 扩增技术。
具体步骤及反应原理如下:
1、使用 oligo dT 针对 RNA 聚合酶 II 转录的 RNA(具有 polyA 结构)进行逆转录(从而避开 rRNA);
2、逆转录到 5 cap 的时候,逆转录酶的末端转移酶活性会不依赖于 RNA 模板, 在 cDNA 的末端加上多个 C(一般是 3 个);
3、然后使用一个带有 GGG 的接头(图中的 TS oligo)与 CCC 进行配对,利用逆转录酶的 template switch 特征,继续以这个 TS oligo 为模板合成 DNA。这样 cDNA 的 3』就会加上 TS oligo 的序列;
4、进而以 TS oligo 配对的引物,对这个 cDNA 分子进行扩增。通过此方式,就可以将全长 RNA 进行指数扩增;
5、然后对扩增获得的 DNA 分子进行文库构建、测序及数据分析。
材料与仪器
步骤
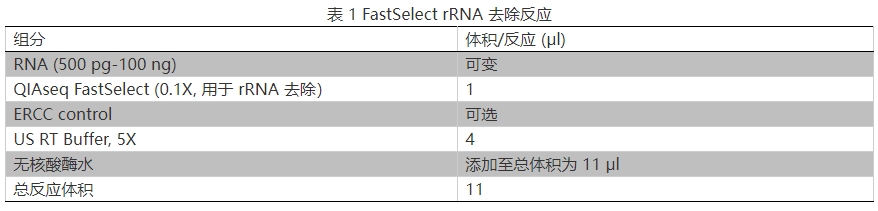
1.按表 1 准备 rRNA 去除反应体系,轻微震荡混匀。
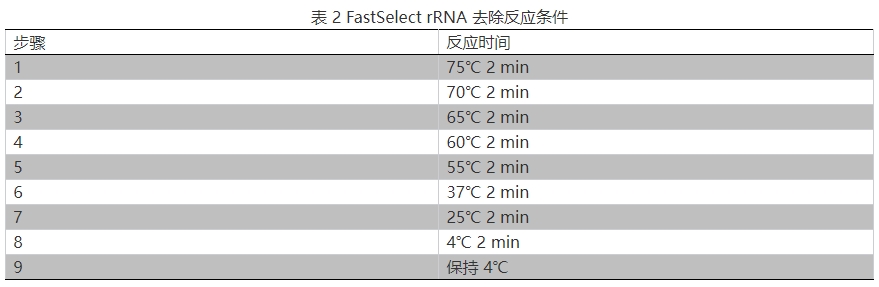
2.按表 2 进行反应:
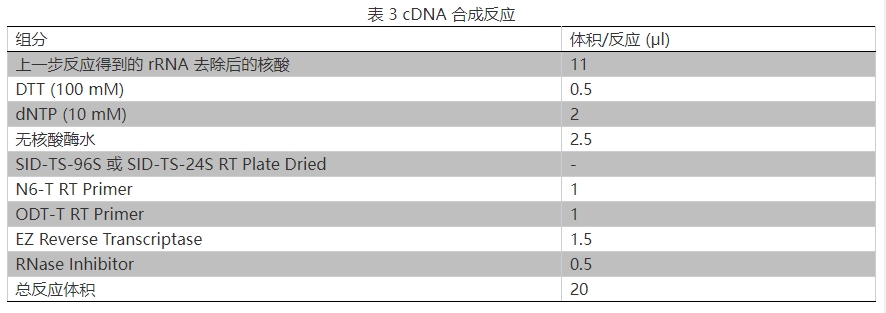
3.按表 3 准备 cDNA 合成反应体系,轻微震荡混匀。
4.将 cDNA 合成反应组分加入到 SID-TS-96S 孔板中,轻微震荡混匀并离心。
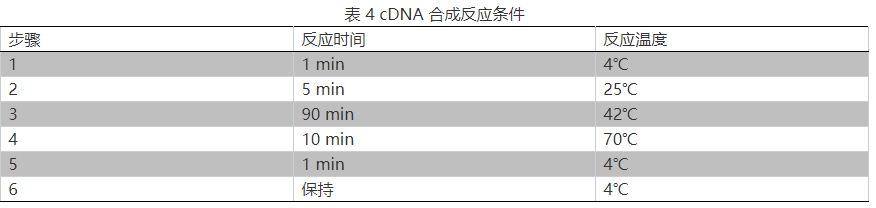
5.按表 4 进行反应:
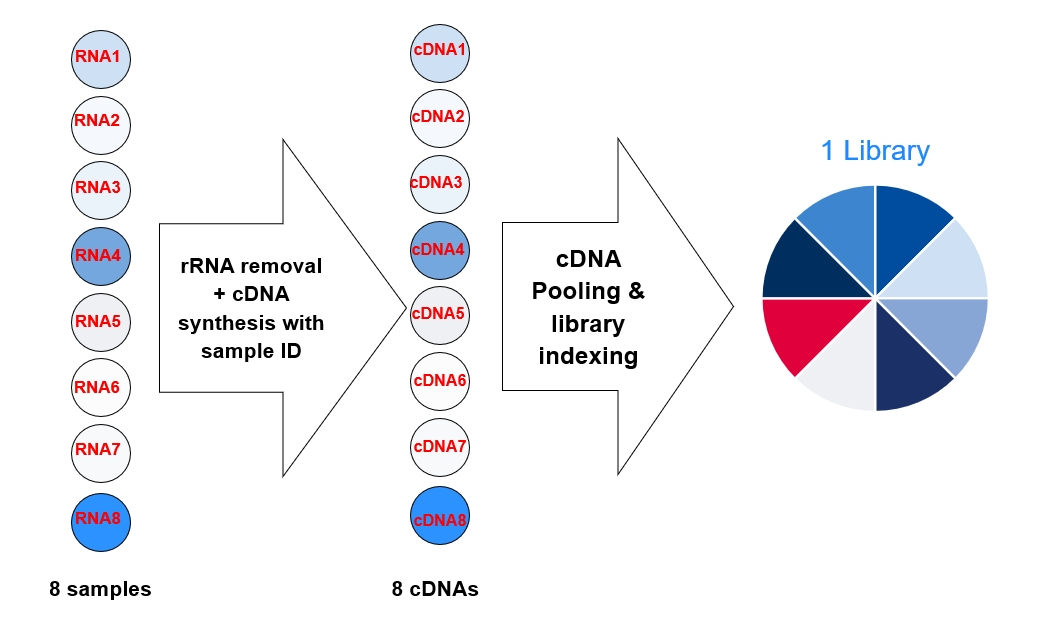
6.合成后的 cDNA,可以选 8—24 个放于 1 个 2 ml 离心管中,多个 cDNA 可以混合构建一个文库。
7.加入 1.1X 体积的 QIAseq beads 对 cDNA 产物进行纯化。
8.室温孵育 5min
9.将反应管放置于磁力架上,使磁珠聚集直到液体变澄清(约 2min),小心移去上清。
10.将反应管放置于磁力架上,加入 200 μl新鲜配制的 80% 乙醇清洗磁珠,小心吸除乙醇,勿干扰磁珠。
11.重复第 14 步的乙醇洗涤一次。
12.在磁力架上孵育 5—10min 或至磁珠晾干,磁珠过度干燥可能会导致 DNA 产量降低。
13.使用 22μl 的无核酸酶水来重悬磁珠,将反应管放置于磁力架上,使磁珠聚集直到液体变澄清,小心吸取 20 μl 的上清至新的 PCR 板中。
14.向每个样本中加入 22μl(1.1X)重悬后的磁珠。重复第 8-12 步。使用 25 μl 的无核酸酶水来重悬磁珠,将反应管放置于磁力架上,使磁珠聚集直到液体变澄清,小心吸取 23 μl 的上清至新的 PCR 板中。
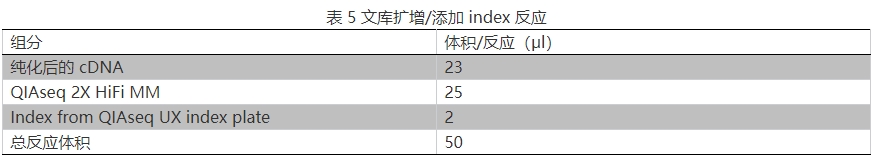
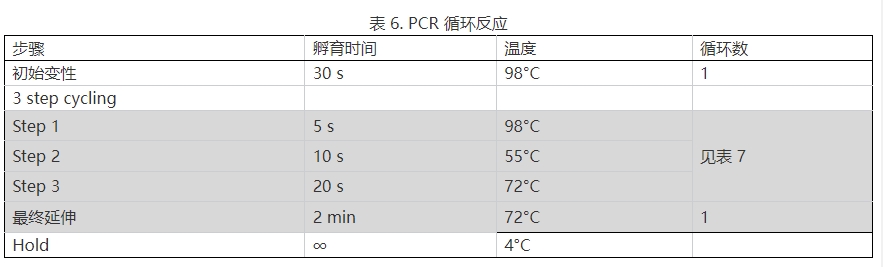
15.按表 5 步骤进行 PCR 仪(需带有热盖)程序设定
16.按表 6 设置 PCR 循环反应,PCR 循环数参考表 7。

17.加入 40 μl磁珠进行纯化,室温孵育 5min
18.将反应管放置于磁力架上,使磁珠聚集直到液体变澄清(约 2min),小心移去上清。
19.将反应管放置于磁力架上,加入 200 μl新鲜配制的 80% 乙醇清洗磁珠,小心吸除乙醇,勿干扰磁珠。
20.重复第 19 步的乙醇洗涤一次。
21.在磁力架上孵育 5—10min 或至磁珠晾干,磁珠过度干燥可能会导致 DNA 产量降低。
22.使用 22μl 的无核酸酶水来重悬磁珠,将反应管放置于磁力架上,使磁珠聚集直到液体变澄清,小心吸取 20 μl 的上清至新的 PCR 板中。
23.向每个样本中加入 16μl(0.8X)重悬后的磁珠。重复第 18-22 步。使用 24 μl 的无核酸酶水来重悬磁珠,将反应管放置于磁力架上,使磁珠聚集直到液体变澄清,小心吸取 22 μl 的上清至新的 PCR 板中。纯化后的文库可保存在-20°C 直至测序。
注意事项
1. SMART 技术的转录酶具有末端转移酶活性,单管反应一步完成逆转录和二链合成,不需要额外的 cDNA 纯化和二链合成反应,简化了建库流程并节省了操作时间。
2. 微量建库测序能否成功,在实验方面一个重要的因素就是降低核糖体在 Total RNA 中的比例。使用 polyT 引物反转录虽然能避免 rRNA,但是很难获得全长 RNA 数据。本方案结合了 rRNA 去除「黑科技」QIAseq FastSelect,整个 rRNA 去除反应完美融入逆转录步骤中,无需任何操作以及额外纯化步骤。这一「黑科技」可将总 RNA 中的 rRNA「封闭」住,使其不能进行反转录、二链合成等反应,最终无法进入构建好的 NGS 文库,显著降低 rRNA 干扰,减少测序成本的同时降低数据分析难度并提高结果的准确性。且这一方法不受起始样本量限制,低至单细胞裂解释放的 RNA 都可以进行 rRNA 去除。
3. 当 RNA 起始量较低时,测序所需的数据量也不必过高,每个样本单独构建文库既浪费建库试剂,又增加了操作时间。本方案支持 8—24 个 cDNA 混合建库,只要将不同样本反转录得到的 cDNA 混合纯化,构建一个混合 RNA-seq 文库即可。高通量测序后,根据反转录时引入的样本标签(Sample ID)拆分出每个样本的基因表达信息即可,节省成本的同时也提高了实验效率。
内容来源:QIAGEN 凯杰
来源:丁香实验