简介
快速发展的高通量测序技术为从不同类型起始样本中精确而灵敏地解析基因密码提供了可能。但在实际的实验过程中,疑难样本的 DNA 常因含量低、质量差而极大地限制了高通量测序技术的应用,主要表现在无法满足建库过程所需的最低条件。很多研究人员面对不足 1 ng 的样本往往束手无策,实际上随着生物技术和酶活性的进步,目前的技术已经可以针对 10 pg 样本进行全基因组建库(如需对 DNA 进行打断则最低起始量为 20 pg)。
原理
基于经典的 TA 克隆连接接头建库技术,基本过程是针对 DNA 末端进行补平修复和加 A,再在 DNA 连接酶的作用下,这种末端带 A 尾的目的片段通过 TA 克隆方式和合成时就带上 T 尾的接头连接。之后可通过 PCR 扩增进行文库富集,也可不扩增进行 PCR-Free 建库,最后经过纯化和分选即完成建库过程。
QIAseq Ultralow Input Library Kit 拥有极低的样本 input 要求,10 pg 即可完成 WGS 文库构建,高效末端修复和连接效率可最大程度将 DNA 转化为有效测序文库,适用于所有核酸类型,包括严重降解的古 DNA、环境 DNA 和 FFPE 核酸。基于酶法打断的建库试剂盒 QIAseq FX DNA Library Kit 支持低至 20 pg 的微量起始样本,FX 核酸片段化酶不受基因序列影响,通过单一酶的作用可对基因组 DNA 实现快速无偏差的均匀打断,将 GC 偏好性降到最低。
材料与仪器
步骤
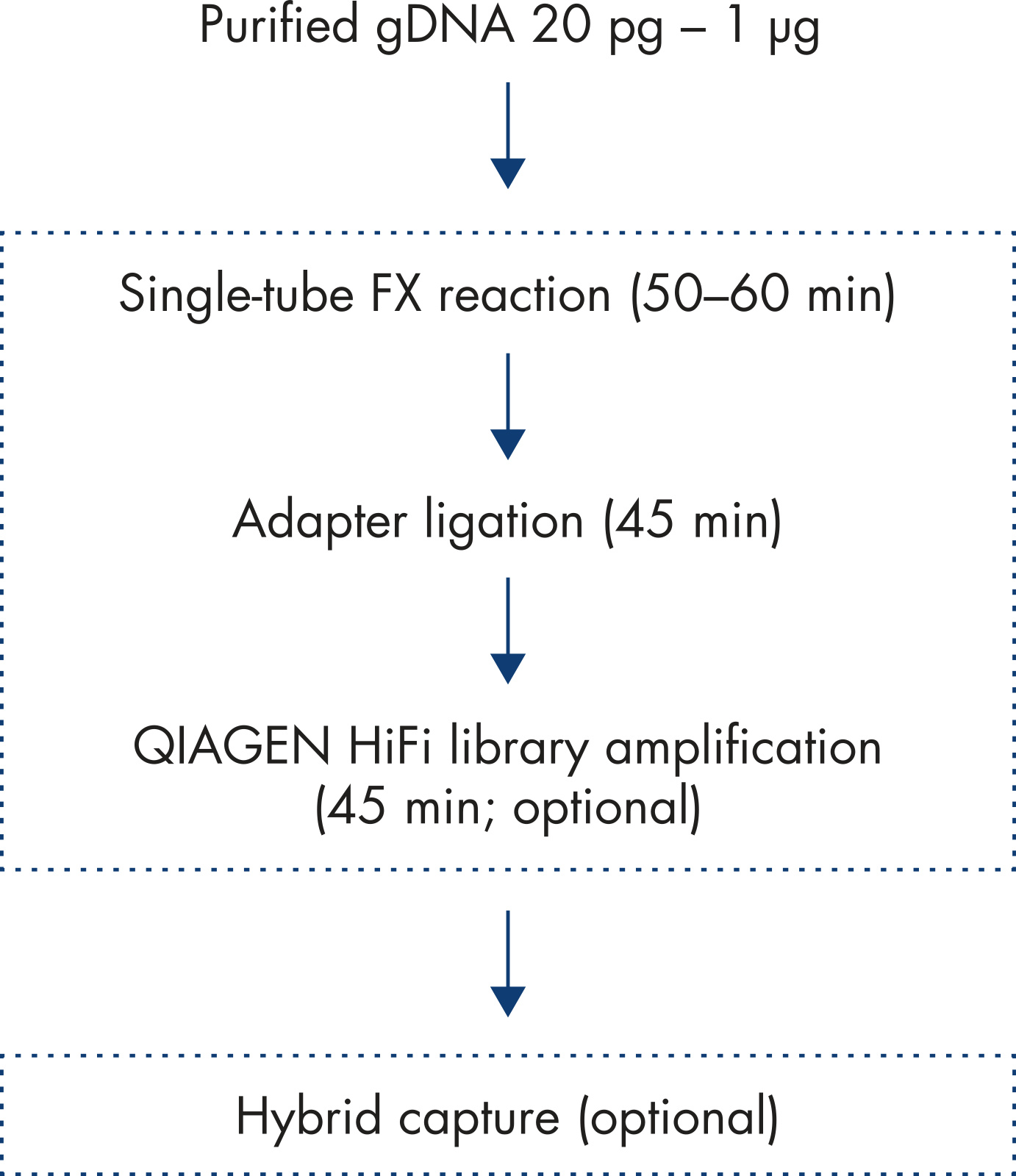
1. 在冰上将所有试剂及接头进行解冻,按下述步骤进行 PCR 仪(需带有热盖)程序设定(表 2),盖温 70℃
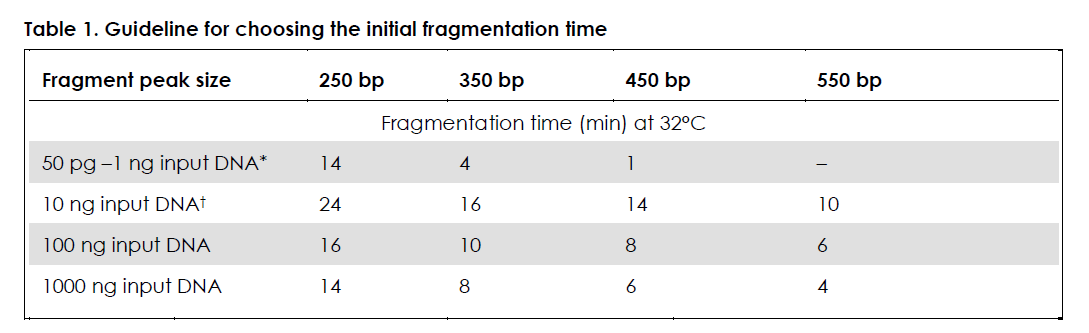
input<50 pg 时,需加入 FX Enhancer,打断时间 25 min,可产生约 250 bp 的 DNA 片段。
表 2. FX 反应循环条件
|
步骤 |
温度 |
孵育时间 |
|
1 |
4°C |
1 min |
|
2 |
32°C |
1—30 min |
|
3 |
65°C |
30 min |
|
4 |
4°C |
Hold |
2. 开始反应程序,至热模块降温到 4°C 时,暂停程序。
3. 根据表 3 和表 4,在冰上配置 FX 反应体系
表 3. FX 反应体系的构建(单个样本,起始 DNA 大于 10 ng)
|
组分 |
体积/反应 (μl) |
|
FX Buffer, 10x |
5 |
|
纯化后 DNA |
可变 |
|
无核酸酶水 |
可变 |
|
总反应体积 |
40 |
表 4. FX 反应体系的构建(单个样本,起始 DNA 小于10 ng)
|
组分 |
体积/反应 (μl) |
|
FX Buffer, 10x |
5 |
|
纯化后 DNA |
可变 |
|
FX Enhancer |
2.5 |
|
无核酸酶水 |
可变 |
|
总反应体积 |
40 |
4. 使用移液器缓慢吹打将体系混匀,请勿涡旋震荡。在冰上向每个反应体系中加入 10μl FX Enzyme Mix,并轻轻吹打 6—8 次将体系进一步混匀。
5. 将含有反应体系的 PCR 板或管进行简短离心后立即转移到已经预冷到 4°C 的 PCR 仪上,恢复上述反应程序,待反应程序结束后,将样本转移至冰上,并继续开始后续试验。
6. 将含有接头的 PCR 板进行涡旋并离心,移去板盖,刺穿锡箔封膜,依次从各孔中吸取 5μl 接头至各样本体系中,记录各样本所使用的接头编码,并将剩余的接头进行冻存。如果 input<10 ng,请按表 5 进行接头稀释。
7. 根据表 6 在冰上配制接头连接反应的体系,吹打混匀。
表 6. 接头连接反应体系(每个样本)
|
组分 |
体积/反应 (μl) |
|
DNA Ligase Buffer, 5x |
20 |
|
DNA 连接酶 |
10 |
|
无核酸酶水 |
15 |
|
总反应体积 |
45 |
8. 向每个样本中加入 45μl 的上述连接反应体系,此时总反应体系为 100 μl,混匀并在 20°C 孵育 15 min。
9. 立即进入接头连接后纯化步骤(第 10-18 步)。
10. 向每个样本中加入 80μl(0.8X)重悬后的 Agencourt AMPure XP beads。
11. 室温孵育 5 min
12. 将反应管放置于磁力架上,使磁珠聚集直到液体变澄清(约 2 min),小心移去上清。
13. 将反应管放置于磁力架上,加入 200 μl新鲜配制的 80% 乙醇清洗磁珠,小心吸除乙醇,勿干扰磁珠。
14. 重复第 14 步的乙醇洗涤一次。
15. 在磁力架上孵育 5—10min 或至磁珠晾干,磁珠过度干燥可能会导致 DNA 产量降低。
16. 使用 52.5μl 的 Buffer EB 或 10 mM Tris-HCl pH 8.0 来重悬磁珠,将反应管放置于磁力架上,使磁珠聚集直到液体变澄清,小心吸取 50 μl 的上清至新的 PCR 板中。
17. 向每个样本中加入 50μl(1X)重悬后的 Agencourt AMPure XP beads。
18. 重复第 11-15 步。使用 26μl 的 Buffer EB 或 10 mM Tris-HCl pH 8.0 来对重悬磁珠,将反应管放置于磁力架上,使磁珠聚集直到液体变澄清,小心吸取 23.5 μl 的上清至新的 PCR 板中。
19. 按下述步骤进行 PCR 仪(需带有热盖)程序设定(表 7)
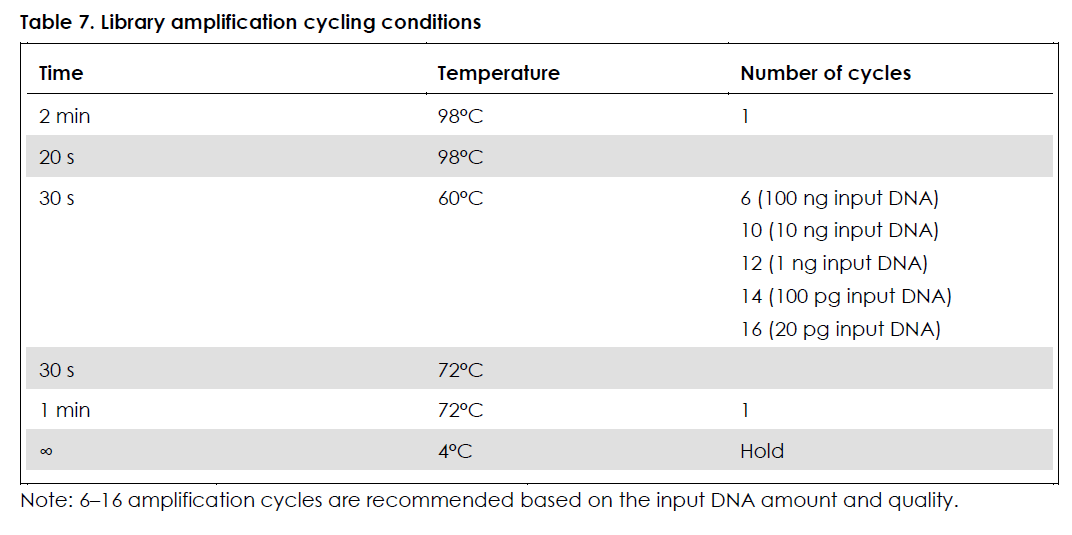
20. 按表 8 在冰上向每个反应体系中加入各试剂,并轻轻吹打 6—8 次来将体系进一步混匀。
表 8. 扩增反应体系
|
组分 |
体积/反应(μl) |
|
文库 DNA |
23.5 |
|
HiFi PCR Master Mix, 2X |
25 |
|
Primer Mix |
1.5 |
|
总反应体积 |
50 |
21. 将 PCR 板放置到 PCR 仪中,开始程序。
22. PCR 反应结束后,向 50μl 的样本中加入 50 μl(1X)的重悬后的 Agencourt AMPure XP beads。
23. 重复上述第 11-15 步。
24. 使用25μl 的 Buffer EB 或 10 mM Tris-HCl pH 8.0 来对重悬磁珠,将反应管放置于磁力架上,使磁珠聚集直到液体变澄清,小心吸取 23 μl 的上清至新的离心管中。纯化后的文库可保存在-20°C 直至测序。
注意事项
1. DNA 纯化过程中的洗脱液应使用 QIAGEN 的 Buffer EB、H2O、10 mM Tris 或低浓度 TE(0.1x TE, 0.1 mM EDTA)。含 EDTA 的 1 x TE buffer 会影响 DNA 打断,如果使用了 TE buffer 需要先用磁珠纯化掉该 buffer。
2. 每次实验需重新配置 80% 乙醇,磁珠使用前室温平衡 20—30 min。
3. 文库扩增非必选步骤。一般起始样本大于 50 ng 可进行 PCR-free 文库构建。
4.可以通过打断时间控制文库片段的分布。
内容来源: QIAGEN 凯杰
来源:丁香实验