盘点 PCR 的七大应用领域
赛默飞世尔科技
PCR 是众所周知的分子生物学技术之一。20 世纪 70 年代,研究人员首次报道了使用合成引物和 DNA 聚合酶从模板复制单链 DNA。然而直到 1983 年,Kary Mullis 才发明出用于扩增目标 DNA 的研究工具,就是我们今天所熟知的 PCR 方法。从此以后,PCR 成为分子生物学研究必不可少的一部分,被广泛应用于基础研究、疾病诊断、农业检测和法医调查等领域。
1.基因表达
通常可通过 PCR 来检测不同细胞类型、组织和生物体在特定时间点的基因表达差异。首先,从目标样品中分离出 RNA 并将mRNA逆转录成 cDNA。随后,通过由PCR扩增的cDNA数量,确定 mRNA 的初始水平。这一过程也被称为逆转录 PCR, RT-PCR (图 1)。
 图 1. RT-PCR.RNA 被逆转录成 cDNA,随后通过 PCR 扩增 cDNA
图 1. RT-PCR.RNA 被逆转录成 cDNA,随后通过 PCR 扩增 cDNA
终点 PCR 可通过凝胶里的扩增产物条带强度对 RNA 的表达进行定量(一种半定量方法)。例如,对起始 cDNA 进行连续稀释并扩增。通过凝胶电泳使不同起始量的终点 PCR 得率可视化(图 2),然后对条带强度进行定量,并以管家基因为参照进行标准化,预估扩增靶点的相对表达水平[1,2]。如今,终点 PCR 已基本被实时 PCR 或 qPCR 取代了,因为它们可获得更可靠和更准确的基因表达定量结果。
 图 2:起始 cDNA 连续稀释液的 PCR 得率,通过在琼脂糖凝胶上对 PCR 产物染色进行可视化观察。
图 2:起始 cDNA 连续稀释液的 PCR 得率,通过在琼脂糖凝胶上对 PCR 产物染色进行可视化观察。
2.基因分型
PCR 可用于检测特定细胞或生物体中等位基因的序列差异。例如,基因敲除和敲入小鼠等转基因生物的基因分型[3]。引物对经设计位于目标区域侧翼,可根据是否存在扩增子及扩增子长度来检测遗传变异(图 3)。
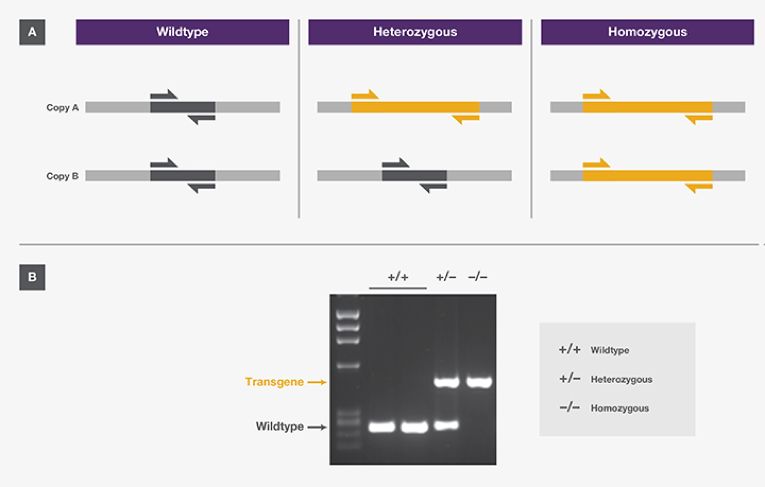 图 3.PCR 用于转基因生物的等位基因分型。(A)可用针对目标区域的特异性PCR引物来检测基因位点是野生型序列(深灰色)还是转基因序列(黄色)。(B)如该凝胶照片所示,PCR 产物可用于确定基因型。在这些实验中,使用了野生型 (+/+)和转基因 (+/–和–/–) 小鼠的基因组 DNA。
图 3.PCR 用于转基因生物的等位基因分型。(A)可用针对目标区域的特异性PCR引物来检测基因位点是野生型序列(深灰色)还是转基因序列(黄色)。(B)如该凝胶照片所示,PCR 产物可用于确定基因型。在这些实验中,使用了野生型 (+/+)和转基因 (+/–和–/–) 小鼠的基因组 DNA。
但是如果为了检测特定核苷酸突变,则必须对扩增的序列进行进一步分析。例如, PCR 扩增子测序就是研究单核苷酸变异(SNVs)和单核苷酸多态性(SNP)的方法之一。强烈推荐使用高保真 DNA 聚合酶,以防止在 PCR 过程中引入多余的突变。
通过 PCR 进行基因分型也是对癌症和遗传病中的 突变进行遗传分析的一个基本方式。
3.分子克隆
PCR 被广泛应用于目标 DNA 片段的克隆,该技术被称为 PCR 克隆。在直接 PCR 克隆中,DNA(如,gDNA、cDNA、质粒 DNA)的目标区域被扩增并插入到特殊设计的兼容载体中。
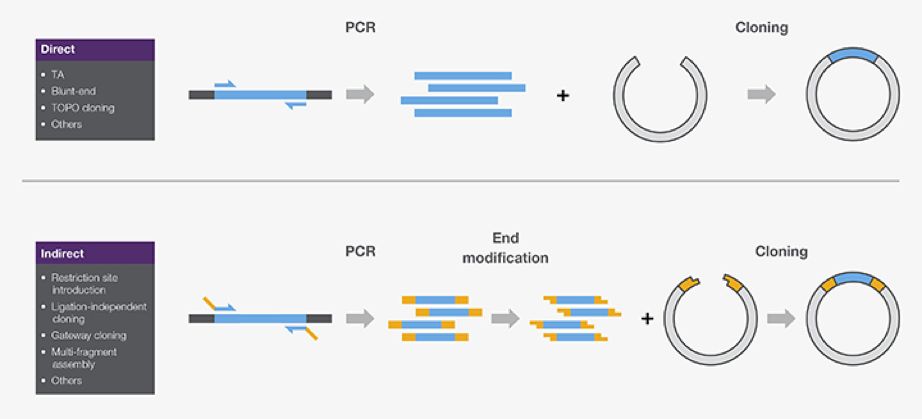 图 4.PCR 克隆:通过 PCR 制备的插入片段被克隆到兼容载体中。(A) 直接 PCR 克隆技术包括 TA 和平端克隆。(B)间接 PCR 克隆,扩增子可在插入到兼容载体之前被修饰,如进行限制性酶切。
图 4.PCR 克隆:通过 PCR 制备的插入片段被克隆到兼容载体中。(A) 直接 PCR 克隆技术包括 TA 和平端克隆。(B)间接 PCR 克隆,扩增子可在插入到兼容载体之前被修饰,如进行限制性酶切。
除了制备插入片段,PCR 也是一种在在克隆后筛选克隆是否携带目标插入片段的有效方法。引物经过设计,可以用于确定载体中插入片段是否存在和插入方向。
4.突变
PCR 克隆的一大优点是能够通过克隆将所需突变引入目的基因中,以便进行突变研究。在定点突变中,经过设计的 PCR 引物可将碱基置换、删除或插入整合到特定序列中。如图 5所示,引物定位到已克隆至质粒中的序列上。随后,含有引入突变的 PCR 产物通过自我连接,重新生成环状质粒,并用于转化感受态细胞。
 图 5.PCR 用于定点突变。该方法使用非重叠引物(红色星号=突变核苷酸,灰色线条 = 删除的序列,蓝色线条 = 插入的序列)。也可考虑使用其他引物设计,如具有 5′ 重叠序列的引物[4-6]。
图 5.PCR 用于定点突变。该方法使用非重叠引物(红色星号=突变核苷酸,灰色线条 = 删除的序列,蓝色线条 = 插入的序列)。也可考虑使用其他引物设计,如具有 5′ 重叠序列的引物[4-6]。
 图 6.使用含突变序列和同源末端序列的 PCR 引物进行的定点突变。该图所示方法阐述了 Invitrogen™ GeneArt™ 定点突变试剂盒的机制,其中,方块代表重组和突变位点。
图 6.使用含突变序列和同源末端序列的 PCR 引物进行的定点突变。该图所示方法阐述了 Invitrogen™ GeneArt™ 定点突变试剂盒的机制,其中,方块代表重组和突变位点。
5.甲基化
PCR 可用于研究位点特异性甲基化。在 甲基化特异性 PCR(MSP)方法中,设计了两个引物对,以区分目标位点的甲基化状态[7,8]。
首先使用重亚硫酸盐处理 DNA 样品,将未甲基化的胞嘧啶(C)转化为尿嘧啶(U)。重亚硫酸盐处理不会影响甲基化的胞嘧啶(m5C) 。为了检测甲基化位点,一对引物经设计带有鸟嘌呤(G),可与目标序列中的 m5C 配对;为了检测未甲基化位点,另一对引物带有腺嘌呤(A),可与重亚硫酸盐转化分子中的U配对(随后,与后续 PCR 循环中的胸腺嘧啶(T)配对)。通过引物配对得到的阳性 PCR 扩增结果可用于确定位点的甲基化状态(图 7)。
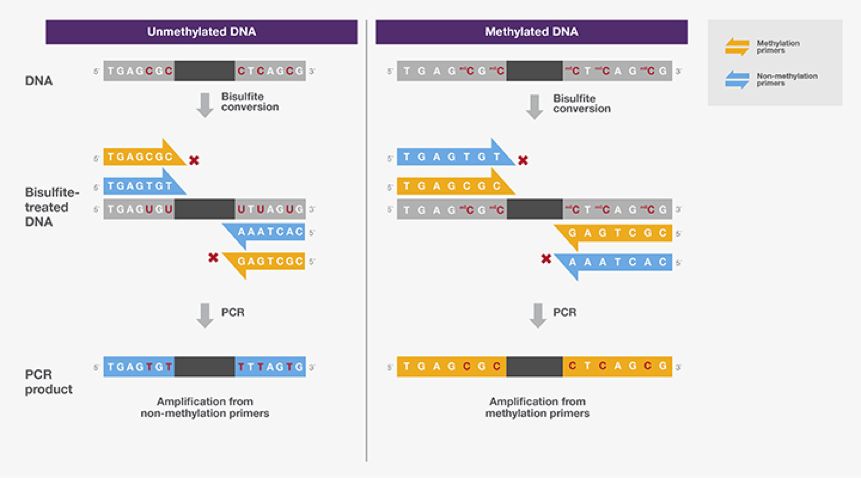 图 7.甲基化特异性 PCR 第一步,使用重亚硫酸盐处理 DNA 样品,将未甲基化的胞嘧啶转化为尿嘧啶。设计两个引物对(甲基化和未甲基化),从而根据重亚硫酸盐处理 DNA 的扩增结果来区分目标位点的甲基化状态[7,8]。
图 7.甲基化特异性 PCR 第一步,使用重亚硫酸盐处理 DNA 样品,将未甲基化的胞嘧啶转化为尿嘧啶。设计两个引物对(甲基化和未甲基化),从而根据重亚硫酸盐处理 DNA 的扩增结果来区分目标位点的甲基化状态[7,8]。
甲基化研究中所使用的 DNA 聚合酶除了能够扩增富含AT的序列,还必须能够读取识别重亚硫酸盐处理后 DNA 中的 U 残基。而高保真 DNA 聚合酶含有来自于古细菌起源的尿嘧啶结合域,所以不适用于 MSP(除非经过特殊修饰)。
实时 PCR 可代替终点 PCR,为 MSP 提供更准确的甲基化定量分析。利用实时 PCR,对 PCR 扩增子的熔解曲线分析是检测目标位点甲基化状态的一种替代性 PCR 方法。
6.测序
PCR 是为测序富集模板 DNA 的一种相对简单的方法。为保证 DNA 序列准确性,强烈建议使用高保真 PCR 来制备测序模板。
在 Sanger 测序中,PCR 扩增片段经纯化并用于测序反应。使用常用的测序引物结合位点(如 M13 或 T7“通用引物”结合位点)对 PCR 引物的 5′末端进行标记,以简化测序工作流程(图 8)。
 图 8.用于 Sanger 测序的 PCR 扩增子制备。使用常用测序引物位点标记 PCR 引物,以简化实验流程。
图 8.用于 Sanger 测序的 PCR 扩增子制备。使用常用测序引物位点标记 PCR 引物,以简化实验流程。
二代测序 (NGS)中,PCR 被广泛用于构建 DNA 测序文库。在 NGS 文库制备中,DNA 样品通过 PCR 反应富集(在起始量有限的情况下)并使用 adaptor(以及用于多重检测的index)标记(图 9)。除了具有高保真度,DNA 聚合酶还应具有最小的扩增偏好性,从而使测序文库具有高覆盖度。
 图 9.使用 PCR 为新一代测序制备 DNA 样品。
图 9.使用 PCR 为新一代测序制备 DNA 样品。
7.医学、法医学和应用科学
PCR 技术不仅可用于基础研究,还适用于日常的临床诊断、法医学调查和农业生物技术研究。这些应用要求可靠的性能、卓越的灵敏度和严格的标准。因此,所使用的 PCR 仪和 PCR 试剂必须符合这些要求和目的。
分子诊断应用包括基因检测、致癌突变检测以及感染性疾病检测。在法医学中,利用 PCR 进行人类身份鉴定是通过对独特的短串联重复序列(STR)进行扩增而区分个体的。在农业学中,PCR 在食物病原体检测、育种植物基因分型和 GMO 测试中具有重要作用。
参考文献:
1.Raeymaekers L (1999) General Principles of Quantitative PCR.In: Kochanowski B, Reischl U (editors), Quantitative PCR Protocols.Totowa, NJ: Humana Press. pp. 31–41.
2.Siebert PD (1999) Quantitative RT-PCR.In: Kochanowski B, Reischl U (editors), Quantitative PCR Protocols.Totowa, NJ: Humana Press. pp. 61-85.
3.Nobel Media AB (2014) The 2007 Nobel Prize in Physiology or Medicine - Advanced Information. nobelprize.org
4.Zheng L, Baumann U, Reymond JL (2004) An efficient one-step site-directed and site-saturation mutagenesis protocol.Nucleic Acids Res32(14):e115.
5.Liu H, Naismith JH (2008) An efficient one-step site-directed deletion, insertion, single and multiple-site plasmid mutagenesis protocol.BMC Biotechnol8:91.
6.Xia Y, Chu W, Qi Q et al.(2015) New insights into the QuikChange™ process guide the use of Phusion DNA polymerase for site-directed mutagenesis.Nucleic Acids Res43(2):e12.
7.Herman JG, Graff JR, Myöhänen S, Nelkin BD, Baylin SB (1996) Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands.Proc Natl Acad Sci U S A93(18):9821–9826.
8.Huang Z, Bassil CF, Murphy SK (2013) Methylation-specific PCR.Methods Mol Biol 1049:75–82.
文章来源:赛默飞世尔科技