【共享】RNA干扰技术概况
丁香园论坛
1387
RNA干涉(RNAi)
1995年,康乃尔大学的Su Guo博士在试图阻断秀丽新小杆线虫(C. elegans)中的par-1基因时,发现了一个意想不到的现象。她们本是利用反义RNA技术特异性地阻断上述基因的表达,而同时在对照实验中给线虫注射正义RNA(sense RNA)以期观察到基因表达的增强。但得到的结果是二者都同样地切断了par-1基因的表达途径。这是与传统上对反义RNA技术的解释正好相反的。该研究小组一直没能给这个意外以合理解释。
直到1998年2月,华盛顿卡耐基研究院的Andrew Fire和马萨诸塞大学医学院的Craig Mello才首次揭开这个悬疑之谜。通过大量艰苦的工作,他们证实,Su Guo博士遇到的正义RNA抑制基因表达的现象,以及过去的反义RNA技术对基因表达的阻断,都是由于体外转录所得RNA中污染了微量双链RNA而引起。当他们将体外转录得到的单链RNA纯化后注射线虫时发现,基因抑制效应变得十分微弱,而经过纯化的双链RNA却正好相反,能够高效特异性阻断相应基因的表达。该小组将这一现象称为RNA干扰(RNA interference ,简称RNAi)。
在随后的短短一年中,RNAi现象被广泛地发现于真菌、拟南芥、水螅、涡虫、锥虫、斑马鱼等大多数真核生物中。这种存在揭示了RNAi很可能是出现于生命进化的早期阶段。随着研究的不断深入,RNAi的机制正在被逐步阐明,而同时作为功能基因组研究领域中的有力工具,RNAi也越来越为人们所重视。
RNAi的作用机制
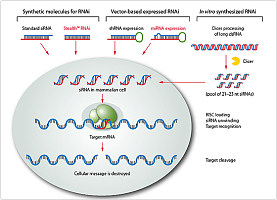
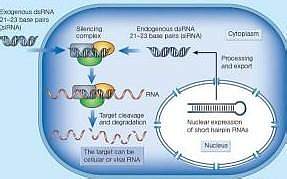
体外实验表明:RNAi反应中,加入的dsRNA被切割为21-23核苷酸长的RNA片段,后者会使目的mRNA被切割为21-23核苷酸长的片段。从已经发生RNAi的果蝇S2细胞中,Hammond等人部分纯化了一种核酸酶,该核酸酶具有序列特异性,它仅降解与引起RNAi的dsRNA具有同源序列的mRNA。那么这种核酸酶是如何确定哪些mRNA该降解而哪些不该呢?由于在纯化该核酸酶时,可以共分离出21-23核苷酸长的dsRNA片段,这暗示该核酸酶对mRNA的切割有可能是以这些片段作模板指导进行的。根据以上的实验结果,人们提出一种RNAi作用的简单模型。当dsRNA导入细胞后,被一种dsRNA特异的核酸内切酶识别,切割成21-23核苷酸长的小片段,这些片段可与该核酸酶的dsRNA结合结构域结合,并且作为模板识别目的mRNA;识别之后,mRNA与dsRNA的有义链发生链互换,原先dsRNA中的有义链被mRNA代替,从酶-dsRNA复合物中释放出来,而mRNA则处于原先的有义链的位置。核酸酶在同样位置对mRNA进行切割,这样又产生了21-23核苷酸长的dsRNA小片段,与核酸酶形成复合物,继续对目的mRNA进行切割,从而使目的基因沉默,产生RNAi现象。通过遗传分析的方法,目前已从线虫中已分离到RDE-2,RDE-3和Mut-7等RNAi相关的基因。
RNAi技术在功能基因组中的应用
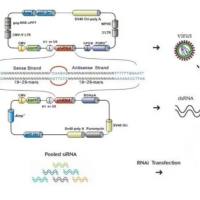
在功能基因组研究中,需要对特定基因进行功能丧失或降低突变,以确定其功能。由于RNAi具有高度的序列专一性,可以特异地使特定基因沉默,获得功能丧失或降低突变,因此RNAi可以作为一种强有力的研究工具,用于功能基因组的研究。将功能未知的基因的编码区(外显子)或启动子区,以反向重复的方式由同一启动子控制表达。这样在转基因个体内转录出的RNA可形成dsRNA,产生RNA干涉,使目的基因沉默,从而进一步研究目的基因的功能,这种技术即为RNAi技术。根据所选用序列的不同,可将其分为编码区RNAi和启动子区RNAi技术。
1.编码区RNAi技术
自1998年在线虫中发现RNAi现象以来,以基因编码区为靶序列的编码区RNAi技术已用于线虫功能基因组的研究。最初这种技术是通过注射或浸泡等方法直接导入到线虫的性腺或早期胚胎中。这些方法虽然可以关闭目的基因的表达,产生突变表型,但这种表型变化却不能遗传。这种早期的RNAi技术可以用于研究与胚胎发育有关的基因的功能,但由于细胞分裂造成dsRNA的稀释,使得这种方法在研究成体的基因功能时有一定的局限性。为弥补早期RNAi技术的上述不足,Tavernarakis等对RNAi技术进行了改进,将目的基因的靶序列以反向重复的方式,由热激启动子控制在转基因生物中表达。热激处理后,反向重复序列在细胞内开始转录,其产物会形成具发夹环结构的dsRNA,从而产生RNAi,使目的基因沉默。这种改进的RNAi技术与传统的RNAi技术相比,具有明显的优点:首先转基因可以遗传给后代,有利于突变的分析;其次dsRNA可以被诱导产生,RNAi能够在发育特定阶段出现,从而使研究发育早期必需基因在发育晚期的功能成为可能;另外,当用细胞特异性启动子控制dsRNA的表达时,可以研究特定基因在不同器官中的功能。Kennerdell J. R.和CarthewR. W.用GAL4/UAS系统控制dsRNA在果蝇中的表达,实现了诱导性或细胞特异性控制RNAi的发生。
随着应用RNAi技术研究线虫功能基因组工作的开展,研究人员对该技术在植物中应用的可能性进行了探索。加州理工大学的Chuang C. F.和Megerowitz E. M.使用此技术研究了拟南芥的AG, CLV3, AP1, PAN四个开花相关基因。结果表明使用RNAi技术可以产生功能丧失或降低突变体,其表型与以前通过其它方法鉴定的突变体类似。RNA原位杂交表明,RNAi突变体的目的mRNA显著降低。该结果说明RNAi技术亦可以成为植物功能基因组研究中的有力工具。
2.启动子区RNAi技术
M. F. Mett等证明含有启动子区的dsRNA在植物体内同样被切割成21-23核苷酸长的片段,这种dsRNA可使内源相应的DNA序列甲基化,从而使启动子失去功能,使其下游基因沉默。
由于多基因家族的各成员之间高度同源,因而使用编码区RNAi技术很难将各个成员区分开来研究,而多基因家族内的启动子序列通常比编码区变化大,采用启动子区RNAi技术有望将多基因家族的各个成员区分开来研究。这样综合编码区RNAi技术和启动子区RNAi技术的信息即可更全面地了解多基因家族地各成员的功能。 RNAi现象存在的广泛性远远超过人们的预期,对此问题的深入研究结果将为进化的观点提供有力佐证。而与其它几种进行功能丧失或降低突变的技术相比,RNAi技术具有明显的优点,它比反义RNA技术和同源共抑制更有效,更容易产生功能丧失或降低突变。而且通过
与细胞特异性启动子及可诱导系统结合使用,可以在发育的不同时期或不同器官中有选择地进行,与T-DNA技术造成的功能永久性缺失相比,这是更受科学家偏爱的。
1995年,康乃尔大学的Su Guo博士在试图阻断秀丽新小杆线虫(C. elegans)中的par-1基因时,发现了一个意想不到的现象。她们本是利用反义RNA技术特异性地阻断上述基因的表达,而同时在对照实验中给线虫注射正义RNA(sense RNA)以期观察到基因表达的增强。但得到的结果是二者都同样地切断了par-1基因的表达途径。这是与传统上对反义RNA技术的解释正好相反的。该研究小组一直没能给这个意外以合理解释。
直到1998年2月,华盛顿卡耐基研究院的Andrew Fire和马萨诸塞大学医学院的Craig Mello才首次揭开这个悬疑之谜。通过大量艰苦的工作,他们证实,Su Guo博士遇到的正义RNA抑制基因表达的现象,以及过去的反义RNA技术对基因表达的阻断,都是由于体外转录所得RNA中污染了微量双链RNA而引起。当他们将体外转录得到的单链RNA纯化后注射线虫时发现,基因抑制效应变得十分微弱,而经过纯化的双链RNA却正好相反,能够高效特异性阻断相应基因的表达。该小组将这一现象称为RNA干扰(RNA interference ,简称RNAi)。
在随后的短短一年中,RNAi现象被广泛地发现于真菌、拟南芥、水螅、涡虫、锥虫、斑马鱼等大多数真核生物中。这种存在揭示了RNAi很可能是出现于生命进化的早期阶段。随着研究的不断深入,RNAi的机制正在被逐步阐明,而同时作为功能基因组研究领域中的有力工具,RNAi也越来越为人们所重视。
RNAi的作用机制
体外实验表明:RNAi反应中,加入的dsRNA被切割为21-23核苷酸长的RNA片段,后者会使目的mRNA被切割为21-23核苷酸长的片段。从已经发生RNAi的果蝇S2细胞中,Hammond等人部分纯化了一种核酸酶,该核酸酶具有序列特异性,它仅降解与引起RNAi的dsRNA具有同源序列的mRNA。那么这种核酸酶是如何确定哪些mRNA该降解而哪些不该呢?由于在纯化该核酸酶时,可以共分离出21-23核苷酸长的dsRNA片段,这暗示该核酸酶对mRNA的切割有可能是以这些片段作模板指导进行的。根据以上的实验结果,人们提出一种RNAi作用的简单模型。当dsRNA导入细胞后,被一种dsRNA特异的核酸内切酶识别,切割成21-23核苷酸长的小片段,这些片段可与该核酸酶的dsRNA结合结构域结合,并且作为模板识别目的mRNA;识别之后,mRNA与dsRNA的有义链发生链互换,原先dsRNA中的有义链被mRNA代替,从酶-dsRNA复合物中释放出来,而mRNA则处于原先的有义链的位置。核酸酶在同样位置对mRNA进行切割,这样又产生了21-23核苷酸长的dsRNA小片段,与核酸酶形成复合物,继续对目的mRNA进行切割,从而使目的基因沉默,产生RNAi现象。通过遗传分析的方法,目前已从线虫中已分离到RDE-2,RDE-3和Mut-7等RNAi相关的基因。
RNAi技术在功能基因组中的应用
在功能基因组研究中,需要对特定基因进行功能丧失或降低突变,以确定其功能。由于RNAi具有高度的序列专一性,可以特异地使特定基因沉默,获得功能丧失或降低突变,因此RNAi可以作为一种强有力的研究工具,用于功能基因组的研究。将功能未知的基因的编码区(外显子)或启动子区,以反向重复的方式由同一启动子控制表达。这样在转基因个体内转录出的RNA可形成dsRNA,产生RNA干涉,使目的基因沉默,从而进一步研究目的基因的功能,这种技术即为RNAi技术。根据所选用序列的不同,可将其分为编码区RNAi和启动子区RNAi技术。
1.编码区RNAi技术
自1998年在线虫中发现RNAi现象以来,以基因编码区为靶序列的编码区RNAi技术已用于线虫功能基因组的研究。最初这种技术是通过注射或浸泡等方法直接导入到线虫的性腺或早期胚胎中。这些方法虽然可以关闭目的基因的表达,产生突变表型,但这种表型变化却不能遗传。这种早期的RNAi技术可以用于研究与胚胎发育有关的基因的功能,但由于细胞分裂造成dsRNA的稀释,使得这种方法在研究成体的基因功能时有一定的局限性。为弥补早期RNAi技术的上述不足,Tavernarakis等对RNAi技术进行了改进,将目的基因的靶序列以反向重复的方式,由热激启动子控制在转基因生物中表达。热激处理后,反向重复序列在细胞内开始转录,其产物会形成具发夹环结构的dsRNA,从而产生RNAi,使目的基因沉默。这种改进的RNAi技术与传统的RNAi技术相比,具有明显的优点:首先转基因可以遗传给后代,有利于突变的分析;其次dsRNA可以被诱导产生,RNAi能够在发育特定阶段出现,从而使研究发育早期必需基因在发育晚期的功能成为可能;另外,当用细胞特异性启动子控制dsRNA的表达时,可以研究特定基因在不同器官中的功能。Kennerdell J. R.和CarthewR. W.用GAL4/UAS系统控制dsRNA在果蝇中的表达,实现了诱导性或细胞特异性控制RNAi的发生。
随着应用RNAi技术研究线虫功能基因组工作的开展,研究人员对该技术在植物中应用的可能性进行了探索。加州理工大学的Chuang C. F.和Megerowitz E. M.使用此技术研究了拟南芥的AG, CLV3, AP1, PAN四个开花相关基因。结果表明使用RNAi技术可以产生功能丧失或降低突变体,其表型与以前通过其它方法鉴定的突变体类似。RNA原位杂交表明,RNAi突变体的目的mRNA显著降低。该结果说明RNAi技术亦可以成为植物功能基因组研究中的有力工具。
2.启动子区RNAi技术
M. F. Mett等证明含有启动子区的dsRNA在植物体内同样被切割成21-23核苷酸长的片段,这种dsRNA可使内源相应的DNA序列甲基化,从而使启动子失去功能,使其下游基因沉默。
由于多基因家族的各成员之间高度同源,因而使用编码区RNAi技术很难将各个成员区分开来研究,而多基因家族内的启动子序列通常比编码区变化大,采用启动子区RNAi技术有望将多基因家族的各个成员区分开来研究。这样综合编码区RNAi技术和启动子区RNAi技术的信息即可更全面地了解多基因家族地各成员的功能。 RNAi现象存在的广泛性远远超过人们的预期,对此问题的深入研究结果将为进化的观点提供有力佐证。而与其它几种进行功能丧失或降低突变的技术相比,RNAi技术具有明显的优点,它比反义RNA技术和同源共抑制更有效,更容易产生功能丧失或降低突变。而且通过
与细胞特异性启动子及可诱导系统结合使用,可以在发育的不同时期或不同器官中有选择地进行,与T-DNA技术造成的功能永久性缺失相比,这是更受科学家偏爱的。