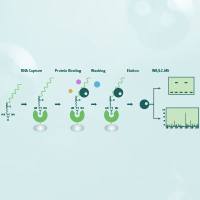
基因突变检测技术
互联网
对未知突变进行分析的方法
1.RNA酶A切割(RnaseAcleavage)
在一定条件下,异源双链核酸分子RNA:RNA或RNA:DNA中的错配碱基可被RNaseA切割,切割产物可通过跑变性胶得到分离。RNA探针可通过将相应的DNA片段克隆至含有SP6或T7启动子的载体中得到,当RNA探针上错配的碱基为嘌呤时,RNaseA在错配处的切割效率很低甚至不切割,而当错配碱基为嘧啶时,则其切割效率较高。所以如果仅分析被检DNA的一条链,其突变检出率只有30%,而如果同时分析被检DNA的正义链和反义链,则有效率可达到70%[1]。尽管RNaseA切割法有其局限性,如需使用同位素,需将PCR产物克隆至表达载体上,从而增加了操作的复杂度,但由于它能对1~2kb的片段进行检测,并能确定突变位置,且无需使用有害化学试剂,故仍被频繁使用。
2.变性梯度凝胶电泳(DGGE)
该方法的原理是:双链DNA分子在一定变性剂浓度的凝胶上电泳时,会在一定的时间发生部分解链,导致电泳迁移率下降,当正常的DNA分子和发生突主的DNA分子之间即使只有一个碱基对的差异时,也会在不同时间发生部分解链,从而被分离成两条
3. 杂合双链分析法(HA)
由突变和野生型DNA形成的异源杂合双链DNA在其错配处会形成一个凸起,在非变性胶中电泳时,会产生与相应的同源双链DNA不同的迁移率,从而使两者被分离开。该方法原理与单链构象多态性(SSCP)相似,只不过SS-CP分离的是单链,而HA法分离的是双链。HA法简单迅速,但只适用于200~300pb的片段,且不能确定突变位置,检出率只有80%左右,有人建议将HA法和SSCP法联合使用,可能将检出率提高到接近100%[4],也有科研工作者建议使用缺失的野生型等位基因来提高该方法的灵敏度
4. 化学切割错配(CCM)
化学切割错配是在Maxam-Gilbert测序的基础上发展起来的一项突变检测技术,在DNA∶DNA或DNA∶RNA异源杂合双链核酸分子中,错配的C能被羟胺(hydroxylamine)和哌啶(piperidine)切割,错配的T能被四氧化锇(Osmiumtetroxide)所切割,切割产物跑变性胶电泳即可确定是否存在突变。
只要操作得当,不会发生非特异性切割,如果对正义链和反义链都进行分析,可使检出率达到100%;使用荧光检测系统将会大大增强该方法的灵敏度,从而可检测出十个细胞中的一个突变细胞[7]。该方法的最大优点是能对长至2kb的片段分析,并能确定突变位置,缺点是步骤多,费时,且需接触有毒的化学物质。
5.碳化二亚胺检测(Carbodiimide,CDI)
与CCM法相似,CDI能够对错配的G和T进行修饰,当用标记的引物对CDI修饰过的双链DNA进行DNA合成时,延伸会在修饰过的碱基处终止下来,合成产物跑变性聚丙烯酰胺凝胶电泳即可检测出是否有突变。该方法的最大优点也是能对1~2kb的片段进行分析,突变检出率达到100%,能确定突变位置,并且CDI是一种没有毒性的物质,有报道曾用该法检测出了7.2kbDNA片段中的一个点突变,但当一个DNA片段中存在多个突变时,该方法就不适用。
6.酶促切割错配(EnzymeMismatchCleavage,EMC)
EMC是一种与RNaseA切割相类似的方法,T4核酸内切酶 是一种分解酶(resolvase),能够识别12种错配的碱基并在错配碱基的附近进行切割,酶切产物跑变性胶或中性胶即可检测出是否有突变[9],电泳产物的检测可用放射自显影,也可用银染。由于该法能对DNA∶DNA异源杂合分子进行分析,因而省去了体外转录的步骤,其最长检测片段可达到1.5kb,该法的缺点是存在非特异性切割现象,并且对有些错配碱基的识别不是100%,因此在每次检测时都必须设有一个内对照。
7.单链构象多态性(Single-StrandCon-formationalPolymorphism,SSCP)
该方法的原理是[10]:单链DNA在中性条件下会形成二级结构,这种二级结构依赖于其碱基组成,即使是一个碱基的不同,也会形成不同的二级结构并引起在非变性电泳条件不同的电泳迁移率
8.切割片段长度多态性
(CleavageFrag-mentLengthPolymorphismCFLP)CFLP技术是在限制性酶切片段长度多态性(RELP)和SSCP基础上发展起来的一种基因突变检测技术,其原理是:双链DNA变性后形成的单链在中性条件下形成二级结构(包含多个折叠的发夹状结构),正如SSCP一样,这种二级结构取决于DNA的核苷酸组成和顺序,即使有一个碱基的差异,也会形成不同数目的折叠状发夹结构,而CleavaseI外切酶能识别这种发夹结构,并在该结构的附近进行切割,切割产物跑变性聚丙烯酰胺凝胶检测,突变的DNA将出现与正常对照不同的酶切图谱
9.DNA测序(Sequencing)
由各种突变检测技术检测到的突变最后都得由测序来确定突变类型及突变位置,而且测序法检测突变的效率达到100%,基于此,有科研工作者提出对一些比较小,外显子比较少的基因的突变检测直接用测序来进行,如CMTX的CX32基因的突变分析[16],但该方法需要较多的劳力,且需接触同位素,由PEABD公司推出的DNA自动测序仪使用四色荧光标记代替了原来手工测序的同位素标记,并且测序和分析数据的时间也大大缩短,但缺点是花费昂贵,所以对一些较大的,外显子较多的基因不宜用测序法直接检测突变,同时也不适用于临床对大量的标本进行检测,此外,测序也不能被当成是突变检测的金标准,杂合突变、胶压缩、GC富集区的存在等问题使得很难通过一次测序获得精确的数据。
10.双脱氧指纹图谱法(DideoxyFinger-printing,ddF)
由于SSCP检测方法的灵敏度受到电泳温度、PCR片段大小等因素的影响,而直接DNA测序的方法又费时、费钱,因此有研究工作者在实际应用中将SSCP和Sanger双脱氧测序法结合起来形成一种新的突变检测方法,即ddF[16]。该方法的基因原理是:将PCR产物回收纯化后用两个引物分别做Sanger双脱氧测序反应,但不同的是仅加入一种双脱氧终止剂,反应产物跑中性聚丙烯酰胺凝胶电泳如果确有突变的话,在病人的电泳图谱上将会丢失或获得一条带或者至少有一条带的迁移率会发生改变。在ddF方法中,DNA链的迁移率不仅取决于其二级结构,而且与其大小相关,因而较SS-CP法更为灵敏。
MartincicD等曾经将SSCP法和ddF法进行了比较,发现ddF能将10种已知的p53抑瘤基因突变全部检测出来,而SSCP法只能检测到10种突变中的6种。ddF法的灵敏度不受电泳温度的影响,所能检测到10种突变中的6种。ddF法的灵敏度不受电泳温度的影响,所能检测的PCR产物长度也能达到近500bp,并且该法还能对突变位置进行相对定位。该法的主要缺点是需要使用同位素,同时对回收的PCR产物纯度要求较高,非特异的PCR产物将严重影响到最后结果的分析。
11.错配接合蛋白检测
从Ecoli中分离的一种DNA错配接合蛋白能在异源杂合双链DNA中的错配碱基处接合DNA,接合后的产物跑测序胶,可通过电泳迁移率的改变检测是否有突变[17]。该方法的优点是操作简单,可一次处理大量的样品,但据报道这种DNA错配接合蛋白对单碱基错配的接合程度不如对几个碱基错配的接合那么强,并且这种蛋白对非错配区也有一定的结合从而导致背景增高,最近有研究工作者建议将几种DNA错配接合蛋白联合起来使用将会提高突变检测的准确率[18],但总的来说该方法有待于进一步发展。
12.变性的高效液相色谱检测(DenaturingHighPerformanceLiguldChromatographyDHPLC)
DHPLC技术是一项在SSCP和DGGE基础上发展起来的新的杂合双链突变检测技术,其原理与DGE类似,即通过HPLC在部分变性的条件下可将发生错配的异源杂合双链DNA和完全匹配的同源双链DNA分离开来[21]。DHPLC技术曾被用来进行比较DNA测序[22],目前在Stanford的一个研究小组正在利用该技术检测与疾病相关的基因突变。与传统的杂合双链分析技术相比较,该技术花时短,分析一个样品一般仅需5分钟,不需使用放射性同位素,自动化程度高,但是需要另处购买一台HPLC仪器。虽然目前该技术主要用来检测200~300bp大小的DNA片段,长的DNA片段的检测尚未见报道,但研究工作者对此持乐观态度。
13.DNA芯片技术(DNAchip)
DNA芯片技术是90年代以来发展起来的一项新技术,该技术结合了集成电路计算机、半导体、激光共聚集扫描,荧光标记探针和寡核苷酸DNA合成等技术,充分体现了生物学技术与其它重要的作用,如基因定位,DNA测序,物理图谱和遗传图谱的构建等,同样,在基因突变检测方法,DNA芯片技术的前景也令人鼓舞,其基本原理是:许多已知顺序的寡核苷酸DNA被排列在一块集成电路板上,彼此之间重叠一个碱基,并覆盖整个所需检测的基因,荧光标记的正常DNA和突变DNA分别与两块DNA芯片杂交,由于至少存在一个碱基的差异,正常DNA和突变DNA将会得到不同的杂交图谱,通过共聚焦显微镜分别检测两种DNA分子产生的荧光信号即可确定是否存在突变。