制备动物模型的基因修饰技术
赛业
摘要:动物模型是现代生命科学研究的重要工具,特别是基因工程小鼠和大鼠,在基因功能研究、人类生理病理机制研究及新药研发中起着不可替代的作用。近几年来,制备动物模型的基因修饰技术层出不穷,这不仅包括传统ES打靶、TALEN、CRISPR/Cas9,还有TetraOneTM 基因敲除新技术。本文将对这四种技术的原理、特点、缺陷、未来发展趋势作系统的介绍和对比,并辅以具体案例说明。
自25年前小鼠胚胎干细胞(ESC)发现以来,科学家已经从ES细胞产生了约25,000种基因修饰小鼠,这使得传统的ES打靶技术成为基因组编辑最为重要的技术之一,而该项技术的发现者也获得了2007年诺贝尔奖。一方面,传统ES打靶技术与TALEN和CRISPR/Cas9新技术相比,其劣势明显:ES细胞产生基因修饰小鼠的时间较长,并且有生殖系传递失败的风险。另一方面,新一代核酸酶基因编辑技术TALEN和CRISPR/Cas9虽然技术简便快速,没有物种限制,但由于其脱靶效应至今无法避免,制备出来的动物经常出现马赛克现象,从而导致后续的动物传代鉴定工作量巨大且难以胜任大片段的基因敲入,所以还远远谈不上成熟。
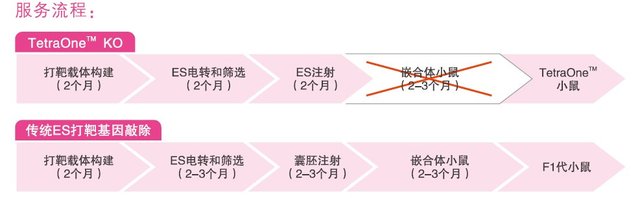
新技术TetraOneTM,它不仅保留了传统ES打靶技术的优点——修饰效果精确可靠,更是将漫长的传统ES打靶技术制作周期(需时8-12个月)压缩到了与核酸酶技术一致(近需4-7个月),通过专利的ES控制和注射技术,可以为研究者提供更好的生殖遗传保障。TetraOneTM技术是目前基因组编辑技术中最为值得关注的新一代革命性技术,它使更快、更准、更有保障的构建基因打靶小鼠成为可能。
1. 基因修饰技术的背景和应用介绍
1.1传统ES打靶基因敲除:技术成熟、修饰准确、效果稳定,但制作周期长达一年
基于胚胎干细胞的同源重组技术俗称“传统的基因打靶技术”。因具有技术成熟、修饰准确、效果稳定等优点而受到众多科学家一致好评,此外,世界上所有重要的的小鼠动物模型均通过此技术制备。尽管新兴核酸酶敲除技术如ZFN、TALEN、CRISPR/Cas9等相继出现,但基于胚胎干细胞的同源重组技术依然是唯一可以满足所有基因组修饰要求的技术。
基因打靶就是通过同源重组技术将外源基因定点整合进靶细胞基因组上某一确定的位点,以达到定点修饰改造染色体上某一基因的目的。基因打靶技术目前已被广泛认为是一种理想的特定修饰与改造生物体遗传物质的最佳方法,其中包括了多种不同的基因敲除和敲入系统,特别是条件性和诱导性基因打靶系统的建立,使得对基因在时间和空间上的靶位修饰更加明确、效果更加精确可靠。
1.2 TALEN基因敲除:周期短,但存在马赛克现象和脱靶现象
TALEN 技术是一种新的分子生物学工具。科学家发现,来自一种植物细菌的TAL蛋白的核酸结合域的氨基酸序列与其靶位点的核酸序列有恒定的对应关系。利用TAL的序列模块,可组装成特异结合任意DNA序列的模块化蛋白,从而达到靶向操作内源性基因的目的,它克服了ZFN方法不能识别任意目标基因序列,以及识别序列经常受上下游序列影响等问题,且具有ZFN相等或更好的灵活性,使基因操作变得更加简单方便。但因为脱靶的问题,利用TALEN技术进行小鼠的基因修饰仍然无法取代传统技术。加上存在马赛克现象,所以在做基因敲入(KI)和其他应用时候也必须谨慎的进行检测。
1.3 CRISPR/Cas9基因敲除:周期短,但存在马赛克现象和脱靶现象
CRISPR/Cas9(Clustered Regularly Interspaced Short Palindromic Repeats)是最新出现的一种由RNA指导Cas核酸酶对靶向基因进行特定DNA修饰的技术。 CRISPR 是细菌和古细菌为应对病毒和质粒不断攻击而演化来的获得性免疫防御机制。在这一系统中,crRNA(CRISPR-derived RNA)通过碱基配对与tracrRNA(trans-activating RNA)结合形成双链RNA,此tracrRNA/crRNA二元复合体指导Cas9蛋白在crRNA引导序列靶定位点剪切双链DNA达到对基因组DNA进行修饰的目的。CRISPR/Cas9系统能够对小鼠和大鼠基因组特定基因位点进行精确编辑, 目前已经成功应用于大小鼠基因的KO/KI模型制备,其原理也是对目的片段产生特异的切割后,修复过程中产生移码突变或者是片段缺失,或者是在切开位置产生片段插入等现象。
1.4 TetraOneTM基因敲除:技术成熟、修饰准确、效果稳定,制作周期只需6个月
TetraOneTM基因敲除是业界推出的新技术,沿用胚胎干细胞同源重组的技术体系,采用独特的建系和基因改造技术建立了具有遗传优势的TetraOneTMES细胞系,通过胚胎发育前期的显微注射,使TetraOneTMES细胞100%代替内源ES细胞,实现跨越“嵌合体”阶段从而快速获得去Neo杂合子小鼠的基因打靶专利技术。
TetraOneTM 技术是基于胚胎干细胞技术的金标准,通过同源重组技术将外源基因定点整合进靶细胞基因组上某一确定的位点,以达到定点修饰改造基因组某一基因的目的。同时TetraOneTM 技术还具有核酸酶技术周期短的优势,将传统ES打靶技术的周期缩短一半,并且不会像核酸酶技术一样带来脱靶效应和马赛克现象,在核酸酶技术没有成熟之前,TetraOneTM技术将是研究者最佳的选择。
2. 不同技术系统的对照:
2.1 不同的基因修饰技术对比分析:
| 技术类型 | 传统ES打靶技术 | TetraOneTM打靶技术 | TAELN | CRISPR/Cas9 |
| 技术方法 |
ES细胞的同源重组 囊胚注射 |
TetraOneTM ES细胞同源重组TetraOneTM注射技术 |
核酸酶技术 原核注射 |
核酸酶技术 原核注射 |
| DNA载体 | 利用同源臂进行同源重组,并利用正负筛选基因,筛选阳性克隆 | 利用同源臂进行同源重组,并利用正负筛选基因,筛选阳性克隆 |
TALE vector pair/FokI-dimer nuclease TALEN识别序列和FokI酶 |
gRNA vector/Cas9 nuclease gRNA识别序列和Cas9核酸酶 |
| 基因随机插入与脱靶效应 | 无 | 无 | 会有一点概率的随机插入和脱靶效应(取决于TALEN识别序列) | 会有一点概率的随机插入和脱靶效应(取决于gRNA识别序列) |
| 结果再现性 | 非常好 | 非常好 | 一般 | 一般 |
| 制作周期 | 8-12月 | 6-8月 | 5-7月 | 5-7月 |
| 技术应用的研究范围 | 完全性基因敲除,条件性基因敲除,基因定点插入,点突变研究,人源化研究 | 完全性基因敲除,条件性基因敲除,基因定点插入,点突变研究,人源化研究 | 完全性基因敲除,基因定点插入,点突变研究,人源化研究 | 完全性基因敲除,基因定点插入,点突变研究,人源化研究 |
| 物种限制 | 小鼠 | 小鼠 | 无 | 无 |
| 成果保证 | 有 | 有 | 基因敲除有保证,其他用途有不确定性 | 基因敲除有保证,其他用途有不确定性 |
| 技术流程要点 | DNA打靶载体,ES阳性克隆,细胞注射,嵌合体鼠获得,F1鼠获得,纯合鼠获得 | DNA打靶载体,ES阳性克隆,细胞注射,去Neo杂合子鼠,纯合鼠获得 | 载体构建,F1杂合子鼠(大鼠与小鼠)获得 | 载体构建,F1杂合子鼠(大鼠与小鼠)获得 |
| 筛选繁殖 | 无需特殊工作,可以直接进行后续试验 | 无需特殊工作,可以直接进行后续试验 | 需要排除马赛克现象对后续生殖遗传的影响,并排除脱靶效应的影响 | 需要排除马赛克现象对后续生殖遗传的影响,并排除脱靶效应的影响(KI需要考虑随机插入的影响) |
综合上表,不同的基因修饰的技术有不同的使用方向,研究者应该依据自己的需求,谨慎进行选择。
3. 各种基因修饰技术的后续繁殖的对比
3.1 不同基因修饰技术存在特殊现象和原因对照表
| 技术类型 | 技术原理 | 随机插入现象 | 脱靶现象 | 马赛克现象 |
| 传统ES打靶技术 | 大片段的同源重组 | 可以排除所有的随机插入,所有随机插入的克隆都可以被Southern Blot检测到并排除 |
无脱靶风险 因为同源重组技术是一个极度保守的技术,只有几个碱基的差异就同源重组发生的概率就会大大下降 |
无 动物由单一克隆发育而成,修饰的基因型一致 |
| TALEN技术 |
KO:核酸酶定点切割 KI:核酸酶定点切割后产生DNA断口,相对提高该位点的短片段同源重组效率 |
无法排除风险 特别是在做KI的应用时候,线性化的DNA会产生较高概率的随机插入 |
无法排除风险 TALEN识别序列较短,获得的动物必须进行脱靶效应的检测 |
存在 如果核酸酶起作用不是在胚胎的单细胞阶段,就会出现一个动物有多种修饰基因型的现象 |
| CRISPR/Cas9技术 |
KO :核酸酶定点切割 KI:核酸酶定点切割后产生DNA断口,相对提高该位点的短片段同源重组效率 |
无法排除风险 特别是在做KI的应用时候,线性化的DNA会产生较高概率的随机插入 |
无法排除风险 gRNA识别序列较短,获得的动物必须进行脱靶效应的检测 |
存在 如果核酸酶起作用不是在胚胎的单细胞阶段,就会出现一个动物有多种修饰基因型的现象 |
| TetraOneTM 技术 | 大片段的同源重组 | 可以排除所有的随机插入,所有随机插入的克隆都可以被Southern Blot检测到并排除 |
无脱靶风险 因为同源重组技术是一个极度保守的技术,只有几个碱基的差异就同源重组发生的概率就会大大下降 |
无 动物由单一克隆发育而成,基因型一致 |
3.2CRISPR/Cas9等核酸酶技术制备的小鼠的马赛克现象对后续研究的影响?
案例1:
Detailed Phenotypic and Molecular Analyses of Genetically Modified Mice Generated by CRISPR-Cas9-Mediated Editing.
PLOS ONE. 2015 Jan 14.
将Tyr(黑色素基因:双位点突变小鼠毛发为白色,野生型和单位点突变小鼠毛发为黑色,同义突变小鼠毛发为黑色)基因靶gRNA,D10A Cas9 mRNA,donor质粒注射到B6(Tyr+/+)受精卵细胞中,得到的Founder(mouse #4.9)发现有3种基因突变型,其毛色呈典型的马赛克的镶嵌现象:
案例2:
Somatic mosaicism and allele complexity induced by CRISPR/Cas9 RNA injections in mouse zygotes.
Developmental Biology. 2014 Jun 20.
将Tyr(黑色素基因:双位点突变小鼠毛发为白色,野生型和单位点突变小鼠毛发为黑色)基因靶向gRNA,Cas9 mRNA注射到Tyr 杂合(B6CBAF1/J x FVB/NJ) 受精卵,得到的founder鼠中有11%的白化小鼠,21%的黑色毛发鼠,68%的小鼠存在马赛克现象。得到的白化小鼠也存在着基因镶嵌现象,在一个白化鼠(mouse #30)上可发现5中突变基因型:
结论:
从上述两个针对比较容易观测的毛色基因Try的敲除效果看,都出现了显著的马赛克现象。而很多基因并不能像毛色基因Try这么容易观察到,而且常规的突变检测方法都是以尾巴或者爪子样品为模板,通过扩增打靶位点周边的一段短序列进行测序分析,一些嵌合度低和一些大片段删除或插入的突变型因此被漏检测。因此即使看起来突变型一致的Founder鼠也不能被认为是等同的,应当作为一个独立的谱系进行传代。
案例3:
这些对同一个位点的不同突变组成的“等位基因”在生殖传递时并不遵循孟德尔遗传分离定律,一些在个体中所占比率比较低的基因型将很难传递到子代中。
如案例1中的文章对mouse #4.8的Allele 4和mouse #4.9的Allele 3:
表格来自:
Detailed Phenotypic and Molecular Analyses of Genetically Modified Mice Generated by CRISPR-Cas9-Mediated Editing.
PLOS ONE 2015 Jan 14.
结论:
马赛克现象无疑增加了Founder鼠的背景复杂性,难以解释基因型和表现型间的对应关系。因此,即使看起来检测到突变型一致的Founder鼠也不能被认为是等同的,都应当作为一个独立的谱系进行传代。而这个也将大大增加后续动物交配和繁殖的困难和工作量。
3.3 CRISPR/Cas9等核酸酶技术制备的小鼠如何获得纯合子?
纯合子动物的获得大致需要经过以下步骤:首先需要对获得的Founder鼠进行组织样品的测序分析,并通过序列比对,可以初步判断各个小鼠的基因型,但是不能排除是否存在马赛克现象,所以一般不直接将获得的Founder鼠进行交配。而是需要将各个Founder鼠与野生型(WT)鼠进行交配,并对每个获得的F1小鼠进行测序检测,通过序列比对可以判断是否属于同一基因型。再将获得的同一基因型的小鼠进行交配,就有可能获得纯合子鼠。具体的操作过程如下:
3.4 CRISPR/Cas9等核酸酶技术制备的小鼠如何进行Off-target检测?
David R. Liu等在2013年利用体外筛选和高通量测序检测CLTA基因的四个靶位点(八条gRNA)的脱靶效应,发现四个靶位点均存在脱靶现象,脱靶效率最高达84%。CRISPR/Cas9与靶位点识别的特异性主要依赖于gRNA与靠近PAM处10-12bp碱基的配对,远离PAM处8-10bp碱基的错配对靶位点的识别影响不明显。
具体详见:
High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity.
Nat Biotechnol 2013 Aug 11.
在利用CRISPR/Cas9等核酸酶技术制备模式小鼠的时候,研究者一定要重点考虑Off-target(脱靶现象)的问题,而对这个问题的分析也是所有发高水平文章的一个必备材料。目前对Off-target的控制和分析的方法有两种,一种是在实验进行之前在体外对gRNA序列进行的检测;另外一种是在获得模式动物之后,对模式动物进行检测分析,筛选没有脱靶现象的动物。
方法一:针对gRNA的筛选降低脱靶的方法
目前在Off-target检测方面尚未有一个金标准。通常做法是,假定脱靶位点与靶位点相似,利用计算机预测潜在的脱靶位点,并对脱靶位点进行PCR扩增和Sanger测序,分析潜在脱靶位点的突变情况。这种方法的局限性在于无法客观真实地检测基因组内的脱靶效应。2015年,Shengdar Q Tsai在Nature Biotechnology 33, 187–197 (2015)发表的genome-wide, unbiased identification of DSBs enabled by sequencing (GUIDE-seq)结合了高通量测序技术,可在基因组范围内检测CRISPR/Cas9的脱靶效应。GUIDE-seq利用一种短双链寡核苷酸来标记CRISPR-Cas诱导的脱靶断裂,测序这些标签所在的基因组区域,就能够确定脱靶突变的位置。Shengdar Q Tsai的研究表明,就算某个脱靶突变的出现频率低至0.1%,GUIDE-seq也能够检测得到。脱靶的研究和风险评估正是学术研究的一个重要热点。
适用范围:
这种方法适合对gRNA的脱靶效应控制非常严格的情况下,例如临床研究和体内研究等;而且应用方面更加适合只针对固定一个靶点的gRNA的脱靶分析。由于这个方法的费用非常高,工作量巨大,需要结合高通量测序和分析技术,国内模式动物的制备过程中没有人使用。
方法二:针对CRISPR/Cas9技术制备动物的检测方法
具体的操作过程如下:
1、Off-target位点的预测
使用CRISPR Design(http://crispr.mit.edu/)设计gRNA时,软件会自动给出该gRNA潜在的Off-target位点,如下,
软件分析出286个Off-target位点,有34个位点位于功能基因上:
也可以使用NCBI的Primer-BLAST功能搜索gRNA-NGG的相似序列,找出潜在Off-target位点。
2、在Off-target位点上下游设计引物进行PCR扩增,并对PCR产物进行测序,分析Cas9在Off-target位点是否进行切割进而引起Off-target位点基因突变,如下(部分结果)
| 位点 | 序列 | 基因 | 突变率 |
| gRNA1(on-target ) | GAAGGGATTCTCCCAGGCCCAGG | Rosa 26 | 12/14 |
| OT1-1(off-target) | GAAGGGATTCTCCCTGGCCCCGG | Fermt1 | 0/10 |
| OT1-2(off-target) | GAATGGATTCTCCCAGCGCCAGG | Fam105a | 0/8 |
| OT1-3(off-target) | AAAGGGACTCTCCCAGGACCAGG | Crocc | 3/9 |
| OT1-4(off-target) | ATGAGGATTCTCCCAGGCCCTGG | Fam5b | 0/10 |
| 备注:红色字体为错配碱基,下划线为PAM序列(NGG) |
gRNA1在On-target位点的切割效率为85.7%;对获得的Founder鼠(部分)进行了4个潜在Off-target位点的活性检测
(OT1-1:1个碱基错配;OT1-2:3个碱基错配;OT1-3:3个碱基错配;OT1-4:4个碱基错配);发现9个Founder鼠中有3个小鼠在OT1-3(3个碱基错配)位点发生Cas9切割现象
从上述结果可以看出,gRNA在存在3个错配碱基的情况下仍有很高的活性。因此在对基因突变模型鼠进行功能分析前必须对其进行深入全面的Off-target分析以排除Off-target位点突变对表现型的干扰。需要注意的是这种检测方法具有片面性,无法检测到未知功能区域,同时Off-target位点预测的准确性也受到软件算法的影响,不能很好的反映基因座组内的实际Off-target情况。