SLAM-seq给您的不仅仅是RNA-seq!
北京启衡星生物科技有限公司
SLAM-seq—RNA合成及降解代谢动力学分析,基因动态表达研究的首选
基因表达是一个动态的过程,取决于细胞内环境的稳态以及对环境的适应性,基因信息调控的转变可能会导致人类疾病的发生。这些基本生物学进程的背后是一个非常精密的分子调控事件,它们控制着RNA转录,加工和降解相关的动力学(图1)。了解基因调控的分子基础需要以转录特异性和系统的方式深入了解RNA合成和降解相关动力学。但是,对于常规的RNAseq,我们只能获得终点水平的RNAs丰度信息,无法区分及定量新合成的新生RNAs水平,因此,常规的RNAseq不能对RNA的合成和降解相关的动力学进行分析。
图1 mRNA水平由转录、加工处理以及降解所决定
RNA动力学研究
为了对RNA的动力学进行分析,研究者们就开发了多种方法来分析新合成的(nascent) RNA;这些方法揭示了在启动子处的差异转录程度,表明RNA聚合酶II(Pol II)在启动子附近的暂停是基因表达的关键调节步骤,证明了nascent RNA有直接调节转录的作用,并表明其序列和结构影响转录的延伸、暂停和停顿,以及发挥染色体修饰结合和增强子的作用。nascent RNA- seq方法旨在区分新近转录的RNA和其它RNAs,这些方法可以分为4类:溴尿苷(BrU)免疫共沉淀深度测序分析( BRIC-seq ),4sU-seq,瞬时转录组测序( TT-seq),代谢标记法(SLAM-seq)(图2)。

图2. RNA动力学研究方法
RNA动力学研究不同方法的优缺点
|
方法 |
优劣势 |
|
溴尿苷(BrU)免疫共沉淀深度测序分析( BRIC-seq ) |
优点:可覆盖90%的转录本,包括不稳定以及稳定的mRNA。 缺点:需要使用BrU抗体,操作繁琐,干细胞或神经元细胞不摄取BrU,RNA input量高; |
|
4sU-seq |
优点:无需抗体,通过生物素标记和链霉亲和素磁珠进行可逆结合捕获4sU标记的RNA; 缺点:结合的效率会影响RNA降解速率的准确性 |
|
瞬时转录组测序(TT-seq) |
优点:不能均一覆盖整个转录本,只有新转录本的3’端被标记上,所以需要去除5’端RNA; 缺点:标记时间短,TT-seq可以分析半衰期小于10min的瞬时增强子、反义和启动子相关的RNA,此外也可以确定转录终止位点。 |
|
SLAM-seq |
优点:无需免疫共沉淀或生物素分离导致的结果的偏差,操作简单,RNA input量少;可结合Quantseq 3’mRNA建库试剂盒使用,节省大量的测序空间,大大节约测序成本。 |
相对于其他的方法,SLAM-seq(图3)表现超强的优势,无需繁琐且技术要求苛刻的免疫共沉淀或生物素分离等操作,操作简单,RNA input量少;可结合Quantseq 3’mRNA建库试剂盒使用,节省大量的测序空间,大大节约测序成本,使其成为RNA代谢动力学研究的首选,同时,SLAM-seq技术方法学发表在了Nature Methods上。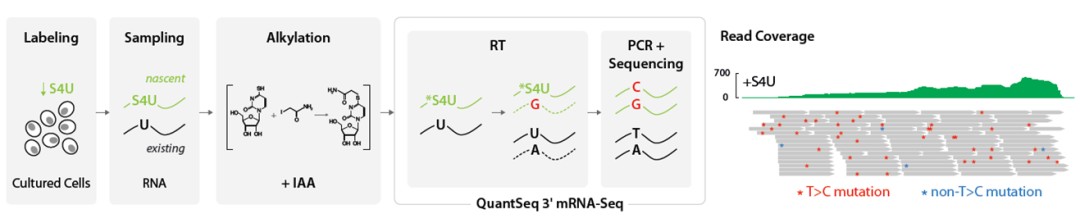
图3 SLAMseq工作流示意图。培养的细胞用4-硫代尿苷(S4U)标记新生RNA(绿色)。抽提纯化总RNA,并加入碘乙酰胺(IAA)诱导4-巯基烷基化。用QuantSeq 3’ mRNA-Seq文库构建试剂盒进行文库构建,在反转录过程中,S4U位点会反转录成鸟嘌呤 (G,红色)而不是腺嘌呤(A,黑色)。因此,在随后的数据分析过程中,可以通过T>C突变信息区分已有的RNA和新合成的RNA。
SLAm-seq可对新合成(s4U-标记)的转录本进行定量
用100μM (毒性浓度远低于EC50值)的s4U对小鼠的胚胎干细胞(mESC)进行标记 (图4)。RNA代谢标记24 h后,提取总RNA,并进行烷基化处理以及3 '端mRNA测序(Quant-seq)。Quantseq可对含poly A 结构的RNA生成靠近3’端序列的文库,每个转录本只产生一个文库片段,可对mRNA进行准确的定量分析。此外,3 '端测序可对特定细胞类型的非编码区域(UTR)进行重新评估注释,从而对mRNA 3’ Isoform特意性表达分析。在s4U代谢标记24小时后,mESCs通过Quant-seq流程生成SLAM seq文库,与未标记条件的比较,可观察很高到T >C转换累积 (图4b)。全转录组分析证实了这一观察(图4c)。在没有s4U代谢标记的情况下,我们观察到对于任何给定的转换,中位数比率≤0.1%,这与Illumina报道的测序错误一致,而s4U标记后具有统计学意义(P <10−4,Mann-Whitney检验),T < C转换率增加50倍 (图4c),在被覆盖基因组区域均匀分布(图4)。值得注意的是,非T > C转化率仍然低于预期的测序错误率(图4c),在没有代谢标记的情况下,用碘乙酰胺处理总RNA并不影响定量基因表达分析
。
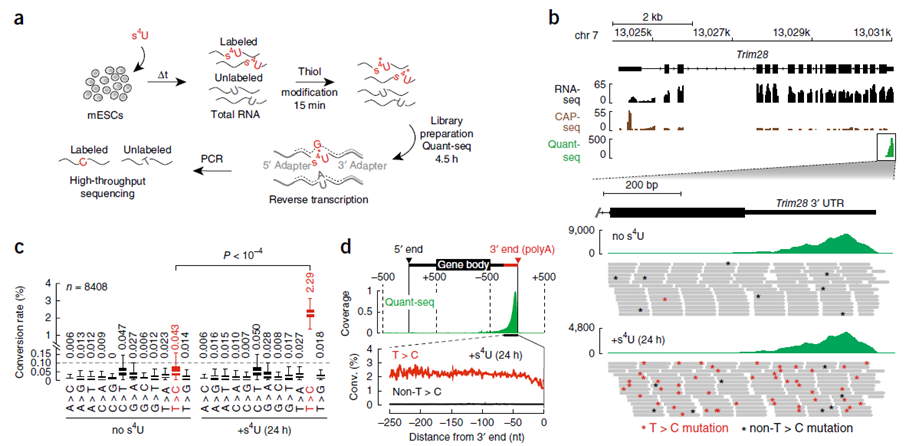
图4 SLAm-seq对s4U-标记的转录本进行定量
SLAm-seq可评估增强子的活性
在丰度相对稳定的,具有可比性的转录本中(~100 cpm),T>C reads数在特定转录本之间存在差异(图5a)。ESC特异性转录因子Sox2以及pri-miR-290-295,T>C reads数水平高,而管家基因GDPH T>C reads水平很低。这可能是因为其高稳态表达水平是通过转录本高稳定性的积累来实现的。
在ESCs中,多能状态主要由少量增强子相关的主要转录因子控制,包括Oct4、Sox2和Nanog,它们驱动维持ESC状态所必需的靶基因的表达。结果显示,在s4U-标记实验中,Oct4、Sox2和Nanog (OSN) 调控的靶基因在T > C reads中有很高的水平(图5b)。从全局来看,与4,994个近端不含OSN增强子的基因相比,含OSN增强子的2029个基因(>5 cpm)的转录本产生的 T> Creads显著增高 (Mann–Whitney test, P < 10−4; 图5c)。此外,在靠近强增强子(strongenhancer, SE)的156个基因显示出最高的转录水平(Mann–Whitneytest, P < 10−4; 图5c)。因此,SLAMseq可以通过定量转录本的表达来评估增强子的活性。
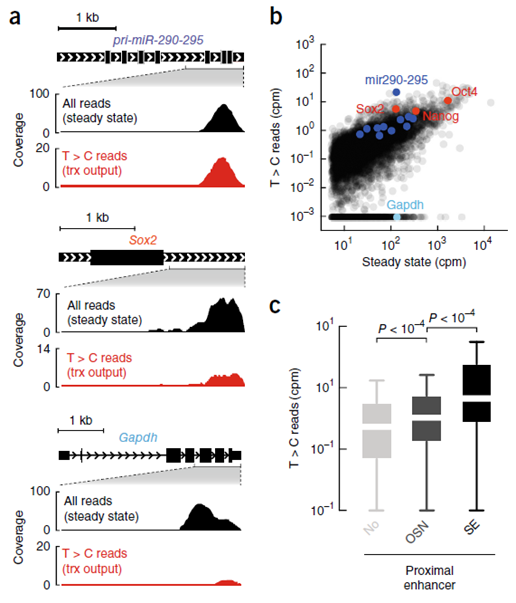
图5 mESCs中多聚腺苷酸转录本的丰度水平。
SLAm-seq提高基因差异表达检测的灵敏度
SLAMseq可以通过区分已有的RNAs以及新合成的RNAs来扣除背景信号,可通过对药物处理后新合成的RNAs进行差异分析,提高基因差异表达检测的灵敏度(图6)。
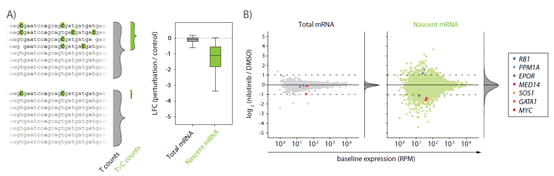
图6 SLAMseq提高基因差异表达检测的灵敏度。A) BCR / ABL抑制剂处理组(尼洛替尼)与DMSO对照组相比,K562细胞新合成RNA(T>C转换reads,绿色显示)水平显示出比总mRNA(灰色)更高的变化倍数。B)新合成mRNA(绿色)与总mRNA水平(灰色)对比分析,前者检测到更多的差异表达基因。图片来源于 Muhar et al, 2018.
SLAm-seq可对转录本稳定性进行检测
以往的研究表明,特定mRNA的半衰期与其生理功能密切相关。对检测准确度高的(r2 > 0.6)6665个转录本的半衰期进行了排序,并对666个最稳定或最不稳定的mRNA进行了富集分析。发现RNA 聚合酶II依赖性转录调节因子,这些转录本的半衰期最短,然后是翻译,呼吸电子传递,最后是氧化磷酸化(图7)。
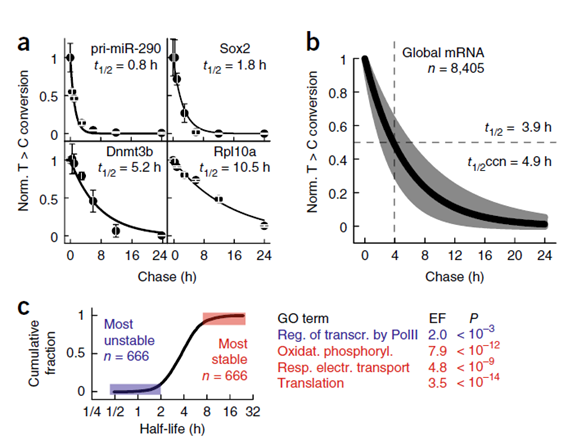
图7 mESCs中整体以及特异性转录mRNA的稳定性。
SLAm-seq可揭示mRNA稳定性的分子决定因素
在分子水平上,miroRNA是核糖核酸蛋白复合物的向导,与靶向mRNA的3’UTR互补位点相结合。microRNAs通过抑制翻译和/或促进mRNA的降解来诱发其功能。通过对转录本是否含有micrRNA以及m6A靶点进行半衰期分析,发现含microRNA靶点以及m6A靶点的转录本半衰期显著小于无靶点的转录本(图8)。
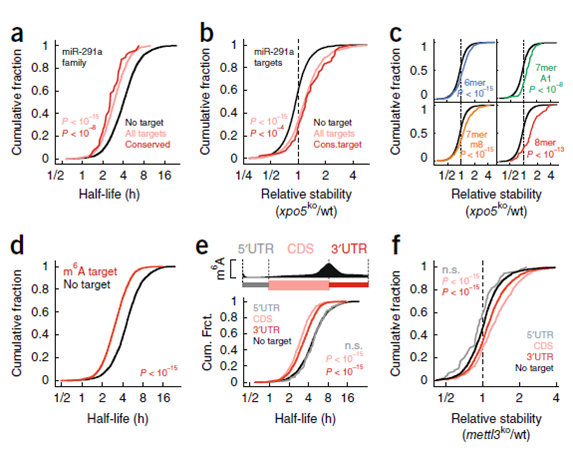
图8 mESCs中mRNA稳定性的分子决定因素。
在生物学和基础医学研究领域,技术革新总能够带来新的研究思路,推动学科的发展。SLAM-seq做为一种新的测序方式,在为RNA的研究提供了新的视角和思路。未来可以预见,SLAMseq这一强大的手段能够为生物学以及基础医学研究带来更多的福音。
Herzog, Veronika A.; Reichholf, Brian; Neumann, Tobias; Rescheneder, Philipp; Bhat, Pooja; Burkard, ThomasR.; …;
Zuber, Johannes; Ameres, Stefan L. (2017): Thiolinked alkylation of RNA to assess expression dynamics. NatureMethods. DOI:10.1038/nmeth.4435.
Muhar, Matthias;Ebert,Anja; Neumann, Tobias; Umkehrer, Christian; Jude, Julian; Wieshofer, Corinna; …; Ameres,StefanL.; Zuber,Johannes.(2018):SLAM-seqdefinesdirectgene-regulatoryfunctionsofthe BRD4-MYCaxis.Science. DOI:10.1126/science.aao2793.