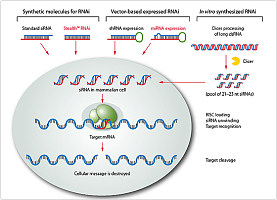
RNAi技术专题之:体外转录合成双链RNA
互联网
目前RNAi技术已经进入了生物科学研究的许多领域,成为了一种主要的生物学研究工具,发展得相当迅速,并受到了此次诺贝尔奖评审委员会 < language=Java1.1 src="/newsf/js/inpic.js">
的青睐,获得2006年诺贝尔奖医学/生理奖。然而这一才历经十个年头的技术依然对于许多研究人员来说还是很陌生的,以下是有关RNAi技术在哺乳动物细胞中应用的具体设计策略,主要来自于《遗传》杂志2005年“RNA干涉(RNAi)技术应用于哺乳动物细胞的研究策略”,希望能给从事或者准备从事RNAi相关实验的研究人员以帮助。
一、靶siRNA序列选择
靶siRNAA序列选择是RNAi实验成败的关键。哺乳动物细胞RNAi实验中,使用最广泛且最有效的是21bp siRNA。siRNA由正义链和反义链组成,两条链3’端均有2个碱基突出,一般为UU或dTdT,其中正义链的前19nt与靶基因序列相同。
1. siRNA设计的原则
SiRNA双链设计时,一般在靶mRNA起始密码下游100—200bp至翻译终止密码上游d0—100bp的范围内搜寻AA序列,并记录每个AA3’端相邻19个核苷酸作为候选si.RNA靶位点。其中AA(N19)TT是最理想的序列,若靶mRNA中无此序列,亦可选用NA(N21)或NAR(N17)YNN(R表示嘌呤,Y表示嘧啶),但在合成时,siRNA的正义链3’端需用dT—dT代替。TuscRl等建议设计的siRNA不要针对mRNA的5’和3’端非编码区,因为这些区域有丰富的调控蛋白结合位点,而UTR结合蛋白或者翻译起始复合物可能会影响siRNP(siRNA protein complex,核酸内切酶复合物)结合mRNA,从而影响siRNA干扰的效果。最后还应将候选siRNA序列在GenBank进行BLAST检索,与非同源基因具有3个或3个以上碱基相异的序列方可选用。
2. 高效siRNA的序列结构特征
除了靶序列位点的选择,siIRNA序列本身结构特征亦会影响到RNAi效率。RNAi实质上可视为一个RNA与蛋白质互作过程,包括siRNA与RISC结合、RISC的激活以及RISC与靶mRNA的结合和切割,这种互作效应的存在,往往造成siRNA链的选择具有偏爱性。Reynolds等通过对2个基因的180条siRNA序列分析,归纳出以下8个与
siRNA高效性相关的特征:G/C含量低(30%-52%),正义链3’端具较低稳定性(有利于siRNA与RISC的结合和解链),无反向重复序列(有利于减小siRNA的有效作用浓度,提高siRNA干扰效率),正义链碱基的偏爱性A19(正义链中第19位碱基为A,如下表示类同);A3;U10;无G/C(正义链中第19位碱基不为G或C);无G13。依此规则,Reynolds等设计的30条siRNA中有29条是有效的。Kumiko等亦认为siRNA反义链5’端为A/U(即有义链U/A)19和最后7个碱基中有5个A/U,会有助于RNAi的高效发挥,且正义链G/C1和序列中无连续9nt以上的GC重复片段与基因沉默效率呈显著相关。有趣的是,该实验还表明这些规则同样适用于DNA介导的RNAi(DNA-based RNAi.)实验。此外,Amarzguioui等发现正义链5’与3’端的前3个碱基中A/U含量不对称(3’端含量应高一些)和A6对siRNA 的活性亦影响很大。根据上述结果,可以初步绘制出一个高效siRNA序列结构的精细与简略示意图。
现已知道siRNA的反义链决定着靶位点识别,那么在不影响siRNA作用功效的前提下,是否可以对其正义链碱基作适当更改或修饰呢?Amarzguioui等研究siRNA不同部位对碱基突变和化学修饰的耐受性,认为siRNA的5’端相对于3’端具有对突变的较高耐受性。有学者认为正义链3’突出端的任何碱基修饰对siRNA作用效果均无明显影响。还有学者认为与反义链互补的正义链3’端发生1—4个碱基的错配反而有助于增强RNAI的活性。
二、siRNA制备方法
目前为止较为常用的5种制备siRNAs的方法包括
化学合成
体外转录
长片断dsRNAs经RNase III 类降解 (e.g. Dicer, E. coli, RNase III)
siRNA表达载体或者病毒载体在细胞中表达siRNAs
PCR制备的siRNA表达框在细胞中表达
前面3种方法包括体外制备siRNAs然后经过转染或者电转导入哺乳动物细胞后面两种方法则依赖能够表达siRNAs的DNA载体或者表达框转染到细胞中。每种方法都有自己的优点和缺点。哪种是最好的方法,其实取决于实验目的。这里简单介绍这5种方法,优点和缺点,以及它们最适合的应用。
siRNA的设计
除了方法3以外,其他的方法都在制备siRNA 前都需要单独设计siRNA序列。关于怎样设计siRNA以及siRNA设计对siRNA功能的影响,尽管我们不断掌握越来越多有关设计原则的信息。但这仍然不是精确的。通常来说,每个目标序列设计3—4对siRNAs,在后面的实验中选择最有效的那个。网上有提供免费的siRNA设计工具。
体外制备
1.化学合成
尽管化学合成是最贵的方法,但是却是最方便的——研究人员几乎不需要做什么工作。包括Ambion和Qiagen公司都可以根据用户要求提供高质量的化学合成siRNA。主要的缺点包括价格高,定制周期长,特别是有特殊需求的。由于价格比其他方法高,为一个基因合成3—4对siRNAs 的成本就更高了,比较常见的做法是用其他方法筛选出最有效的序列再进行化学合成。
最适用于:已经找到最有效的siRNA的情况下,需要大量siRNA进行研究
不适用于:筛选siRNA等长时间的研究,主要原因是价格因素
2.体外转录
通过体外转录的方法可以合成siRNAs,这样的成本相对化学合成法而言比较低,是一种性价比高的筛选siRNAs的好方法。更重要的是采用这种方法能够比化学合成法更快的得到siRNAs。以Silencer siRNA Construction Kit为例,一旦得到DNA Oligo模版(这个还是需要DNA合成的,不过DNA合成的成本就比较低了),只要24小时就可以,不需要等很久。这个方法的不足之处是实验的规模受到限制,虽然一次体外转录合成能提供足够做数百次转染的siRNAs,但是反应规模和量始终有一定的限制。而且和化学合成相比,还是需要占用研究人员相当的时间——毕竟,化学合成只需要定购就可以了。值得一提的是体外转录得到的siRNAs只要较低的浓度就可以达到化学合成siRNAs较高浓度得到的效果(0.5-20 nM vs. 50-100 nM per transfection)
最适用于:筛选siRNAs,特别是需要制备多种siRNAs,化学合成的价格成为障碍时。
不适用于:实验需要大量的,一个特定的siRNA。长期研究。
3.用RNase III 消化长片断双链RNA制备siRNA
其他制备siRNA的方法的缺陷是需要设计和检验多个siRNA序列以便找到一个有效的siRNA。而用这种方法——制备一份混合有各种siRNAs “混合鸡尾酒” 就可以避免这个缺陷。这个方法是选择通常是200—1000碱基的靶mRNA模版,用体外转录的方法制备长片断双链dsRNA ,然后用RNase III (or Dicer) 在体外消化,得到一众siRNAs“混合鸡尾酒”。在除掉没有被消化的dsRNA后,这个siRNA混合物就可以直接转染细胞,方法和单一的siRNA转染一样。由于siRNA混合物中有许多不同的siRNAs,通常能够保证目的基因被有效地抑制。
dsRNA消化法的主要优点在于可以跳过检测和筛选有效siRNA序列的步骤,为研究人员节省时间和金钱(注意:通常用RNAse III通常比用Dicer要便宜)。不过这种方法的缺点也很明显,就是有可能引发非特异的基因沉默,特别是同源或者是密切相关的基因。现在多数的研究显示这种情况通常不会造成影响。Ambion公司曾经用它的Silencer siRNA Cocktail Kit(RNase III)试剂盒成功关闭几个基因,包括c-fos, GAPDH, La, ?-actin, 和Ku-70。
最适用于:快速而经济地研究某个基因功能缺失的表型
不适用于:长时间的研究项目,或者是需要一个特定的siRNA进行研究,特别是基因治疗
体内表达
前面的3种方法主要都是体外制备siRNAs,并且需要专门的RNA转染试剂将siRNAs转到细胞内。而采用siRNA表达载体和基于PCR的表达框架则属于:从转染到细胞的DNA模版中在体内转录得到siRNAs。这两种方法的优点在于不需要直接操作RNA。
4. siRNA表达载体
多数的siRNA表达载体依赖3种RNA聚合酶III 启动子(pol III)中的一种,操纵一段小的发夹siRNA在哺乳动物细胞中的表达(1–4)。这3类启动子包括大家熟悉的人源和鼠源的U6启动子和人H1启动子。之所以采用RNA pol III启动子是由于它可以在哺乳动物细胞中表达更多的小分子RNA,而且它是通过添加一串(3到6个)U来终止转录的。要使用这类载体,需要订购2段编码短发夹RNA序列的DNA单链,退火,克隆到相应载体的pol III 启动子下游。由于涉及到克隆,这个过程需要几天的时间,同时也需要经过测序以保证克隆的序列是正确的。不过幸好载体容易大量扩增,这个优点足以弥补所有缺陷,特别是当载体在实验中确实有效的时候。
毫无疑问,siRNA表达载体的优点在于这是众多方法中唯一可以进行长期研究的方法——带有抗生素标记的载体可以在细胞中持续抑制靶基因的表达,持续数星期甚至更久。即使是对转染带有筛选标记质粒的细胞进行瞬时筛选,也有助于富集带质粒的细胞。这也可以帮助解决一些难转染的细胞由于转染效率低造成的问题。
最近多个厂家开始研究逆转录病毒和其他病毒载体用于siRNA表达,其优势在于可以直接高效率感染细胞进行基因沉默的研究,避免由于质粒转染效率低而带来的种种不便。
最适用于:已知一个有效的siRNA序列,需要维持较长时间的基因沉默,或者需要用抗生素筛选能表达siRNA的细胞。长期研究。
不适用于:筛选siRNA序列(其实主要是指需要多个克隆和测序等较为费时、繁琐的工作)
5. siRNA表达框架
siRNA表达框架(siRNA expression cassettes,SECs)是一种由PCR得到的siRNA表达模版,能够直接导入细胞进行表达而无需事前克隆到载体中。这个方法最早是由Castanotto和其同事(5)采用,包括一个RNA pol III启动子,一段发夹结构siRNA,一个RNA pol III终止位点。和siRNA表达载体不同的是,SECs不需要载体克隆、测序等颇为费时的步骤,可以直接由PCR得到,不用一天的时间。因此,SECs成为筛选siRNA的最有效工具,甚至可以用来筛选在特定的研究体系中启动子和siRNA的最适搭配!如果在PCR两端添加酶切位点,那么通过SECs筛选出的最有效的siRNA后,可以直接克隆到载体中构建siRNA表达载体。构建好的载体可以用于稳定表达siRNA和长效抑制的研究。
这个方法的主要缺点是PCR产物很难转染到细胞中。如果有适合的新型转染试剂能提高SEC的转染效率的话,那问题就可以解决了。另外,没有克隆到载体中的PCR片断并不适于大规模制备。
Silencer Express siRNA Expression Cassette Kits提供人源和鼠源的U6启动子或者人源H1启动子元件可供选择,并已经过c-fos基因抑制的检验。
最适用于:筛选siRNA序列,在克隆到载体前筛选最佳启动子
不适用于:长期抑制研究。(如果克隆到载体后就可以了)
三、siRNA的转染
将siRNA、siRNA表达载体或SECs导入哺乳动物细胞中是诱导RNAI发生的关键。有许多转染试剂可供选用,最常用的是脂质体转染试剂。某些情况下电穿孔法亦可用,但易导致较高的细胞毒性反应。对于同一细胞系,使用不同的转染试剂或方法,其效率往往会有所不同;而对于不同的细胞系,使用同一种转染试剂或方法,效果亦会不同。因此在实验过程中,有必要尝试多种试剂或方法来确定最优条件。转染效率可通过测定细胞的密度,转染的时间和siRNA与转染试剂的比例来评价。
四、实验对照设置
实验对照是衡量RNAi.实验数据可信度的一个重要因素。设立阳性对照目的是通过检测不同浓度的siRNA转染效率及其最终干扰效果,来确定合适的转染条件和最低有效的SIRNA浓度。有实验表明RISC复合物具有饱和性,且过多siRNA会导致细胞毒性和死亡。对于大多数细胞,持家基因(如GAPDH,c-cmyc, β—acion等)是较好的阳性对照,针对这些基因的高效SIRNA序列已有报道。阴性对照是用来检测RNAi的特异性。通常作为阴性对照的siRNA与选中的siRNA序列有相同碱基组成,但与靶mRNA无明显同源性。阴性对照的siRNA序列设计有2种方法,一是将特异性siRNA的序列打乱,即“零乱”siRNA(scrambled siRNA),再对其进行BLAST比对,防止与目的靶细胞中的其他基因有同源性;另一种是在特异性siRNA中引入几个错配碱基。若引入的是一个碱基错配,应需考虑它与突变mRNA(即SNP位点)结合能力,最好在设计siRNA时就避免SNP区。
五、RNAi效果检测
RNAi效果可从mRNA和蛋白质两个水平来进行量化。在mRNA水平上,Northern Blot和实时定量PCR是两种常用方法。值得注意的是在进行实时定量PCR时,cDNA合成要用oligo(dT),而不是随机引物,且预扩增片段最好位于靶mRNA序列中siRNA位点的上游。蛋白质水平可以通过Western Blot、荧光免疫检验法、流式细胞分析术和表型分析来检测。RNAi一般在转染后24小时内发生,其引发基因沉默的程度和持续时间依赖于靶蛋白质的降解率(turnover rate of the protein)、siRNA在细胞内的寿命以及其逐渐被稀释的程度。