STRING 玩转功能蛋白质组研究,高级又简单
丁香园
蛋白质不是孤立存在与生物体中的,它们之间的相互作用往往发挥着重要的生物学功能,因此,功能蛋白质相互作用研究已经越来越受到研究者的重视。STRING 是集研究蛋白质相互作用组研究、基因组研究和蛋白质组研究于一体的非常强大的软件。现在我分析数据已经越来越离不开他了。我们可以进行在线数据分析。
图 1
可以通过输入蛋白质名称,多个蛋白名称,氨基酸序列等搜索 STRING。当我们不清楚物种是什么时也不用担心,因为网站有自动生成器,物种能够被自动识别出来。这里以分析大肠杆菌中的 77 个蛋白为例。步骤如下:

图 2
1. 选择「Multiple proteins」;
2. 在蛋白名处输入所有蛋白名或基因号;
3. 在「Organism」处输入「Escherichia coli」;
4. 点击 SEARCH。
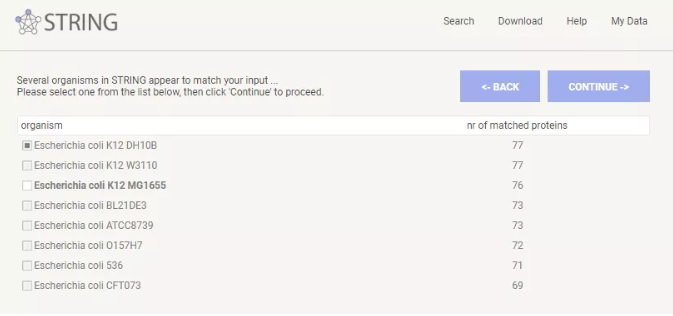
图 3
5. 下一页是确认物种页面,可以从中选择需要的物种。或者如果不确定物种时,可以选择匹配到蛋白数目最多的。点击「CONTINUE」。
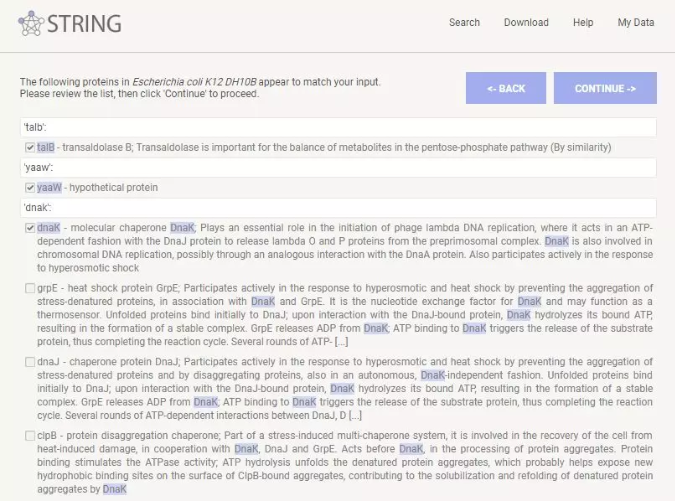
图 4
6. 下一页是确认蛋白页面,对于我们输入的每一个蛋白,网站自动进行匹配,将最有可能的蛋白排在第一位并打勾,并对于每一个蛋白进行简单的标注。这时我们就可以检查这些匹配的蛋白是不是我们所关心的蛋白。当确认了所有蛋白后就可以「CONTINUE」了。
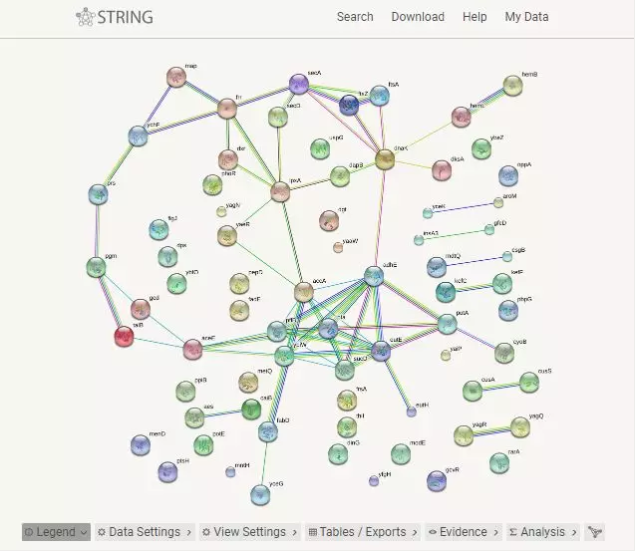
图 5
7. 这时,就会出现所有蛋白的预测相互关系网络图了。
下面我会详细介绍这张蛋白网络图和下面每一个选项的作用:
① STRING 使用弹簧模型来生成网络图像。节点被模拟为弹簧的质量和连线;通过最小化系统的「能量」来计算图像中节点的最终位置。首先,图中连线的两个节点间的物理距离没有意义。其次,虽然算法是确定的,但是新的节点添加到网络会导致新图像中节点位置完全改变。最后,虽然输入节点是抽象意义上的网络的「中心」,但它可能并不位于网络图像的中心位置。
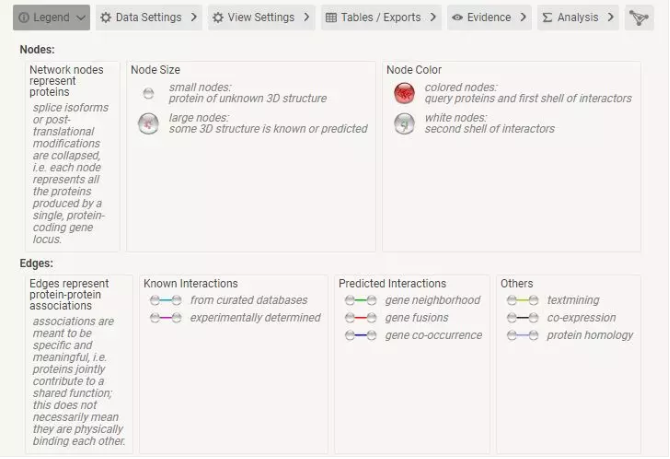
图 6
② 网络节点是蛋白质。大节点表示此蛋白的 3D 结构已知或已被预测,小节点表示 3D 结构未知。蛋白间的连线表示预测的功能关联。连线有 7 个不同颜色:
红线—基因融合
绿线—基因相近
蓝线—基因共同产生
紫线—实验证明
黄线—文本挖掘证据
浅蓝线—辅助数据库证据
黑线—基因共同表达。
线的厚度表示相互作用的置信预测度。点击节点和连线会给出蛋白质详情和证据详情。
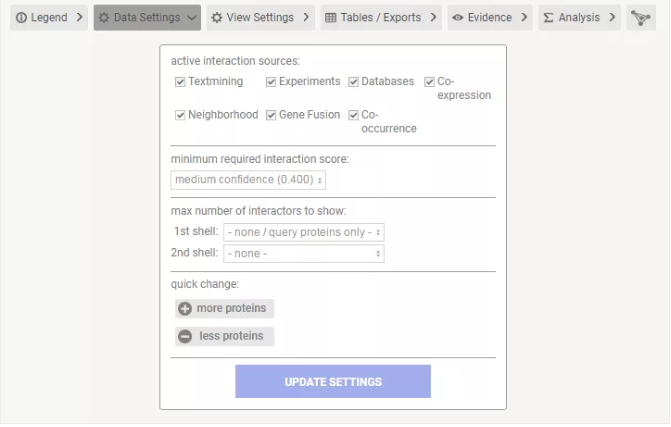
图 7
③ Data Settings
「active interaction sources」:可以选择哪种类型的证据将有助于预测分数。
「minimum required interaction score」:将置信度得分设置为阈值,使得只有大于该分的关系才包括在蛋白网络中。较低的分数意味着更多的互动,更多的假阳性。

图 8
④View Settings
「meaning of network edges」:1)证据:其中颜色表示交互证据的类型;2)置信线厚度表示数据支持的强度;3)分子作用线形状表示预测的作用模式。
「network display mode」:1)静态图像:图像是一个简单位图图像;2)交互式 svg:图像是一个可扩展的矢量图形;3)交互式闪存:蛋白网络显示在 Flash 程序中,可实现更多功能(如,聚类)。
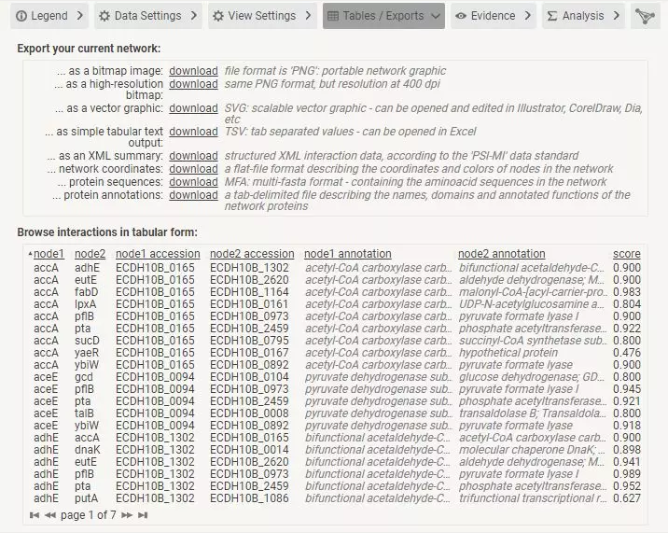
图 9
⑤Tables/Exports
可以将蛋白网络导出为以下格式:
位图:PNG 文件格式的网络图像。
高分辨率位图:PNG 格式的图像,分辨率为 400 dpi。
矢量图形:可以在 Illustrator,CorelDraw,Dia 等中打开和编辑的 SVG 格式图像。
表格文本: TSV 格式的数据。可以在 Excel 中打开。
XML 摘要:结构化 XML 格式的数据。
网络坐标:描述网络中节点坐标和颜色的平面文件格式。
蛋白质序列 - MFA:多基因格式,含有网络中的氨基酸序列。
蛋白质注释:制表符分隔的文件,描述网络蛋白质的名称,结构域和功能。
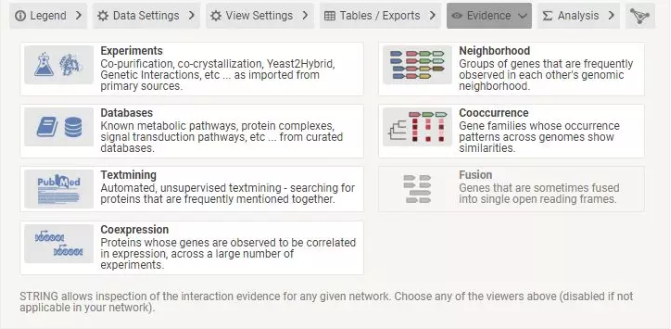
图 10
⑥Evidence
「Neighborhood」:基因组中邻近发生的基因。
「Co-occurrence」:物种间存在或不存在交联蛋白。蛋白质列在页面顶部,具有物种名称的系统发育树在左侧。在后面的网格中,物种中蛋白质用红色正方形标记。
「Fusion」:物种中的基因融合。发生融合的物种列在左边。根据页面底部的表进行着色。
「Co-expression」:在相同或不同物种中共表达的基因。红色方块表示共表达,深的颜色表示更多关联。
「Experiments」:从其他蛋白 - 蛋白相互作用数据库收集的蛋白相互作用组的列表
「Databases」:从辅助数据库收集的蛋白相互作用组的列表。
「Text mining」:从文献中提取的蛋白相互作用组的列表。
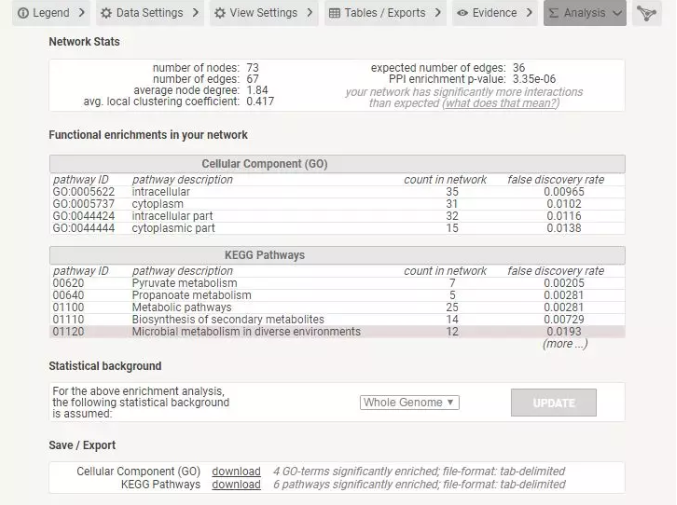
图 11
⑦Analysis
给出了蛋白网络的简要统计,如节点数和边数。平均节点度是蛋白质在网络中平均有多少相互作用。聚类系数是网络节点连接的度量。
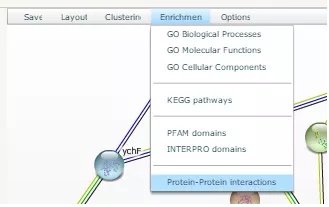
图 12
8. 最后,我们也可以对这些相互作用蛋白进行通路分析。在交互式闪存模式下,选择「enrichment」选项下我们可以进行 GO、KEGG、PFAM 等蛋白分析。选择「Clusters」,我们则可以对蛋白进行聚类分析。