图说基因点突变
丁香园
基因的体外定点突变是常用的分子实验技术,主要用于研究蛋白质结构与功能关系或者验证 microRNA 与靶基因是否结合。今天为大家介绍下基因突变质粒的构建原理与方法。
第一阶段:野生型质粒构建
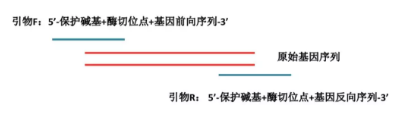
1. 引物设计:根据需要验证的基因序列,设计需要扩增的基因片段引物。引物设计方法如下:
(1)引物 F:5’ 端 - 保护碱基序列 + 限制性内切酶 1 酶切位点序列 + 基因正向引物序列 - 3’端
(2)引物 R:5’ 端 - 保护碱基序列 + 限制性内切酶 2 酶切位点序列 + 基因反向引物序列 - 3’端

通过加入基因组 DNA 模板以及设计好的引物进行目的基因的扩增,得到 PCR 产物后进行琼脂糖凝胶电泳验证条带大小,然后通过胶回收,获得纯化目的片段产物。
由于加入保护碱基,回收产物需要进行限制性内切酶切从而暴露黏性末端。
2. 线性化质粒:将原始的质粒载体,比如 pcDNA3.1 质粒,选择合适的酶切位点,比如 NheI/KpnI 进行酶切,获得线性化的质粒。
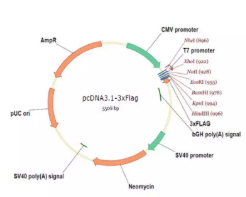
酶切体系一般选择 50ul,试剂加好之后 37°C 孵育 6~8 小时,或适当延长时间,保证质粒酶切完全。每隔一段时间振荡一下并离心以防液滴蒸发至管盖上。酶切后载体通过切胶回收线性化载体。
3. 质粒连接:将线性化的载体与目的基因的连接,推荐在 10~20μl 反应体系中进行连接反应,可以选择 T4 酶进行黏性末端的连接。
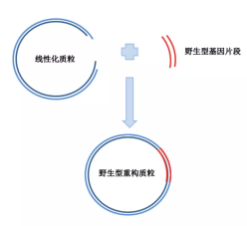
4. 转化涂板:将连接产物转化到受体菌中(一般为 DH5a),涂板,培养过夜。次日,挑取平板上单克隆菌落进行摇菌, 送菌液测序,得到野生型质粒。
第二阶段: 突变型质粒构建
在得到野生型质粒之后,下一步就是对目的基因片段进行突变啦,即将目的基因上面的一个碱基或多个碱基换成另外的碱基。
质粒突变的原理: 即通过设计引物, 并利用 PCR 将模板扩增出来, 待突变的质粒通常来源于大肠杆菌等细菌,在细菌中会被甲基化修饰,而在体外通过 PCR 扩增得到的质粒不会被甲基化。
这样用甲基化酶 DpnI 酶处理,可以消化掉待突变的质粒模板,而使通过 PCR 扩增出来的含有突变位点的质粒被选择性地保留下来。
1.突变引物设计: 向质粒进行单点或者多点突变,只需要设计一对引物,进行 PCR 的扩增,替换掉野生型基因的突变位点碱基。
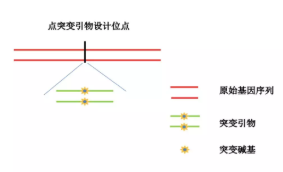
(1) 通常突变引物长度为 25~45bp, 一般都是以要突变的碱基为中心, 各加上两边的一段序列, 两边长度至少为 10–15bp,同时合成反向互补的引物。
(2) 设计引物时最好将突变引物设在目的基因的中央位置。
(3) 设计引物时需要注意 GC 含量应大于 40%,Tm 值至少达到 78℃。
2.突变质粒的扩增:突变质粒的扩增过程中,需要选用高保真酶 Pfu 酶进行质粒扩增, 防止引进新的突变。通过 PCR 过程,突变型质粒也得到了扩增。


3. 得到 PCR 产物后,将产物立即置于冰上,然后在 PCR 反应体系中加入 1 微升 Dpn I,混匀后 37℃孵育 1-2 小时。DpnI 能够识别甲基化位点并将其酶切。从大肠杆菌里提取原始质粒模板一般都被甲基化的,DpnI 可以将原始野生型质粒模板进行酶切, 而质粒扩增的突变型质粒不能被酶切从而保留下来。
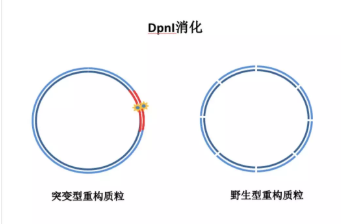
4. 最后,将酶切后产物转化入大肠杆菌,通过涂板挑取单克隆菌落后,摇菌测序,这样就可以得到突变基因的质粒啦。