兵贵神速:且看 R 软件绘制高级论文图
丁香园
曼哈顿图,学名 Manhattan plot,是全基因组关联分析(Genome-Wide Association Study, GWAS)的标配,可以展示所有染色体上的不同位置的 SNP 的显著水平,因酷似曼哈顿的海边景色(高楼林立的)所以称为曼哈顿图。
本质上,曼哈顿图是以 SNP 在基因组上的坐标/位置为 X 轴,以显著水平(通常是-log(P))为 Y 轴的散点图,所以理论上说所有能做散点图的工具都能画曼哈顿图。
但是你想一想全基因组有多少 SNP,然后还要坐成能用来发表的酷炫的样子,像 Excel 这种强(fei)大(jin)的工具还是算了吧(卡死电脑是必然的,然后调整染色体的标签,标注不同的颜色等都很麻烦)……除了是 GWAS 的标配外,你做一个 EWAS 也可以画曼哈顿图,做一个表达分析也可以做一个曼哈顿图,只要是你想要看基因组上不同的位置的都可以做。
由于二代测序技术的发展,测序的价格不断下降,GWAS 研究越来越多得应用于各种疾病的研究中,而用于做曼哈顿图的工具也开始有所增加,比如 Java 程序 Haploview, python 的 geneview 库等。
今天要跟大家介绍的是 R 语言的 2017 年 3 月 20 号更新的 CMplot 包,优点如下:
操作简单,一行代码搞定曼哈顿图;
形式多样,长方形、环形,甚至可以做出 Circos 的效果;
可以比较多个不同性质的结果;
QQ 图,density 图一网打尽。
操作流程
1 自行下载并安装 CMplot 包
在 R 中 输入 install.packages(「CMplot」),然后选择一个镜像,一般选择 China 开头的,离你所在的城市近的就好啦
2 加载 CMplot 包
输入 library(CMplot)
3 准备/读入数据
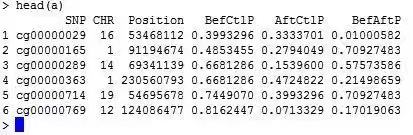
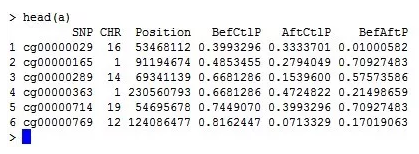
a<-read.csv(「cmplot.csv」,header=T)
数据的格式为一个至少四列的数据表:
第一列是标记名如 SNP 编号,甲基化探针名等;
第二列是染色体编号,推荐使用纯数字表示;
第三列是标记的基因组坐标;
第四列及以后各列是相关性 p 值,一种性状的相关性 p 作为一列,可以有多列。如下图所示:
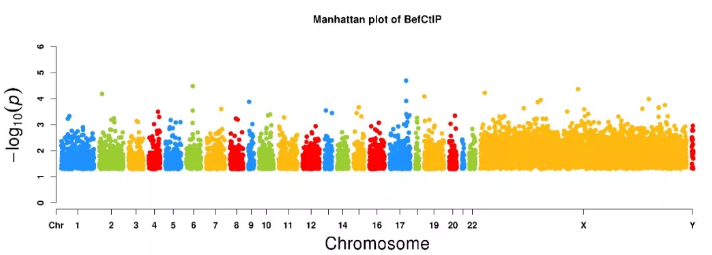
4 一行代码搞定曼哈顿图
CMplot(a)
结果如图所示:
另外 CMplot 还给出了其他形式的图:
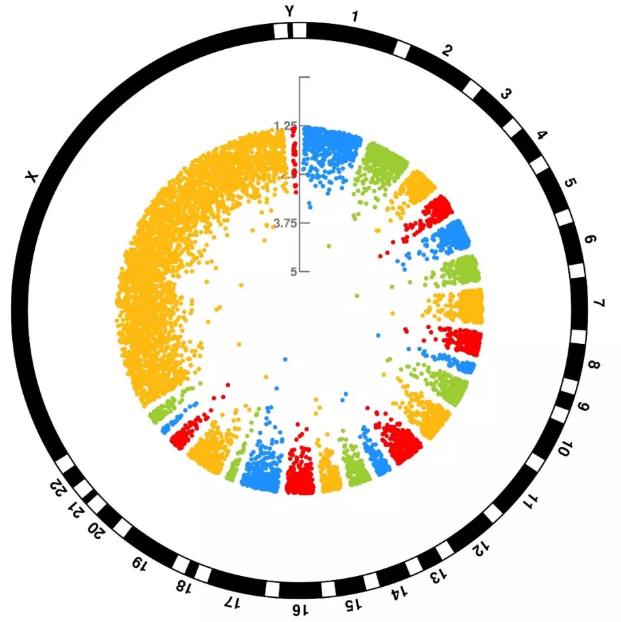
环状曼哈顿图
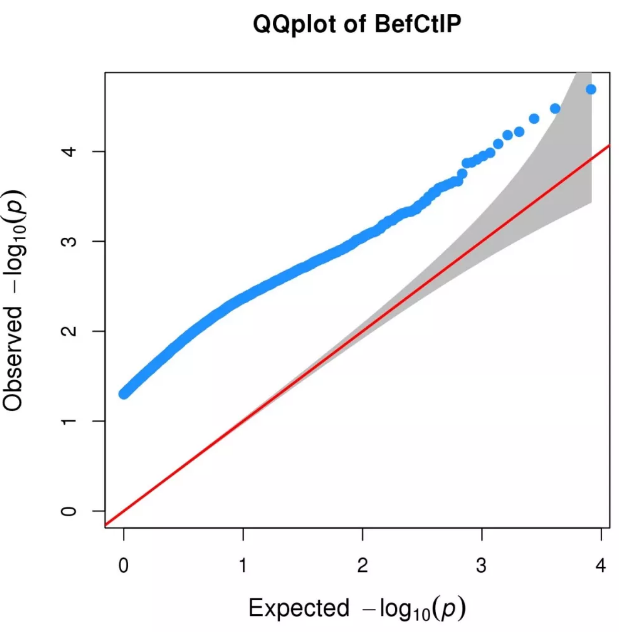
QQ 图
密度图

5 美化曼哈顿图
(已经很美了 T-T)还是说自定义吧
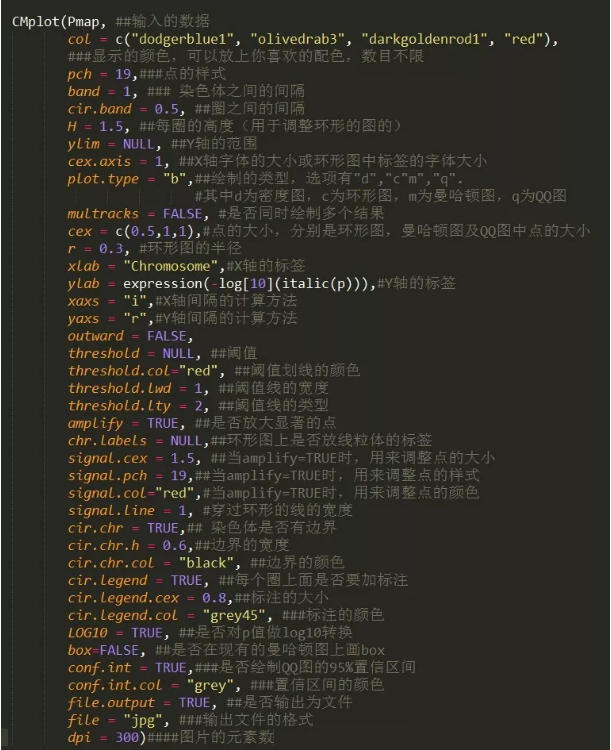
CMplot 的参数及解释 ↓
![[精选]SCI论文写作投稿资料包:100+资料& 投稿答疑30问](https://img1.dxycdn.com/p/s14/2023/1105/569/6102685706544601271.jpg!wh200)