手把手教你如何用数据库查找 miRNA 结合位点 | 实验时间
丁香园
元元上回用 targetscan 和 starbase 告诉大家查找 miRNA 结合位点,如何用数据库查找 miRNA 结合位点(今天学的是寻找基因的 3’UTR 和 miRNA 的碱基序列,最终预测 miRNA 与目标 mRNA 在 3’UTR 的结合位点)例如咱们在 starbase 看到如下结果:
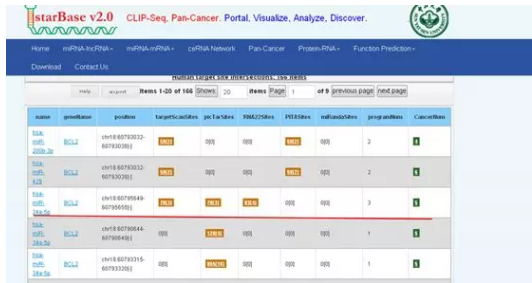
划红线的结果是 miR34A-5P 与 BCL2 存在结合位点,有三个数据库预测到这种关系。
现在咱们用 RNA22Sites 这个网站也来寻找 miRNA 结合位点。
进入这个网站:
https://cm.jefferson.edu/rna22/Interactive

可以见到两个文本框,第一个是写 miRNA 的序列,miRNA 查找基因序列一般用 miRBase 查找,第二个文本框是写预测基因的 UTR 序列,一般 miRNA 结合在 mRNA 的 3’UTR 区,所以一般下面输的是某 mRNA 的 3’UTR 序列,直接进入 NCBI 里找。
现在咱们先用 miRBase 查找 miRNA 的序列。
进入网站:
http://www.mirbase.org/search.shtml
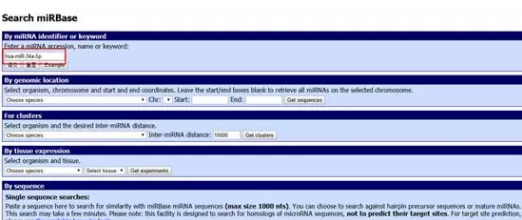
在文本框里输入 miRNA 的名字例如 hsa-miR-34a-5p, 点击提交,进入

点击进入下一页面

这个结果就能输入到第一个文本框里头
接下来到 NCBI 里找到 BCL2 的 3’UTR 区,进入 pubmed
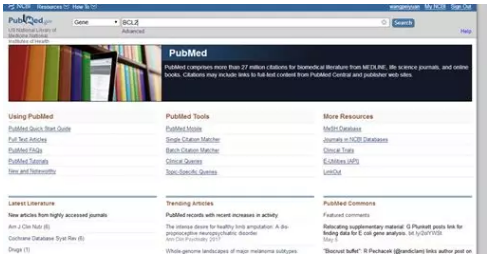
点击 search 进入下一页
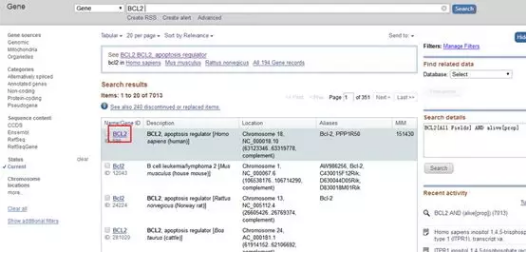
点击红色方框人源的 BCL2 进入下一页

找到这一页底下的 mRNA and Protein(s),点击红色方框的 NM000633.2

找到 CDS(这是 Coding sequence 编码序列的意思),点击它进入

上图深色的是编码区,深色上方的四百多个碱基是 5’UTR 区,深色下方所有碱基均为 3’UTR 区,咱们需要的是 FASTA 格式的碱基序列,所以先复制一小段 3’UTR 起始段,如 agtcaac,然后移动到此页上方找到
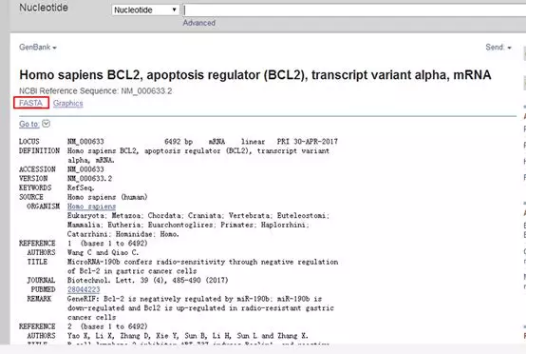
点击 FASTA, 进入下一页,用 CTRL+F,在小方框内输入 agtcaac 如下图
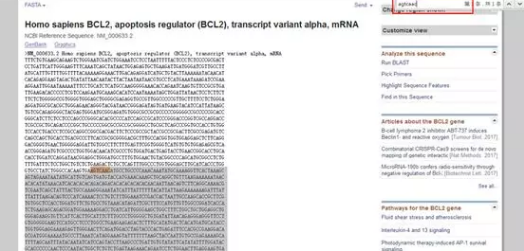
找到 3’UTR 区起始部,将下方的所有碱基全部复制到第二个文本框里头,如下图
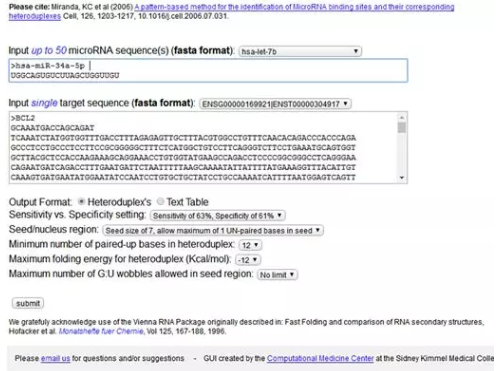
如上图在碱基序列的前方一定要 「> 某基因」,这样这个网站才能识别,接着点击 submit, 进入下一页结果
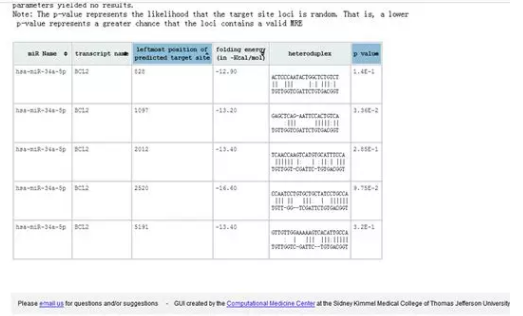
此图即为预测到的结果图。