6 步搞定肿瘤相关基因的功能注释和富集分析
丁香园
DAVID(the Database for Annotation, Visualization and Integrated Discovery),它是一个生物信息数据库,整合了生物学数据和分析工具,为大规模的基因或蛋白列表(成千上百个基因 ID 或者蛋白 ID 列表)提供系统综合的生物功能注释信息,帮助用户从中提取生物学信息(想了解更多,转战度娘~)。
Tips: 通过 DAVID 对肿瘤相关基因做功能注释和富集分析。
<1>
打开 DAVID 平台
http://david.abcc.ncifcrf.gov/
观察 DAVID 提供的功能有:寻找富集的生物主题,尤其是 GO 条目;发现富集的功能相关的基因组;聚合冗余注释条目;可视化基因的 BioCarta 与 KEGG 通路图;2-D 视图展示相关的多基因对多条目关系;搜索其它未在列表中的功能相关基因;列出相互作用蛋白;批量浏览基因名称;连接基因与疾病的关系;高亮蛋白功能结构域和模体;重定向到相关文献;转换基因标识和更多功能。

<2>
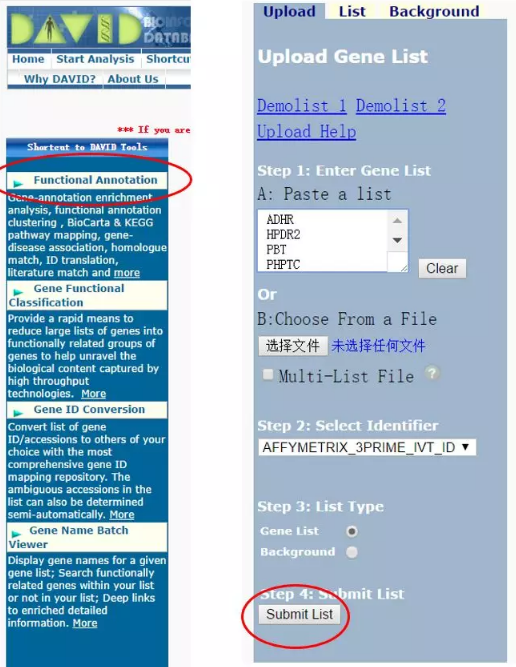
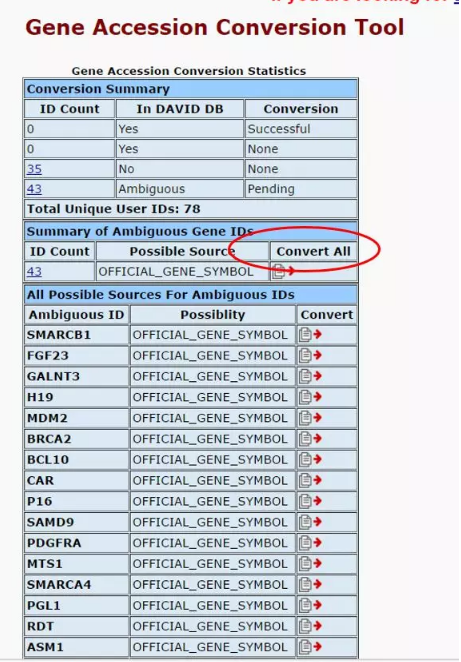
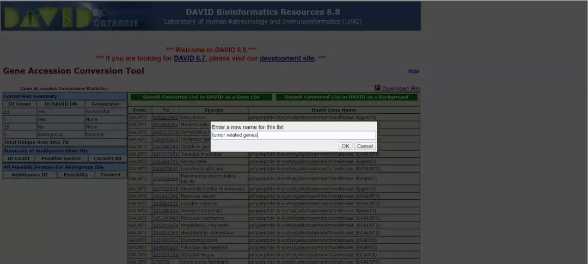
使用左侧 「Functional Annotation Tool」 工具,将 OMIM 数据库处理的所有肿瘤疾病关联的致病基因输入到 Gene List 中,「Selected Identifier」选项先随意选择,list type 选 「gene list」,随后提交;此时会报错说识别符选择错误,1)可以选择观察系统鉴定后的识别符类型,从头开始以该识别符进行提交;2) 也可以选择继续进行,网站建议利用 「Gene Accession Conversion Tool」进行转换,此时进行提交,点击 「convert all」 将基因名称进行转换,随后点击 「submit converted list to DAVID as a gene list」,此时会让你对该 「list」 命名,写成 「tumor related genes」 便于识别。

<3>
在 「Gene ListManager」 中选择物种为 Homo sapiens,「ListManager”」中可以看到提交的基因列表 「tumor related genes」,选择 「use」;在右侧 「Analysis Wizard」 下点击 「Functional AnnotationTool」 即可得到结果。

<4>
在 「AnnotationSummary Results」 结果页面中可以查看对这些基因进行富集分析与功能注释的结果,点击如图部分 「chart」 按钮可展示 GeneOntology 中 Biological Process 结果,即可观察最强的前 5 个是什么,如下图。
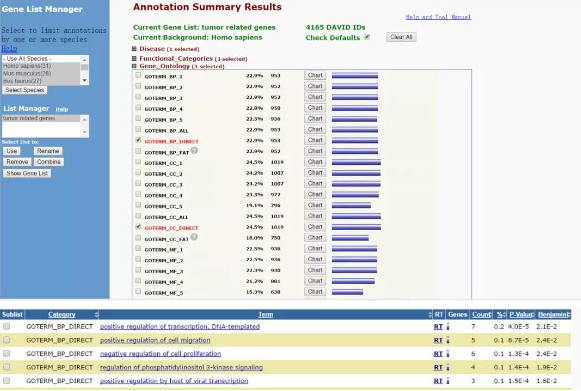
<5>
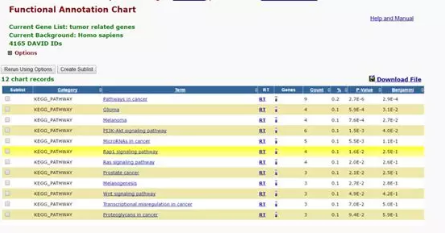
点击如图 「chart」 按钮可展示 KEGG 的相关通路结果,即可观察其做多关联的前 10 个 KEGGpathway 是什么,如下图。

<6>
点击进入 KEGG 通路,在页面右侧有 「Download File」 右键另存即可得到相关 「txt」 文件。将这些文本文件导入 Excel 中,导入时选择 「数据包含标题」,而分隔符用默认的 「tab」,会得到对应数据集。这是观察提交的基因中在该通路中出现的分别是什么。

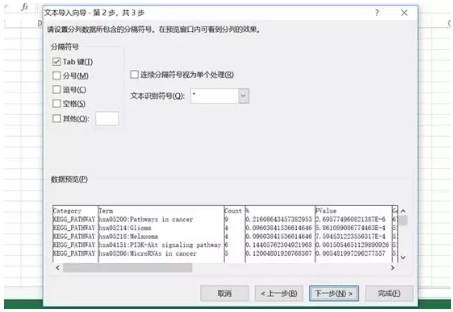
在 KEGG 通路结果中,点击 Pathways in cancer,可以观察提交的基因在通路中出现的位置,在最下面有该通路涉及多少个基因,而提交的基因中有多少个基因在该通路中,分别单击对应按钮,可以得到基因信息列表。

还可以得到通路图如下,是不是很炫酷~
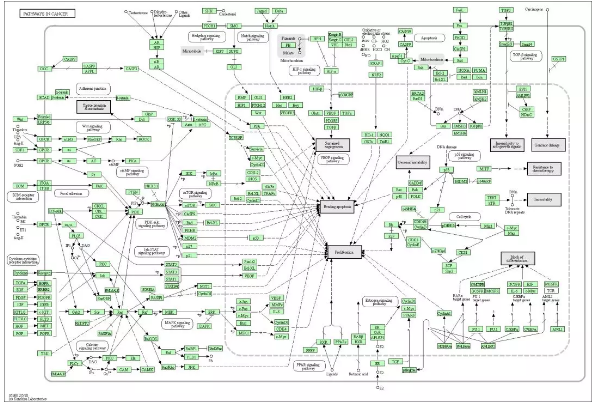
哇咔咔,大概就是酱紫啦~~
作者:笑笑
封面:全景网