如何检测 CRISPR 的脱靶效应
简书-小光amateur
经典的 CRISPR/Cas9 系统具有较多优点,却存在很高的脱靶风险——不仅切割其靶向目标位点,在具有相似序列的位置也可能会发生切割 [1]。
下面给大家介绍一下 CRISPR 脱靶的检测方法。
目前有很多可以设计 gRNA 并预测脱靶位点的网站,除了常用的 elevation 和 CRISPOR 外,还有 synergizing CRISPR 以及 deep CRISPR 等模型。
但是如何从计算机的模拟预测中挑出一个完美 gRNA 呢?
目前是 gRNA 切割后,测序检测其脱靶位点。但是还有预测工具是否可靠,预测范围外是否脱靶,如何高通量检测脱靶位点等很多问题需要解决。所以科学家们正在积极寻找简单高效的检测 CRISPR 脱靶方法。
细胞外检测方法
1、Digenome-seq
Digested genome sequencing,通过核酸酶切割提基因组 DNA,高通量测序,生物信息学分析来检测脱靶情况——显然这种脱离细胞本身的检测方法无法检测真实细胞内发生的突变 [2]。
2、CIRCLE-seq
将提取的 DNA 断裂成 300 bp 后再环化,加入 gRNA 和核酸酶,含有靶标序列的环形 DNA 会被切割成线性 DNA,而没有酶切位点的环形 DNA 则不会。再对线性的 DNA 进行高通量测序,其与 Digenome-seq 相比,是一种更为灵敏的细胞外检测方法 [3]。
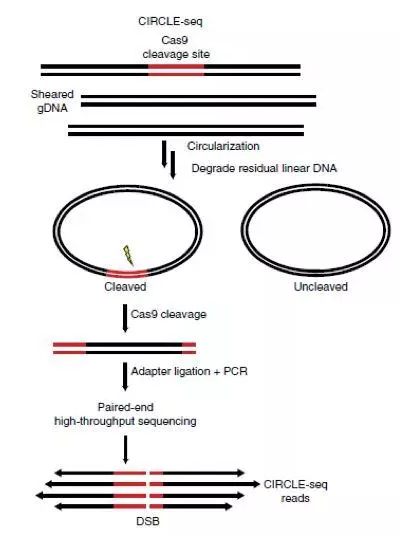
CIRCLE-seq 流程示意图
3、SITE-Seq
在细胞外将 DNA 在 sgRNPs 处理后进行高通量测序,该技术使用了 sgRNA 编程的 Cas9 对基因组 DNA 中的剪切位点序列进行识别 [4]。该方法将生化识别法与细胞活性检测法相结合,具有高精高敏的脱靶位点鉴别能力。
细胞内检测方法
1、GUIDE-seq
在细胞内通过核酸酶切割产生双链断裂后,以非同源性末端接合 (Non-homologous endjoining, NHEJ) 的方法,引入一种短双链寡核苷酸来标记 CRISPR-Cas 诱导的脱靶断裂,高通量测序找到脱靶位置 [5]。该方法能很好的模拟细胞内脱靶现象,且能找到非预测范围内的脱靶位点,是一种较为可靠的方法。
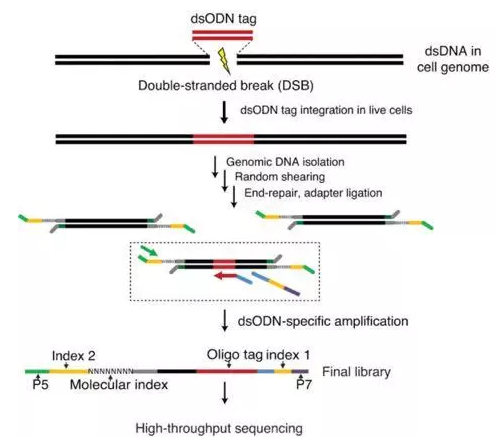
GUIDE-Seq 方法的流程示意图 [5]
2、DISCOVER-Seq
最近加州大学伯克利分校和阿斯利康公司的研究人员开发出一种利用 DNA 修复因子结合染色质免疫共沉淀和高通量测序的方法——DISCOVER-Seq。该方法不仅简化了识别脱靶效应的过程,也提高了检测准确性 [6]。它是改善临床前研究和使 CRISPR 治疗更接近实际患者需要的关键步骤。
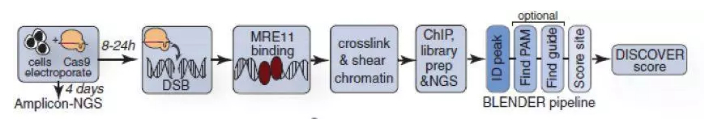
DISCOVER-Seq 工作流程 [6]
3、GOTI
GOTI,Genome-wide Off-target analysis by Two-cell embryo Injection 技术是由中国科学家和斯坦福大学的科学团队共同开发的新型脱靶检测技术。该方法应用双细胞胚胎注射 gRNA 和核酸酶来评估核酸酶的脱靶效应。研究者们在小鼠受精卵分裂到二细胞期时,只编辑其中一个卵裂球,并用红色荧光蛋白标记。在小鼠胚胎发育到 14.5 天时,对小鼠胚胎细胞进行流式细胞分选,分选出基因编辑细胞和未编辑细胞,再测序比较两组差异 [7]。
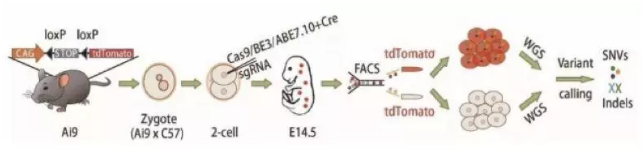
参考文献:
[1] Fu Y, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nature Biotechnology, 2013.
[2] Lin J, et al. Off-target predictions in CRISPR-Cas9 gene editing using deep learning. Bioinformatics. 2018.
[3] Tsai SQ, et al. CIRCLE-seq: a highly sensitive in vitro screen for genome-wide CRISPR–Cas9 nuclease off-targets. Nature Methods, 2017.
[4] Cameron P, et al. SITE-Seq: A Genome-wide Method to Measure Cas9 Cleavage. Nature. 2017.
[5] Tsai S Q, et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. NatureBiotechnology, 2014.
[6] Beeke Wienert, et al. Unbiased detection of CRISPR off-targets in vivo using DISCOVER-Seq, Science, 2019.
[7] Erwei Zuo, et al. Cytosine base editor generates substantial off-target single nucleotide variants in mouse embryos, Science, 2019.