这是项什么神仙技术!Cell 封面文章揭示首个能自动检测癌细胞转移的新策略
通常情况下,转移瘤(metastases)从原发部位侵入淋巴管、血管或其他途经被带到它处继续生长,形成与原发部位肿瘤相同类型的肿瘤。
大多数癌症患者死于由具有原发性或获得性耐药的扩散性肿瘤细胞发展而来的远处转移瘤。
因此,为了更好地了解和治疗癌症转移,开发对扩散性肿瘤细胞和针对肿瘤细胞的靶向药物进行全面且无偏检测的技术显得至关重要。
此类技术将有助于更可靠地探索影响临床前小鼠模型中肿瘤转移和药物靶向的机制,从而为改进治疗方法的发展做出重大贡献。
但是到目前为止,由于缺乏可靠地检测小鼠体内所有单个转移瘤和扩散肿瘤细胞的成像技术,以及快速准确地量化大规模成像数据的算法,使得相关研究一直难以有突破性成果。
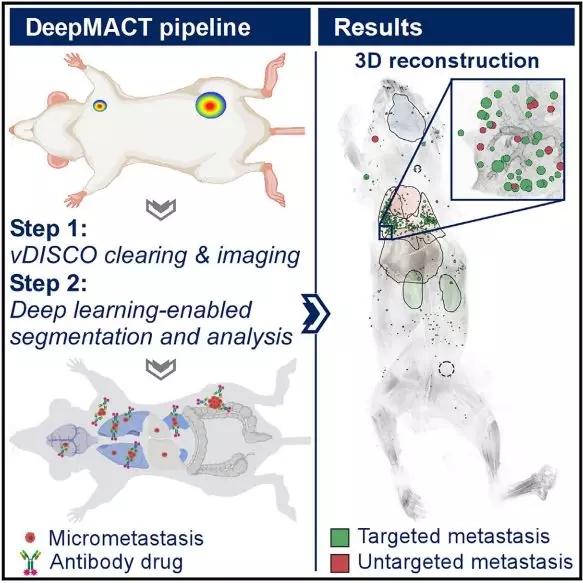
2019 年 12 月 12 日,德国亥姆霍兹慕尼黑研究中心 - 组织工程与再生医学研究所的 Chenchen Pan 等 1 在 Cell 杂志上发表题为《Deep Learning Reveals Cancer Metastasis and Therapeutic Antibody Targeting in the Entire Body》的文章。该研究基于深度学习开发的新型算法 ——DeepMACT,可在全身范围内对多种转移性癌症模型的微转移以及靶向低至单个弥散性肿瘤细胞的治疗性抗体的自动检测和定量进行无偏分析。
图片来源:cell
主要内容
通常,通过荧光蛋白(例如 GFP,YFP 和 mCherry)的内源表达来实现癌细胞在体内外的荧光标记。
但是,由于小鼠体内许多组织在此范围内显示出很高的自发荧光,这妨碍了基于其内源性荧光信号检测小鼠体内的单个癌细胞或小细胞簇。
为了解决这个问题,研究人员使用了之前研发的 vDISCO 技术。
该技术利用与 Atto- 594 或 Atto- 647N 染料偶联的抗 mCherry 或抗 GFP 纳米抗体来增强癌细胞的荧光信号,可在透明的组织中将癌细胞的荧光蛋白信号增强 100 倍以上,使其不仅可以对大范围转移瘤而且还可以对全身的微小转移瘤进行可靠的成像。

图片来源:cell
光学透明小鼠体内肿瘤转移的 vDISCO 成像
研究人员将表达 mCherry 和萤火虫荧光素酶的人 MDA-MB- 231 乳腺癌细胞,移植进 NOD scid gamma (NSG) 小鼠乳腺脂肪垫,并允许肿瘤生长和转移 6 - 10 周。
为了测试 vDISCO 是否可以使小鼠体内的癌症微转移成像。研究人员在应用 DeepMACT 之前先获得了传统的小鼠生物发光图像,与之对比使用偶联于 Atto- 647N 的抗 mcherry 信号增强纳米抗体来应用 vDISCO,并使用辐射荧光对小鼠体进行 2D 成像,使用光片显微镜对整个固定透明小鼠进行 3D 成像,以检测全身的单个微转移。
结果表明,与之前的发现一致,通过生物发光检测了小鼠腋窝淋巴结中最早移植的 MDA-MB- 231 细胞的大转移。但是,生物发光成像未显示出例如大小或形状,并且未能显示出微转移的存在。
而应用 vDISCO 在 2D 成像中,可以很容易地看到原发性肿瘤和腋窝淋巴结的主要转移瘤,3D 成像中可以在肺部和淋巴结中发现各种转移瘤,并检测小鼠全身扫描中单个细胞的微转移。

图片来源:cell
基于深度学习的新型算法检测不同肿瘤模型的微转移
为了在透明小鼠中实现所有癌细胞转移的自动化和快速映射,研究人员基于卷积神经网络 (convolutional neural networks, CNNs) 的高效深度学习方法,并针对 vDISCO 成像数据和转移分布模式进行了优化,以此开发出新型算法 ——DeepMACT。
该算法可以在转移瘤检测中达到与传统检测相同的准确度,但速度却提高了 300 倍以上。并且可在数小时内完成原本需要几个月的传统检测工作,这使得以单细胞分辨率进行高通量癌转移瘤分析成为可能。
利用这一算法,研究人员对小鼠全身进行了分析,结果发现除了原发性肿瘤和腋窝淋巴结的主要转移瘤,还检测到数百种大小不等的微转移,尤其是在肺部。
总体而言,DeepMACT 在这只特定的小鼠中识别出了整个全身 520 个微转移,其中肺中有 306 个微转移,其他器官中有 214 个微转移。
为了验证 DeepMACT 在更大范围环境下的可靠性和适用性,研究人员选用了 3 种不同的肿瘤模型:
裸鼠心内注射人 MCF- 7 雌激素受体 (ER) 阳性乳腺癌细胞;
C57BL/ 6 小鼠移植小鼠同基因 R254 胰腺癌细胞;
裸鼠移植的人脑转移性肺癌细胞系 H2030 -BrM3
结果显示,DeepMACT 能可靠地检测具有不同嗜有机性的多种肿瘤模型中的微转移,包括不同的免疫缺陷或具有免疫能力的小鼠品系,同系肿瘤和异种移植。
此外,可以通过 DeepMACT 对难以通过其他方法实现的器官(例如骨骼和大脑)中的转移进行量化和评估。
重要的是,对于所有测试的癌症模型,无论是在整个时程研究中总体肿瘤负担的增加,还是在不同器官间转移瘤的差异分布都无法通过生物发光图像清楚地揭示出来。
因此 DeepMACT 是第一个能够对全身扫描中的微转移进行定量分析的技术,从而极大地增强了全面评估癌细胞转移过程的能力。

图片来源:cell
检测靶向单一转移水平的治疗性抗体
最后,为了确定治疗性抗体在人体癌细胞单个微转移水平上的分布。研究人员使用 DeepMACT 来评估了针对人碳酸酐酶 XII(CA12)的治疗性单克隆抗体 6A10 的生物分布(CA12 在各类癌症中过表达,抗体 6A10 阻断其活性可降低肿瘤生长,增加肿瘤对化疗的敏感性)。
通过使用 DeepMACT 在单个微转移水平上系统地评估和量化抗体药物靶向在全身扫描中的效率。
虽然抗体总共靶向 77% 的转移瘤,但发现与身体其他部位(66%)相比,针对肺部的微转移明显更多(85%)。
并通过量化抗体信号对比来评估抗体浓度,进一步评估药物靶向肺内微转移相对于身体其他部位的有效性。
以上这些数据表明 DeepMACT 提供了一个强大的平台来跟踪治疗性抗体以及小鼠体内微转移的生物分布。
因此,它代表了第一种方法,可以在全身范围内以低至单个微转移水平的分辨率定量分析基于抗体的药物靶向效率。

图片来源:cell
研究亮点
1. DeepMACT 是基于深度学习的算法,可对癌症转移进行全面分析。
2. DeepMACT 可在全身的 3D 扫描中识别微转移和单个癌细胞。
3. DeepMACT 揭示了全身靶向抗体药物的功效。
4. DeepMACT 表明了药物靶向治疗受到肿瘤微环境的影响。
讨论与分析
在单个癌细胞微转移水平上全面无偏地检测癌症转移和靶向肿瘤治疗药物的生物分布将大大加快临床前癌症研究的速度。
为了实现这一目标,该研究利用了功能强大的组织透明成像方法以及基于深度学习的分析方法,使我们能够可视化和分析透明小鼠体内的癌症转移情况。
由此产生的 DeepMACT 是一种用于全身分析微转移和治疗性抗体药物分布的简单方法,其解析度可在数天内检测到单个微转移,否则将需要数月甚至数年的人工检测。
因此,基于 DeepMACT 对整个全身透明小鼠的评估,而不是对选定的组织 / 器官的评估,可以比传统方法更有效地促进临床前研究新疗法的开发和转化。
扩展阅读
迄今为止,大多数全身组织透明和影像学研究都依赖于内源性荧光信号的可视化,然而该信号强度不足以对透明小鼠体内的转移瘤进行成像和量化。
Rios,Anne C 等 2 开发了一种快速、高通量单细胞分辨率 3D 成像技术(Large-scale Single-cell Resolution 3D imaging, LSR- 3D),联合多颜色谱系示踪技术和 RNA 测序技术,对乳腺肿瘤内细胞可塑性进行研究。

图片来源:Cancer Cell
Liu,Yun 等 3 利用人工智能算法(LYmph Node Assistant, LYNA)评估了载玻片上的每个组织斑块,从而获得了比病理学家更高的肿瘤水平敏感性。

图片来源:Arch Pathol Lab Med
此外,现有纳米抗体与包括近红外范围内的各种光谱的荧光染料的缀合将有助于为多重实验提供更多的选择,包括对一种以上类型的荧光标记细胞以及缀合的治疗性抗体进行成像。
因此,基于深度学习的癌症转移检测方法将逐渐针对各种成像方式(除了显微镜之外)形成可观的发展势头。
参考文献
1. Pan, C., Schoppe, et al. (2019). Deep Learning Reveals Cancer Metastasis and Therapeutic Antibody Targeting in the Entire Body. Cell 179, 1661 - 1676 e1619.
2. Rios, A.C., Capaldo, B.D.,et al. (2019). Intraclonal Plasticity in Mammary Tumors Revealed through Large-Scale Single-Cell Resolution 3D Imaging. Cancer Cell 35, 618 - 632 e616.
3. Liu, Y., Kohlberger, T., Norouzi, M., et al. (2019). Artifificial Intelligence-Based Breast Cancer Nodal Metastasis Detection. Arch. Pathol. Lab. Med. 143, 859–868.