CRISPR 基因编辑利器:Nature 子刊整合 30 种 gRNA 设计工具,强推这 2 种!
2020 年 10 月,Emmanuelle Charpentier 与 Jennifer Doudna 因为在「魔剪」CRISPR 用于基因编辑的原创性工作获得了诺贝尔化学奖。

此时,离她们关于「魔剪」最重磅论文的发表,才仅仅过去 8 年。虽然 CRISPR 因其方便好用著称,如此短的发展历史,意味着这项技术还有极大的提升空间。
在 CRISPR-Cas9 系统中,Cas9 核酸内切酶需要与一段长度约为 100 核苷酸的 Guide RNA(gRNA)的形成复合物,并在 gRNA 与目标序列结合的引导才能进行基因编辑。
要高效、准确地使用「魔剪」进行基因编辑,最首要的任务就是设计一个出色的 gRNA。

2020 年 11 月 5 号,Nature 子刊 Nature Methods 刊登了其编辑 Vivien Marx 所撰写的,题为 Guide RNAs: it’s good to be choosy 的评论文章 [1],重申了 gRNA 选择的重要性,并为 gRNA 的选择,提供了最新的指引。
gRNA 的组成
虽然统称为 gRNA,但在自然界中,细菌的 gRNA 是由 crRNA(CRISPR RNA)与 tracrRNA(trans-activating crRNA)组合而成的。
crRNA 上的间隔区(spacer)负责与名为原间隔序列临近基序(protospacer adjacent motif, PAM,通常为 NGG 序列) 下游的 20 个碱基互补。当 CRISPR-Cas9 定位到 PAM 和互补序列后,Cas9 酶就一举将目标剪短,造成双链断裂(DSB, double-stranded breaks),这细菌利用 CRISPR/Cas9 系统在自然界对抗病毒的原理。
除了 Cas9 之外,其他一些 Cas 酶也被发现具有不同的剪切特性。比如不含 tracrRNA 的 Cas12a 就可以识别除 NGG 之外的多种 PAMs。
病毒的 DNA 断裂后意味着病毒的死亡,但在更高等的生物中,一次小小的断裂并不足以致命。因为很多细胞有着修复 DBS 的能力。在高等动物中,名为非同源末端连接(non-homologous end joining ,NHEJ) 与同源重组 (Homology directed repair , HDR) 的修复能力,能够帮助我们抵御 DNA 损伤所带来的危害。
也正是这两种保护机制,使得张锋等人发现了 CRISPR-Cas 基因编辑的潜力。
在 CRISPR-Cas 系统创造 DBS 后,NHEJ 与 HDR 系统会将中断的 DNA 连接起来,我们也因此可以实现基因的敲除与敲入。根据修复机制的选择情况,科学家们提出了不同的 gRNA 设计策略,以此保证基因编辑的高效性以及准确性。
NHEJ 机制下的 gRNA 选择
NHEJ 的修复机制下的基因编辑像是一把「锤子」。
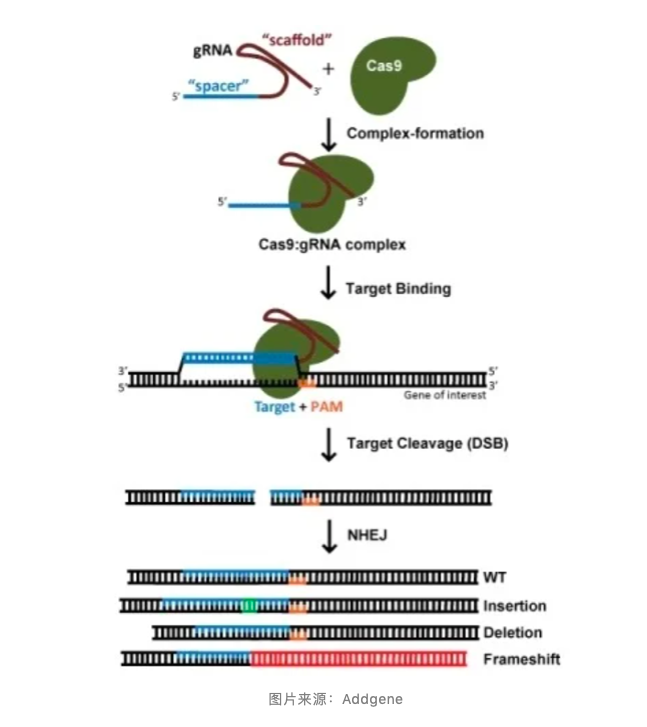
通过剪断 DNA,CRISPR-Cas 系统会在目标序列中创造一个得失位(indel),无论得失位在目标基因的编码区还是非编码区,都足以导致基因序列的移码,并以此导致该基因被失活敲除。
在选择 NHEJ 修复机制基因敲除的 gRNA 时,第一原则是避免目标序列在蛋白的 N 端,以降低细胞利用位于附近的启动子重启基因的可能。
此外,也应该尽量避免在目标蛋白的 C 端选择 gRNA,以免敲除后产生的等位基因依旧具有功能。
在这种情况下,如果我们在目标蛋白的编码序列 5% 到 65% 的区域中进行编辑,由于每 8 个碱基中就会出现,我们依旧会有大量的备选项,所以具体如何选择,Nature Method 也给出了建议,后文中我们会进一步介绍。
HDR 机制下的 gRNA 选择
比起锤子来说,HDR 机制下的基因编辑更像是「拼图」。
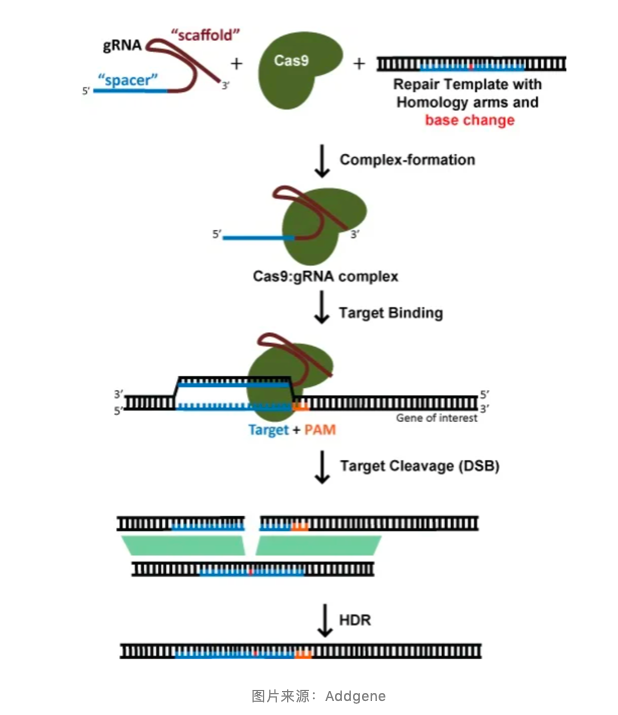
与锤子不同,使用 HDR 进行编辑时,需要同时在系统中引入一定量的同源序列(修复模版)。这段同源序列中应该携带需要替换的碱基,并伴随着同源重组的发生替换、并连接 CRISPR-Cas 系统造成的 DBS。通过将同源序列拼进基因组,达到碱基编辑的目的。
如果编辑位点离修复模版远端的距离大于 30 碱基,HDR 机制的编辑效率将会显著下降,且由于 PAM 的限制,比起 NHEJ 来说,HDR 机制下的 gRNA 的选择范围则窄的多。
gRNA 设计工具
除了编辑效率和脱靶效果的考虑,作为心细的科学家,我们还要考虑这些 gRNA 的制备是否容易,毕竟 RNA 造价不菲。所以在设计过程中还要尽力避免多聚核苷酸序列,诸如多聚胸腺嘧啶(Poly(T))的序列,能有效终止 RNA 的转录,使得 gRNA 难以生产。
为了帮助科学家设计 gRNA,学术界已经开发了超过 30 种基于网页的 gRNA 设计工具。
Nature Method 编辑着重介绍了由来自哈佛大学与 MIT 联合成立的博德研究所学者 John G. Doench 于 2020 年 4 月 13 年发表在 Nature Biotechnology 的综述文章[2],题为 Design and analysis of CRISPR–Cas experiments。
在这篇综述中,John G. Doench 从市面上常见的 gRNA 设计工具总结出了 2 大 gRNA 设计规律组合,并建立了 gRNA 准确性的定量模型。Doench 团队的 gRNA 数据库现在可以在 Addgene 网站上找到。
而来自微软研究院的 Jennifer Listgarten(先任职于加州大学伯克利分校)与 Nicolò Fusi 在 Doench 的帮助下,分别设计了 gRNA 的 on-target 模型 Azimuth 与 off-target 模型 Elevation。
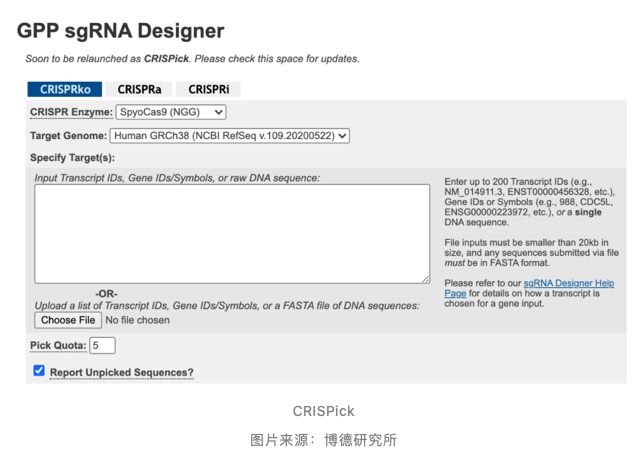
这两个基于人类全编码区的模型现在已经整合为 CRISPick 工具,并在博德研究所的网站中可用。该工具是评估 gRNA 潜在脱靶效应与编辑效率的利器,也可以用于基于 CRISPR 系统的基因抑制与激活的评估,
与此同时,Doench 也建立了一种名为 CFD (cutting frequency determination) 的评分规则,用以评估 gRNA 与特定 DNA 相互作用下的脱靶潜力。这个名为 GUIDES 的工具现在也可以线上使用。
目前 GUIDES 能够提供 spCas9 系统在人与小鼠中的评估,而 CRISPick 能够评估 SpCas9, SaCas9, AsCas12a 与 enCas12a 在人、小鼠与大鼠中的应用。
除此之外,诸如 GPP sgRNA Designer, CHOPCHOP, CRISPOR, inDelphi 等工具,也能用于预测 gRNA 活性以及编辑结果。
除此之外,尽管应用更少,起步更晚。一些用于碱基编辑(Base Editing)与刘如谦教授的引导编辑(Prime Editing)的 gRNA 设计工具也开始涌现。
在类似博德研究所所提供的免费工具之外,一系列实验耗材巨头也先后推出的 gRNA 设计服务。
安捷伦提供了免费 gRNA 设计工具,且能够在合成 gRNA 进行特定的修饰。IDT 也提供了类似的工具,且适用于人以及包括小鼠、大鼠、斑马鱼与线虫在内的常见模式动物。
CRISPR 的出现,彻底改变了很多生物学家做实验的方法。是包括 Doench 在内的各位科学家的无私奉献,让 CRISPR 成为人人触手可及的神器。
如何设计 gRNA,你学会了吗?