zt分子生物学常用软件
丁香园论坛
3584
分子生物学软件
目前,有很多软件可以解决分子生物学研究人员从立项到最后写论文的实际问题。但由于软件的数目太多,而且各个软件开发环境、运行平台和操作方法都各不相同——即使功能的某些方面是相似的,这就给使用者造成了许多的不便。赛百盛公司信息部在对相同作用的各类软件进行比较后,推荐一些最实用软件给从事生物研究的工作人员。
一、实验准备阶段 这时一般要查一些与实验相关的文献,以便对自己所要做的课题的最新的进展有一个基本的了解,从而确定自己的实验策略。推荐使用软件:Reference Manager 9.0 Reference Manager 9.0可以在线通过查找关键词搜索PubMed和609个Z39.50数据库中的专业资料,同时保存查找的资料为本地文件。资料内容和记录分上下两个屏幕,如有全文或想连接网络时敲键盘就可以到相应的全文文章和摘要。可以直接在WORD中查找资料,并插入引用。在文章中对引用的文献可以格式化,引用的参考资料格式有很强的用户自定义功能,可以符合各种杂志对引用格式的要求,引用时不用多窗口切换。
同类软件:
Endnote 3.1.2 也是一个在线专业资料查找系统,可以保存查找资料,并在文章中对引用格式化.
二、实验实施阶段 随着实验的进行,就必须对实验过程中的DNA、RNA和蛋白质的信息进行各种处理,包括限制酶分析、引物设计、同源序列比较、质粒作图、结构域(motif)查找、RNA二级结构预测、蛋白二级结构分析、三维结构显示等方面的内容。
1、综合软件 推荐软件:Omiga 2.0 实际上,大部分对核酸蛋白的序列分析功能,在Omiga 2.0中都能找到;而且界面非常友好。Omiga作为强大的蛋白质、核酸分析软件,它还兼有引物设计的功能。主要功能:编辑、浏览、蛋白质或核酸序列,分析序列组成。用Clustal. W进行同源序列比较,发现同源区。实现了核酸序列与其互补链之间的转化,序列的拷贝、删除、粘贴、置换以及转化为RNA链,以不同的读码框、遗传密码标准翻译成蛋白质序列。查找核酸限制性酶切位点、基元(Motif)及开放阅读框(ORF),设计并评估PCR、测序引物。查找蛋白质解蛋白位点(Proteolytic Sites)、基元、二级结构等。查寻结果可以以图谱及表格的显示,表格设有多种分类显示形式。利用Mange快捷键,用户可以向限制性内切酶、蛋白质或核酸基元、开放阅读框及蛋白位点等数据库中添加或移去某些信息。每一数据库中都设有多种查寻参数,可供选择使用。用户也可以添加、编辑或自定义某些查寻参数。可从MacVectorTM、Wisconsin PackageTM等数据库中输入或输出序列。另外,该软件还提供了一个很有特色的类似于核酸限制酶分析的蛋白分析,对蛋白进行有关的多肽酶处理后产生多肽片段。
同类软件:
DNASIS 2.5 DNASIS for Windows 2.5版是日立软件公司(Hitachi Sofeware Engineering Co.,Ltd.)97年推出的一个功能强大的序列分析软件。包含有大部分分子生物学软件的常用功能,可进行DNA,RNA,蛋白质序列的编辑和分析,甚至还能进行质粒作图、数据库查询等功能,足可满足一般实验室的要求。在DOS时代,DNASIS 7等版本便是流传甚广并曾给过许多人以帮助的分子生物学软件,因此我们有理由期待Win版的DNASIS 会带给我们惊喜。 DNATools 5.1 与Omiga, DNAsis, PCgene等软件属于同一类的综合性软件,操作简单功能多。DNATools设计的用户友好、强壮,以便快速、方便地获取、贮藏和分析序列及数据库查询获得的序列相关信息。
DNATools包容性很好,能把几乎所有文本文件打开作为序列。当程序不能辨别序列的格式时(通过寻找常用序列格式的特征),会显示这个文件的文本形式,以便你编辑生成正确的蛋白质或DNA序列,编辑后可以再被载入程序。若你的序列是DNATools格式时(DNA或寡核苷酸序列),程序不加注解的载入序列,程序模式调整成可以接受载入的数据类型(蛋白质、DNA和寡核苷酸引物序列)。在一个项目中可以加入几千个序列或引物,并在整个项目中分析这些序列及标题。这个程序的一个特点是给每个序列或引物添加文本标题。这样就可以用自定义的标题识别序列,而不必通过它们的文件名。为避免丢失数据-和你的工作-DNATools包括几个挽救丢失数据的功能:一个5层的撤销/重复功能,重新获得原始序列的恢复功能,项目中加入新文件时的安全备份功能,以及在一定的时间间隔作完全备份或备份你的工作。
2、限制酶切位点分析 这是分子生物学研究过程中最基本的操作,虽然可以通过文本编辑软件来查找,但过程烦琐(特别是序列长而分析的酶切位点多时)。通过专门的限制酶分析软件,不但方便,不易出错;而且输出的格式多样,满足用户的各种要求。推荐软件:DNAssist 1.0 能进行限制酶分析的软件很多,而我们建议使用DNAssist 1.0;原因是大多软件只对线性序列进行分析,那么cNNNNN…NNNgaatt环状的序列就找不到EcoR I的位点。DNAssist 1.0能很容易把这个EcoR I位点找出来。另外DNAssist在输出上非常完美,除了图形、线性显示外,还有类似DNASIS的列表方式,列出有的位点(按酶排列,按碱基顺序排列)。
同类软件:Primer Premier 5.0 见下。
DNAClub: DNA Club是一个简单的对DNA进行与PCR有关的操作的软件。它的功能有: 1、输入DNA序列; 2、查找ORF序列; 3、把DNA翻译成蛋白序列; 4、查找酶切位点; 5、查找PCR引物序列。 功能虽然都很简单,但非常实用,一般的用户都可以很方便地使用。
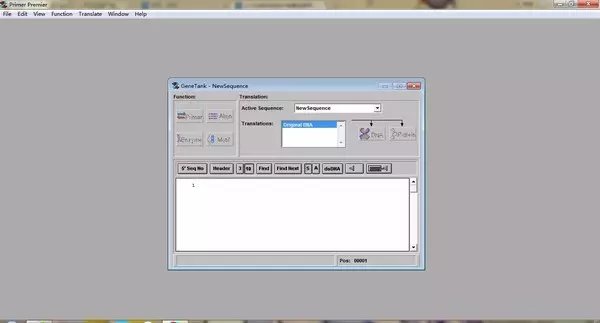
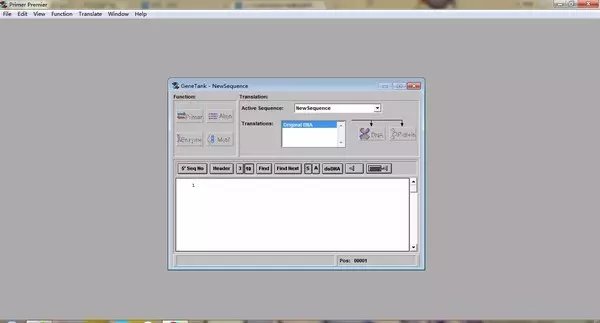
3、引物设计 优秀的引物设计软件能从模板序列中按用户的要求挑选出一系列引物序列,同时把这些序列的所有特性(包括分子量、Tm值、二级结构、上下有引物间的错配、3’端的稳定性及GC含量等等)分析出来。当然,这里的引物包括PCR引物、测序引物和探针。推荐软件:Primer Premier 5.0 Primer Premier5.0是由加拿大的Premier公司开发的专业用于PCR或测序引物以及杂交探针的设计和评估的软件,和Plasmid Premier2.02一起是该公司推出的最新的软件产品。其主要界面同样也是分为序列编辑窗口(Genetank),引物设计窗口(Primer Design),酶切分析窗口(Restriction Sites)和Motif分析窗口。这里我们主要介绍其引物设计功能,顾名思义,该软件就是用来进行引物设计的。可以简单地通过手动拖动鼠标以扩增出相应片段所需的引物,而在手动的任何时候,下面显示各种参数的改变和可能的二聚体、异二聚体、发夹结构等。也可以给定条件,让软件自动搜索引物,并将引物分析结果显示出来。而且进行这些操作非常简单。其功能绝不在Oligo 5.0之下!
同类软件:DNAClub 见上。
4、序列的同源比较(Alignment)具有这一功能的软件或软件包很多,但功能全面,界面友好,同时输出结果美观实用的不多。推荐软件: GeneDoc 3.2 GeneDoc能用用亮丽的色彩来区分相互间序列的同源性,输出的格式一目了然,而且可以报告为进化树的格式。选择项多,可以达到所需的要求。功能多又强,但要完全掌握,并不是很轻松的事。
同类软件:
MACAW 2.05多序列构建与分析工作台软件(Multiple Alignment Construction & Analysis Workbench, MACAW)是一个用来构建与分析多序列片段的交互式软件。MACAW具有几个特点:1. 新的搜索算法查寻类似区,消除了先前技术的许多限制。2. 应用一个最近发展的数学原理计算block类似性的统计学显著性。3. 使用各种视图工具,可以评估一个候选block包含在一个多序列中的可能性。4. 可以很容易地编辑每一个block。在多序列中查找一个类似片段并不是一件简单的事,主要是因为要查找的量极大。这正式MACAW所要解决的问题。 Clustal X 用来对核酸与蛋白序列进行多序列比较(multiple sequence alignment)的软件。多序列比较在分子生物学中是一个基本方法,用来发现特征序列,进行蛋白分类,证明序列间的同源性,帮助预测新序列二级结构与三级结构,确定PCR引物,以及在分子 进化分析方面均有很大帮助,Clustal X很适合这些方面的要求。
5、质粒绘图 就象限制酶切位点分析一样,也是最常用的功能。好的质粒绘图软件首先能对对已知序列自动作图;如果是未知序列,但根据用户已知的质粒信息也能绘出漂亮的图谱来。推荐软件:Gene Construction Kit 2.0 Gene Construction Kit 2.0这一个非常好的质粒构建软件包。与大多数分析的软件不同,它制作并显示克隆策略中的分子构建过程;包括质粒构建,模拟电泳条带;当然还可以质粒作图(有无序列均可)。通过它绘出来的图还可以继续用来构建克隆策略图谱。只是该软件由于功能太强,使用起来也不简单;幸亏它附有详细的使用帮助,认真研究后,应该没有问题。 同类软件:Winplas 2.6该软件用来绘制发表质量的质粒图,可广泛应用与论文、教材的质粒插图。其特性包括:1.知道序列或不知序列结构均能绘制质粒图;2. 可读入各种流行序列格式文件引入序列信息;3. 自动识别限制位点 可构建序列结构,功能包括:从文件插入序列、置换序列、序列编辑、部分序列删除等;4. 绘图功能强大,功能包括:位点标签说明、任意位置文字插入、生成彩图、线性或环形序列绘制、可输出到剪贴板、可输出到图像文件;5. 限制酶消化分析报告输出与序列输入报告功能。 Plasmid Premier2.02是由加拿大的Premier Biosoft公司推出的用于质粒作图的专业软件,主要用于进行质粒作图,质粒特征分析和质粒设计。其主要界面分为序列编辑窗口(Genetank),质粒作图窗口(Plasmid Design),酶切分析窗口(Restriction Sites)和纹基分析窗口(Motif)。打开程序就可进入序列编辑窗口,可以直接打开Genbank或Vector数据库中已知质粒的序列文件,将序列读入,并将有关于质粒的各种特征,包括编码区,启动子,多克隆位点以及参考文献等信息保存在Header中;也可以直接输入序列进行未知质粒的设计。
6、结构域(motif)查找。 推荐软件:Primer Premier 5.0 Primer Premier 5.0的结构域查找功能与它的引物设计一样强,结果能以图形、表格、序列三种方式输出。同时还提供了一些未知的结构域的列表;当然软件本身也提供了大量的已知结构域的序列。
7、RNA二级结构预测 二级结构分析和预测的软件很多,但绝大部分都只能在Mac和Unix下使用,在Windows平台下的很少。 推荐软件:RNAdraw 1.1b2 RNAdraw是一个进行RNA二级结构计算的软件。 1. 它 是Windows下的多文档窗口 (multiple document interface) 软件,允许你同时打开多个数据处理窗口。主窗口的工具条提供一些基本功能:打开文件、导入文件、关闭文件、设置程序参数、重排窗口、以及即时帮助和退出程序。2. RNAdraw中一个非常非常重要的特征是鼠标右键菜单打开的菜单显示对鼠标当前所指向的对象/窗口可以使用的功能列表。 3. RNA文库(RNA Library)用一种容易操作的方式来组织你所有的RNA数据文件。 同类软件:RNAStructure 3.5 RNA Structure 根据最小自由能原理,将Zuker的根据RNA一级序列预测RNA二级结构的算法在软件上实现。预测所用的热力学数据是最近由Turner实验室获得。提供了一些模块以扩展Zuker算法的能力,使之为一个界面友好的RNA折叠程序。
8、蛋白二级结构分析 推荐软件:Antheprot 4.5 蛋白序列分析软件包ANTHEPROT 4.5是位于法国的蛋白质生物与化学研究院(Institute of Biology and Chemistry of Proteins)用十多年时间开发出的蛋白质研究软件包。软件包包括了蛋白质研究领域所包括的大多数内容,功能非常强大。应用此软件包,使用个人电脑,便能进行各种蛋白序列分析与特性预测。更重要的是该软件能够提供蛋白序列的一些二级结构信息,使用户有可能模拟出未知蛋白的高级结构。
9、三维结构显示 通过各种方法计算出来的各种三维结构的数据,只要在三维结构浏览软件的帮助下,才能显示出来;从而使用户形象地看到生物大分子的三维立体结构。推荐软件:RasMol 2.7.1 RasMol 2.7是计算化学与分子图形学以及信息产业的同步高速发展的成果,使一个普通的科研工作人员,在自己的个人电脑上,就可以从Internet上的各种免费数据库中,下载所需观察与研究的分子坐标文件,进而通过RasMol 2.7以各种模式、各种角度,甚至按照自己的意愿旋转着,观察此分子神秘的微观三维立体结构,进而了解化合物分子结构和各种微观性质与宏观性质之间的定量关系。RasMol 2.7的作者是Glaxo&Wellcome公司(世界第一大制药公司)研发中心的科学家Roger Sayle。 RasMol 2.7最大的特点是界面简单,基本操作简单,运行非常迅速,对机器的要求较低,对小的有机分子与大分子,如蛋白质、DNA或RNA, 均能适用,且显示模式非常丰富。而且它程序短小,功能强大。尚无在功能上出其右者。其显示窗口可以很简单通过选择相应的菜单来显示分子的三维结构。用命令行窗口,通过输入命令可以执行和显示复杂和要求极高的三维图形。 同类软件:Cn3D Cn3D是由NCBI开发的用于观看蛋白质三维结构的软件,其设计的主要目的是为NCBI在其站点中的蛋白质结构数据库MMDB提供专业的结构观察软件,其主要的操作界面分为两个窗口,如右图所示,一个为结构窗口,另一个为序列窗口。与其他的类似的软件,如Rasmol,Weblabview等相比,其在结构观察方面主要功能上基本相似,但是图形格式上比Rasmol和Weblabview要差一些。而在与网络连接上,该软件能依托NCBI所建立的所建立的MMDB结构数据库,能直接根据输入的序列号从数据库中利用其内嵌的Entrez搜索引擎调出蛋白结构来进行观察,比其他软件要简便。而Cn3D主要的特点是能够将两个蛋白放在一起直观地进行三维结构上的比较,如下图中是将两种核酸外切酶的三维结构通过VAST对准得到的结构比较图:同样,Cn3D在结构比较方面也能利用其内嵌的Blast搜索引擎直接访问Genbank数据库找到具有局部相似性的结构数据,并在三维结构图中显示出二者具有相似性的结构区域。
10、格式转换 各种软件为了自己软件的需要有不同的格式,对我们使用数据带来了困难;格式转换软件能帮助用户解决这一问题。推荐软件:Seqverter 1.3 Seqverter 1.3使用简单,填写输入文件和输出文件名就可以完成格式转换。而且能够转换的格式非常之多是其它软件所无法比拟的。另外,它可在线升级以读写更多格式文件。 同类软件:Visual Sequence Editor 1.1 1.可以同时显示编辑多达200个核酸蛋白序列 2.将所有程序打包成一个库文件 3.可以输入输出到很多格式序列: IG/Stanford;GenBank/GB;NBRF;EMBL;GCG;DNA Strider;Fitch;Pieron/FASTA;PIR/CODATA以及普通文本 4.可以直接读取MACAW的文件,并能将各序列中的同源顺序标出 5.根据遗传密码(可以自定义)读出一种或六种阅读框 6.查看的字体大小,字母间隔等灵活可调 7.模糊查找特定序列。
11、电泳图谱分析 推荐软件:band leader 3.0 提供处理DNA或蛋白分子凝胶电泳图象和从凝胶电泳图象获得相关数据的工具。它可以对电泳图谱进行半定量分析,识别扫描得到的WINDOWS图象格式 .BMP,是一个难得的好软件。
三、 实验结果输出阶段
1、生成出版质量的图片 论文发表时,一般分子结构图的质量要求较高,如果直接从RasMol等软件中抓取图片,往往都不够精美。 推荐软件:Pov-Ray for Windows 3.1 Pov-Ray for Windows 3.1 相当于一种语言,它可以修改pov格式文件,用户还可以自己设定光源、摄像机、材质、阴影等因素,把生物分子的表面用材质填充;并根据光源、摄像机得到生物大分子的三维图。分辨率最大可达128undefined1024。
同类软件:molmol 2.5.1该软件能将pdb等格式的蛋白文件通过转换,存成普通的图形文件。
2、向数据库递交序列 推荐软件:Sequin 2.90 Sequin 2.90能向GenBank、EMBL、DDBJ三大核酸序列库递交序列。可以不用仔细考虑各数据库文件的具体格式,以问答填表格的形式,将需递交的序列和相关资料填好。可以在线直接发送,也可以生成相应格式文件,以e-mail形式发送。
Annhyb AnnHyb2.22是设计PCR引物和DNA探针的辅助软件。主要功能有: 1.对寡核苷酸序列,计算溶解温度(nearest-neighbour algorithm,可以设定盐浓度和引物浓度), 序列长度、GC百分比,分子量, 摩尔消光系数,IUB/IUPAC序列、反向序列、互补序列。 2.对50个bp以上的序列, 计算长度,GC百分比、不同盐浓度下的溶解温度 3.对长序列可以进行反向互补序列。 4.可以读出序列
Cervus CERVUS 是一个根据共显性资料来推测家系似然性的WINDOWS95程序,它可以用来计算等位基因发生概率,运行模拟系统来判定似然比率的临界值和分析动植物种群中的家系。这种模拟系统还可能应用于评估家系推测中所使用的系列单位点标记物的分辨能力。
Clonemap Clonemap是由CGC公司设计的用于辅助克隆设计的质粒图谱绘制软件。该软件除了能实现质粒图谱的绘制功能以外,还有其他一些辅助功能,如对序列进行翻转,按六个阅读框进行蛋白翻译,寻找开读框,搜索特定序列。 Dicroprot DICROPROT software for Windows Release 2.0 beta 是法国蛋白生化研究所蛋白构象研究室Gilbert Deleage博士编制的用于观察蛋白质圆二色谱的软件,是一个较为小巧、相对较为专业、操作简单的研究辅助工具。
Digest DIGEST 能够扫描DNA序列并查找限制性内切酶位点.它支持使用者限定搜索特定的酶,如果此酶在限制性酶切酶库文件WISCONSI.920中,它会列出酶切位置并对酶切片段按长度排序.它使用Don Gilbert的读序模块UREADSEQ,所以读出大部分的序列文件格式。
DNAMend DNAmend是一名德国人编写的用于对DNA序列进行分析,编辑的专业软件,主要的用途是在基因克隆过程中,利用其提供的对DNA序列的限制性酶切分析,开读框搜索,序列转译,末端修剪等功能对DNA序列进行酶切、连接、末端补平等模拟操作,为克隆流程设计及克隆过程监视提供直观的控制工具,便于对克隆中可能出现的问题进行分析,并可将结果输出用于发表文章。
ISIS DRAW 非常非常棒的化学结构式绘制软件,能绘制各种复杂的化学分子(包括有机大分子)的平面及立体结构,还能绘制复杂的化学反应方程式,是写论文和考试试卷的常备工具。强烈推荐给有关生物化学研究及教学的工作人员。 MatInd 使用MatInd对一组DNA序列或一个核酸分布矩阵进行分析后,能够产生描述转录因子结合位点的矩阵型来。这些矩阵型又可以被MatInspector用来扫描核甘酸序列并找到与之匹配的结合位点。
MatInspector 利用MatInd所生成的矩阵型对核甘酸序列进行扫描并使之与结合位点匹配。在输出文件中包括匹配位置的核心序列、矩阵相似性、具体位置及序列等。
Molgen OLGEN软件包可以根据分子式计算并估计其所有的异构体,MOLGEN为科学和教育在阐明分子的结构组成方面提供了有效方便而廉价的分析工具。MOLGEN结合MOLED和MOLVIEW两个软件,通过描述目的分子包含的亚结构的限制,减少异构体的数量,以便更快的获得目的分子的空间的结构的具体构像;通过MOLVIEW可以计算分子的最稳定合理的立体的结构,并且通过旋转可以从个个角度观察分子的结构情况,和每一个原子的空间的位置。
Mvsp MVSP是一个多变量统计分析软件包。它能进行多种等级特征分析:1,主成分分析(PCA), 2, 主坐标分析(PCO), 对应/对应分离趋势分析(CA/DCA)和典型对应分析(CCA)等。 另外,它还可以通过使用20种不同的距离测量或相似测量及7种分族策略进行族分析。分析得到的多样性指数就可以用于生态学数据的计算,这些指数包括辛普森指数,香农信息指数和Brillouin指数。目前这一版本是一个评估版(限时30天,限次15次),如果你想继续使用就必须付钱买正式版,价格为$140。
PCMolecule PCMolecule 是观察三维分子结构的软件,能够观察以.mcm格式保存的三维分子。
PCrare PC-Rare是应用八聚体频率出现的不一致性的方法(OFD)来设计PCR的引物,此程序简单而有效。
Promsed ProMSED是一个在Windows 3.11/95下运行的简单易用的程序,通过它能够自动或手动进行蛋白质多序列的比较和编辑。该程序能自动识别以下文件格式:NBRF/PIR、Pearson(Fasta)、EMBL/SwissProt、CLUSTAL和GCG/MSF等,同时它的界面及主要功能与通用的文本编辑语言相似。其中自动比较只对Clustal V 运算法则有效,当使用群体操作或用不同颜色表示不同性质及功能氨基酸时,人工比较和序列可视分析就显得更方便一些。随着目前计算机内存的日益扩大,程序中使用序列的大小及个数都没有什么限制了。更可贵的是ProMSED能够比较全部序列、全部序列中的任何子集和序列中选定的某一区域,并且提供一些用来进行序列分析、可视编辑及以图像展示结果的工具。
Redsoft Plasmid Redasoft Plasmid 的设计是用来帮助生命科学家快速而轻松地绘制专业级质量的质粒载体图,Redasoft Plasmid 主要的性能包括:快速和轻松地生成一个清晰,色彩鲜明的环行或线性的载体图。自动识别和解析序列文件。新增的web浏览功能允许你链接到数据库站点,并支持下载和自动将序列文件转换成载体图。强大的限制性内切酶分析功能和拥有将近1000种来自REBASE的限制性内切酶。片段的删除和插入完全模拟克隆实验并和其他图形兼容。所看即所得 (What You See Is What You Get)的编辑环境使得你的打印结果和你在屏幕上看到的保持一致。自动标记各种区段的碱基位置以避免重叠。可选择的图形比例尺使几个构造图间很容易比较。允许质粒图拷贝到剪贴板并粘贴到其他Windows应用程序。可以用e-mail的方式传递载体图。
Simplot本软件与Phylip95协同应用, 它基于Don Gilbert's ReadSeq的编码的形式能够识别多种序列的格式。首先,将序列对齐,将它们以标准的格式保存,如FASTA/Pearson的格式。SimPlot同时可做26组的序列或26个单独的序列,当然,可以只选择感兴趣的序列。 TreeView Tree View是用来生成与打印进化树的软件,它可以读取NEXUS与PHYLIP生成的进化树格式文件,生成进化树,并输出到打印机。
10、格式转换 各种软件为了自己软件的需要有不同的格式,对我们使用数据带来了困难;格式转换软件能帮助用户解决这一问题。推荐软件:Seqverter 1.3
Seqverter 1.3使用简单,填写输入文件和输出文件名就可以完成格式转换。而且能够转换的格式非常之多是其它软件所无法比拟的。另外,它可在线升级以读写更多格式文件。
同类软件:Visual Sequence Editor 1.1 1.可以同时显示编辑多达200个核酸蛋白序列 2.将所有程序打包成一个库文件 3.可以输入输出到很多格式序列: IG/Stanford;GenBank/GB;NBRF;EMBL;GCG;DNA Strider;Fitch;Pieron/FASTA;PIR/CODATA以及普通文本 4.可以直接读取MACAW的文件,并能将各序列中的同源顺序标出 5.根据遗传密码(可以自定义)读出一种或六种阅读框 6.查看的字体大小,字母间隔等灵活可调 7.模糊查找特定序列。
11、电泳图谱分析 推荐软件:band leader 3.0
提供处理DNA或蛋白分子凝胶电泳图象和从凝胶电泳图象获得相关数据的工具。它可以对电泳图谱进行半定量分析,识别扫描得到的WINDOWS图象格式 .BMP,是一个难得的好软件。
三、 实验结果输出阶段
1、生成出版质量的图片 论文发表时,一般分子结构图的质量要求较高,如果直接从RasMol等软件中抓取图片,往往都不够精美。 推荐软件:Pov-Ray for Windows 3.1
Pov-Ray for Windows 3.1 相当于一种语言,它可以修改pov格式文件,用户还可以自己设定光源、摄像机、材质、阴影等因素,把生物分子的表面用材质填充;并根据光源、摄像机得到生物大分子的三维图。分辨率最大可达128undefined1024。
同类软件:molmol 2.5.1该软件能将pdb等格式的蛋白文件通过转换,存成普通的图形文件。
2、向数据库递交序列 推荐软件:Sequin 2.90
Sequin 2.90能向GenBank、EMBL、DDBJ三大核酸序列库递交序列。可以不用仔细考虑各数据库文件的具体格式,以问答填表格的形式,将需递交的序列和相关资料填好。可以在线直接发送,也可以生成相应格式文件,以e-mail形式发送。
Annhyb AnnHyb2.22是设计PCR引物和DNA探针的辅助软件。主要功能有: 1.对寡核苷酸序列,计算溶解温度(nearest-neighbour algorithm,可以设定盐浓度和引物浓度), 序列长度、GC百分比,分子量, 摩尔消光系数,IUB/IUPAC序列、反向序列、互补序列。 2.对50个bp以上的序列, 计算长度,GC百分比、不同盐浓度下的溶解温度 3.对长序列可以进行反向互补序列。 4.可以读出序列
Cervus CERVUS 是一个根据共显性资料来推测家系似然性的WINDOWS95程序,它可以用来计算等位基因发生概率,运行模拟系统来判定似然比率的临界值和分析动植物种群中的家系。这种模拟系统还可能应用于评估家系推测中所使用的系列单位点标记物的分辨能力。
Clonemap Clonemap是由CGC公司设计的用于辅助克隆设计的质粒图谱绘制软件。该软件除了能实现质粒图谱的绘制功能以外,还有其他一些辅助功能,如对序列进行翻转,按六个阅读框进行蛋白翻译,寻找开读框,搜索特定序列。
Dicroprot DICROPROT software for Windows Release 2.0 beta 是法国蛋白生化研究所蛋白构象研究室Gilbert Deleage博士编制的用于观察蛋白质圆二色谱的软件,是一个较为小巧、相对较为专业、操作简单的研究辅助工具。
Digest DIGEST 能够扫描DNA序列并查找限制性内切酶位点.它支持使用者限定搜索特定的酶,如果此酶在限制性酶切酶库文件WISCONSI.920中,它会列出酶切位置并对酶切片段按长度排序.它使用Don Gilbert的读序模块UREADSEQ,所以读出大部分的序列文件格式。
DNAMend DNAmend是一名德国人编写的用于对DNA序列进行分析,编辑的专业软件,主要的用途是在基因克隆过程中,利用其提供的对DNA序列的限制性酶切分析,开读框搜索,序列转译,末端修剪等功能对DNA序列进行酶切、连接、末端补平等模拟操作,为克隆流程设计及克隆过程监视提供直观的控制工具,便于对克隆中可能出现的问题进行分析,并可将结果输出用于发表文章。
ISIS DRAW 非常非常棒的化学结构式绘制软件,能绘制各种复杂的化学分子(包括有机大分子)的平面及立体结构,还能绘制复杂的化学反应方程式,是写论文和考试试卷的常备工具。强烈推荐给有关生物化学研究及教学的工作人员。
MatInd 使用MatInd对一组DNA序列或一个核酸分布矩阵进行分析后,能够产生描述转录因子结合位点的矩阵型来。这些矩阵型又可以被MatInspector用来扫描核甘酸序列并找到与之匹配的结合位点。MatInspector 利用MatInd所生成的矩阵型对核甘酸序列进行扫描并使之与结合位点匹配。在输出文件中包括匹配位置的核心序列、矩阵相似性、具体位置及序列等。
Molgen OLGEN软件包可以根据分子式计算并估计其所有的异构体,MOLGEN为科学和教育在阐明分子的结构组成方面提供了有效方便而廉价的分析工具。MOLGEN结合MOLED和MOLVIEW两个软件,通过描述目的分子包含的亚结构的限制,减少异构体的数量,以便更快的获得目的分子的空间的结构的具体构像;通过MOLVIEW可以计算分子的最稳定合理的立体的结构,并且通过旋转可以从个个角度观察分子的结构情况,和每一个原子的空间的位置。
Mvsp MVSP是一个多变量统计分析软件包。它能进行多种等级特征分析:1,主成分分析(PCA), 2, 主坐标分析(PCO), 对应/对应分离趋势分析(CA/DCA)和典型对应分析(CCA)等。 另外,它还可以通过使用20种不同的距离测量或相似测量及7种分族策略进行族分析。分析得到的多样性指数就可以用于生态学数据的计算,这些指数包括辛普森指数,香农信息指数和Brillouin指数。目前这一版本是一个评估版(限时30天,限次15次),如果你想继续使用就必须付钱买正式版,价格为$140。
PCMolecule PCMolecule 是观察三维分子结构的软件,能够观察以.mcm格式保存的三维分子。
PCrare PC-Rare是应用八聚体频率出现的不一致性的方法(OFD)来设计PCR的引物,此程序简单而有效。
Promsed ProMSED是一个在Windows 3.11/95下运行的简单易用的程序,通过它能够自动或手动进行蛋白质多序列的比较和编辑。该程序能自动识别以下文件格式:NBRF/PIR、Pearson(Fasta)、EMBL/SwissProt、CLUSTAL和GCG/MSF等,同时它的界面及主要功能与通用的文本编辑语言相似。其中自动比较只对Clustal V 运算法则有效,当使用群体操作或用不同颜色表示不同性质及功能氨基酸时,人工比较和序列可视分析就显得更方便一些。随着目前计算机内存的日益扩大,程序中使用序列的大小及个数都没有什么限制了。更可贵的是ProMSED能够比较全部序列、全部序列中的任何子集和序列中选定的某一区域,并且提供一些用来进行序列分析、可视编辑及以图像展示结果的工具。
Redsoft Plasmid Redasoft Plasmid 的设计是用来帮助生命科学家快速而轻松地绘制专业级质量的质粒载体图,Redasoft Plasmid 主要的性能包括:快速和轻松地生成一个清晰,色彩鲜明的环行或线性的载体图。自动识别和解析序列文件。新增的web浏览功能允许你链接到数据库站点,并支持下载和自动将序列文件转换成载体图。强大的限制性内切酶分析功能和拥有将近1000种来自REBASE的限制性内切酶。片段的删除和插入完全模拟克隆实验并和其他图形兼容。所看即所得 (What You See Is What You Get)的编辑环境使得你的打印结果和你在屏幕上看到的保持一致。自动标记各种区段的碱基位置以避免重叠。可选择的图形比例尺使几个构造图间很容易比较。允许质粒图拷贝到剪贴板并粘贴到其他Windows应用程序。可以用e-mail的方式传递载体图。
Simplot本软件与Phylip95协同应用, 它基于Don Gilbert's ReadSeq的编码的形式能够识别多种序列的格式。首先,将序列对齐,将它们以标准的格式保存,如FASTA/Pearson的格式。SimPlot同时可做26组的序列或26个单独的序列,当然,可以只选择感兴趣的序列。
TreeView Tree View是用来生成与打印进化树的软件,它可以读取NEXUS与PHYLIP生成的进化树格式文件,生成进化树,并输出到打印机。
目前,有很多软件可以解决分子生物学研究人员从立项到最后写论文的实际问题。但由于软件的数目太多,而且各个软件开发环境、运行平台和操作方法都各不相同——即使功能的某些方面是相似的,这就给使用者造成了许多的不便。赛百盛公司信息部在对相同作用的各类软件进行比较后,推荐一些最实用软件给从事生物研究的工作人员。
一、实验准备阶段 这时一般要查一些与实验相关的文献,以便对自己所要做的课题的最新的进展有一个基本的了解,从而确定自己的实验策略。推荐使用软件:Reference Manager 9.0 Reference Manager 9.0可以在线通过查找关键词搜索PubMed和609个Z39.50数据库中的专业资料,同时保存查找的资料为本地文件。资料内容和记录分上下两个屏幕,如有全文或想连接网络时敲键盘就可以到相应的全文文章和摘要。可以直接在WORD中查找资料,并插入引用。在文章中对引用的文献可以格式化,引用的参考资料格式有很强的用户自定义功能,可以符合各种杂志对引用格式的要求,引用时不用多窗口切换。
同类软件:
Endnote 3.1.2 也是一个在线专业资料查找系统,可以保存查找资料,并在文章中对引用格式化.
二、实验实施阶段 随着实验的进行,就必须对实验过程中的DNA、RNA和蛋白质的信息进行各种处理,包括限制酶分析、引物设计、同源序列比较、质粒作图、结构域(motif)查找、RNA二级结构预测、蛋白二级结构分析、三维结构显示等方面的内容。
1、综合软件 推荐软件:Omiga 2.0 实际上,大部分对核酸蛋白的序列分析功能,在Omiga 2.0中都能找到;而且界面非常友好。Omiga作为强大的蛋白质、核酸分析软件,它还兼有引物设计的功能。主要功能:编辑、浏览、蛋白质或核酸序列,分析序列组成。用Clustal. W进行同源序列比较,发现同源区。实现了核酸序列与其互补链之间的转化,序列的拷贝、删除、粘贴、置换以及转化为RNA链,以不同的读码框、遗传密码标准翻译成蛋白质序列。查找核酸限制性酶切位点、基元(Motif)及开放阅读框(ORF),设计并评估PCR、测序引物。查找蛋白质解蛋白位点(Proteolytic Sites)、基元、二级结构等。查寻结果可以以图谱及表格的显示,表格设有多种分类显示形式。利用Mange快捷键,用户可以向限制性内切酶、蛋白质或核酸基元、开放阅读框及蛋白位点等数据库中添加或移去某些信息。每一数据库中都设有多种查寻参数,可供选择使用。用户也可以添加、编辑或自定义某些查寻参数。可从MacVectorTM、Wisconsin PackageTM等数据库中输入或输出序列。另外,该软件还提供了一个很有特色的类似于核酸限制酶分析的蛋白分析,对蛋白进行有关的多肽酶处理后产生多肽片段。
同类软件:
DNASIS 2.5 DNASIS for Windows 2.5版是日立软件公司(Hitachi Sofeware Engineering Co.,Ltd.)97年推出的一个功能强大的序列分析软件。包含有大部分分子生物学软件的常用功能,可进行DNA,RNA,蛋白质序列的编辑和分析,甚至还能进行质粒作图、数据库查询等功能,足可满足一般实验室的要求。在DOS时代,DNASIS 7等版本便是流传甚广并曾给过许多人以帮助的分子生物学软件,因此我们有理由期待Win版的DNASIS 会带给我们惊喜。 DNATools 5.1 与Omiga, DNAsis, PCgene等软件属于同一类的综合性软件,操作简单功能多。DNATools设计的用户友好、强壮,以便快速、方便地获取、贮藏和分析序列及数据库查询获得的序列相关信息。
DNATools包容性很好,能把几乎所有文本文件打开作为序列。当程序不能辨别序列的格式时(通过寻找常用序列格式的特征),会显示这个文件的文本形式,以便你编辑生成正确的蛋白质或DNA序列,编辑后可以再被载入程序。若你的序列是DNATools格式时(DNA或寡核苷酸序列),程序不加注解的载入序列,程序模式调整成可以接受载入的数据类型(蛋白质、DNA和寡核苷酸引物序列)。在一个项目中可以加入几千个序列或引物,并在整个项目中分析这些序列及标题。这个程序的一个特点是给每个序列或引物添加文本标题。这样就可以用自定义的标题识别序列,而不必通过它们的文件名。为避免丢失数据-和你的工作-DNATools包括几个挽救丢失数据的功能:一个5层的撤销/重复功能,重新获得原始序列的恢复功能,项目中加入新文件时的安全备份功能,以及在一定的时间间隔作完全备份或备份你的工作。
2、限制酶切位点分析 这是分子生物学研究过程中最基本的操作,虽然可以通过文本编辑软件来查找,但过程烦琐(特别是序列长而分析的酶切位点多时)。通过专门的限制酶分析软件,不但方便,不易出错;而且输出的格式多样,满足用户的各种要求。推荐软件:DNAssist 1.0 能进行限制酶分析的软件很多,而我们建议使用DNAssist 1.0;原因是大多软件只对线性序列进行分析,那么cNNNNN…NNNgaatt环状的序列就找不到EcoR I的位点。DNAssist 1.0能很容易把这个EcoR I位点找出来。另外DNAssist在输出上非常完美,除了图形、线性显示外,还有类似DNASIS的列表方式,列出有的位点(按酶排列,按碱基顺序排列)。
同类软件:Primer Premier 5.0 见下。
DNAClub: DNA Club是一个简单的对DNA进行与PCR有关的操作的软件。它的功能有: 1、输入DNA序列; 2、查找ORF序列; 3、把DNA翻译成蛋白序列; 4、查找酶切位点; 5、查找PCR引物序列。 功能虽然都很简单,但非常实用,一般的用户都可以很方便地使用。
3、引物设计 优秀的引物设计软件能从模板序列中按用户的要求挑选出一系列引物序列,同时把这些序列的所有特性(包括分子量、Tm值、二级结构、上下有引物间的错配、3’端的稳定性及GC含量等等)分析出来。当然,这里的引物包括PCR引物、测序引物和探针。推荐软件:Primer Premier 5.0 Primer Premier5.0是由加拿大的Premier公司开发的专业用于PCR或测序引物以及杂交探针的设计和评估的软件,和Plasmid Premier2.02一起是该公司推出的最新的软件产品。其主要界面同样也是分为序列编辑窗口(Genetank),引物设计窗口(Primer Design),酶切分析窗口(Restriction Sites)和Motif分析窗口。这里我们主要介绍其引物设计功能,顾名思义,该软件就是用来进行引物设计的。可以简单地通过手动拖动鼠标以扩增出相应片段所需的引物,而在手动的任何时候,下面显示各种参数的改变和可能的二聚体、异二聚体、发夹结构等。也可以给定条件,让软件自动搜索引物,并将引物分析结果显示出来。而且进行这些操作非常简单。其功能绝不在Oligo 5.0之下!
同类软件:DNAClub 见上。
4、序列的同源比较(Alignment)具有这一功能的软件或软件包很多,但功能全面,界面友好,同时输出结果美观实用的不多。推荐软件: GeneDoc 3.2 GeneDoc能用用亮丽的色彩来区分相互间序列的同源性,输出的格式一目了然,而且可以报告为进化树的格式。选择项多,可以达到所需的要求。功能多又强,但要完全掌握,并不是很轻松的事。
同类软件:
MACAW 2.05多序列构建与分析工作台软件(Multiple Alignment Construction & Analysis Workbench, MACAW)是一个用来构建与分析多序列片段的交互式软件。MACAW具有几个特点:1. 新的搜索算法查寻类似区,消除了先前技术的许多限制。2. 应用一个最近发展的数学原理计算block类似性的统计学显著性。3. 使用各种视图工具,可以评估一个候选block包含在一个多序列中的可能性。4. 可以很容易地编辑每一个block。在多序列中查找一个类似片段并不是一件简单的事,主要是因为要查找的量极大。这正式MACAW所要解决的问题。 Clustal X 用来对核酸与蛋白序列进行多序列比较(multiple sequence alignment)的软件。多序列比较在分子生物学中是一个基本方法,用来发现特征序列,进行蛋白分类,证明序列间的同源性,帮助预测新序列二级结构与三级结构,确定PCR引物,以及在分子 进化分析方面均有很大帮助,Clustal X很适合这些方面的要求。
5、质粒绘图 就象限制酶切位点分析一样,也是最常用的功能。好的质粒绘图软件首先能对对已知序列自动作图;如果是未知序列,但根据用户已知的质粒信息也能绘出漂亮的图谱来。推荐软件:Gene Construction Kit 2.0 Gene Construction Kit 2.0这一个非常好的质粒构建软件包。与大多数分析的软件不同,它制作并显示克隆策略中的分子构建过程;包括质粒构建,模拟电泳条带;当然还可以质粒作图(有无序列均可)。通过它绘出来的图还可以继续用来构建克隆策略图谱。只是该软件由于功能太强,使用起来也不简单;幸亏它附有详细的使用帮助,认真研究后,应该没有问题。 同类软件:Winplas 2.6该软件用来绘制发表质量的质粒图,可广泛应用与论文、教材的质粒插图。其特性包括:1.知道序列或不知序列结构均能绘制质粒图;2. 可读入各种流行序列格式文件引入序列信息;3. 自动识别限制位点 可构建序列结构,功能包括:从文件插入序列、置换序列、序列编辑、部分序列删除等;4. 绘图功能强大,功能包括:位点标签说明、任意位置文字插入、生成彩图、线性或环形序列绘制、可输出到剪贴板、可输出到图像文件;5. 限制酶消化分析报告输出与序列输入报告功能。 Plasmid Premier2.02是由加拿大的Premier Biosoft公司推出的用于质粒作图的专业软件,主要用于进行质粒作图,质粒特征分析和质粒设计。其主要界面分为序列编辑窗口(Genetank),质粒作图窗口(Plasmid Design),酶切分析窗口(Restriction Sites)和纹基分析窗口(Motif)。打开程序就可进入序列编辑窗口,可以直接打开Genbank或Vector数据库中已知质粒的序列文件,将序列读入,并将有关于质粒的各种特征,包括编码区,启动子,多克隆位点以及参考文献等信息保存在Header中;也可以直接输入序列进行未知质粒的设计。
6、结构域(motif)查找。 推荐软件:Primer Premier 5.0 Primer Premier 5.0的结构域查找功能与它的引物设计一样强,结果能以图形、表格、序列三种方式输出。同时还提供了一些未知的结构域的列表;当然软件本身也提供了大量的已知结构域的序列。
7、RNA二级结构预测 二级结构分析和预测的软件很多,但绝大部分都只能在Mac和Unix下使用,在Windows平台下的很少。 推荐软件:RNAdraw 1.1b2 RNAdraw是一个进行RNA二级结构计算的软件。 1. 它 是Windows下的多文档窗口 (multiple document interface) 软件,允许你同时打开多个数据处理窗口。主窗口的工具条提供一些基本功能:打开文件、导入文件、关闭文件、设置程序参数、重排窗口、以及即时帮助和退出程序。2. RNAdraw中一个非常非常重要的特征是鼠标右键菜单打开的菜单显示对鼠标当前所指向的对象/窗口可以使用的功能列表。 3. RNA文库(RNA Library)用一种容易操作的方式来组织你所有的RNA数据文件。 同类软件:RNAStructure 3.5 RNA Structure 根据最小自由能原理,将Zuker的根据RNA一级序列预测RNA二级结构的算法在软件上实现。预测所用的热力学数据是最近由Turner实验室获得。提供了一些模块以扩展Zuker算法的能力,使之为一个界面友好的RNA折叠程序。
8、蛋白二级结构分析 推荐软件:Antheprot 4.5 蛋白序列分析软件包ANTHEPROT 4.5是位于法国的蛋白质生物与化学研究院(Institute of Biology and Chemistry of Proteins)用十多年时间开发出的蛋白质研究软件包。软件包包括了蛋白质研究领域所包括的大多数内容,功能非常强大。应用此软件包,使用个人电脑,便能进行各种蛋白序列分析与特性预测。更重要的是该软件能够提供蛋白序列的一些二级结构信息,使用户有可能模拟出未知蛋白的高级结构。
9、三维结构显示 通过各种方法计算出来的各种三维结构的数据,只要在三维结构浏览软件的帮助下,才能显示出来;从而使用户形象地看到生物大分子的三维立体结构。推荐软件:RasMol 2.7.1 RasMol 2.7是计算化学与分子图形学以及信息产业的同步高速发展的成果,使一个普通的科研工作人员,在自己的个人电脑上,就可以从Internet上的各种免费数据库中,下载所需观察与研究的分子坐标文件,进而通过RasMol 2.7以各种模式、各种角度,甚至按照自己的意愿旋转着,观察此分子神秘的微观三维立体结构,进而了解化合物分子结构和各种微观性质与宏观性质之间的定量关系。RasMol 2.7的作者是Glaxo&Wellcome公司(世界第一大制药公司)研发中心的科学家Roger Sayle。 RasMol 2.7最大的特点是界面简单,基本操作简单,运行非常迅速,对机器的要求较低,对小的有机分子与大分子,如蛋白质、DNA或RNA, 均能适用,且显示模式非常丰富。而且它程序短小,功能强大。尚无在功能上出其右者。其显示窗口可以很简单通过选择相应的菜单来显示分子的三维结构。用命令行窗口,通过输入命令可以执行和显示复杂和要求极高的三维图形。 同类软件:Cn3D Cn3D是由NCBI开发的用于观看蛋白质三维结构的软件,其设计的主要目的是为NCBI在其站点中的蛋白质结构数据库MMDB提供专业的结构观察软件,其主要的操作界面分为两个窗口,如右图所示,一个为结构窗口,另一个为序列窗口。与其他的类似的软件,如Rasmol,Weblabview等相比,其在结构观察方面主要功能上基本相似,但是图形格式上比Rasmol和Weblabview要差一些。而在与网络连接上,该软件能依托NCBI所建立的所建立的MMDB结构数据库,能直接根据输入的序列号从数据库中利用其内嵌的Entrez搜索引擎调出蛋白结构来进行观察,比其他软件要简便。而Cn3D主要的特点是能够将两个蛋白放在一起直观地进行三维结构上的比较,如下图中是将两种核酸外切酶的三维结构通过VAST对准得到的结构比较图:同样,Cn3D在结构比较方面也能利用其内嵌的Blast搜索引擎直接访问Genbank数据库找到具有局部相似性的结构数据,并在三维结构图中显示出二者具有相似性的结构区域。
10、格式转换 各种软件为了自己软件的需要有不同的格式,对我们使用数据带来了困难;格式转换软件能帮助用户解决这一问题。推荐软件:Seqverter 1.3 Seqverter 1.3使用简单,填写输入文件和输出文件名就可以完成格式转换。而且能够转换的格式非常之多是其它软件所无法比拟的。另外,它可在线升级以读写更多格式文件。 同类软件:Visual Sequence Editor 1.1 1.可以同时显示编辑多达200个核酸蛋白序列 2.将所有程序打包成一个库文件 3.可以输入输出到很多格式序列: IG/Stanford;GenBank/GB;NBRF;EMBL;GCG;DNA Strider;Fitch;Pieron/FASTA;PIR/CODATA以及普通文本 4.可以直接读取MACAW的文件,并能将各序列中的同源顺序标出 5.根据遗传密码(可以自定义)读出一种或六种阅读框 6.查看的字体大小,字母间隔等灵活可调 7.模糊查找特定序列。
11、电泳图谱分析 推荐软件:band leader 3.0 提供处理DNA或蛋白分子凝胶电泳图象和从凝胶电泳图象获得相关数据的工具。它可以对电泳图谱进行半定量分析,识别扫描得到的WINDOWS图象格式 .BMP,是一个难得的好软件。
三、 实验结果输出阶段
1、生成出版质量的图片 论文发表时,一般分子结构图的质量要求较高,如果直接从RasMol等软件中抓取图片,往往都不够精美。 推荐软件:Pov-Ray for Windows 3.1 Pov-Ray for Windows 3.1 相当于一种语言,它可以修改pov格式文件,用户还可以自己设定光源、摄像机、材质、阴影等因素,把生物分子的表面用材质填充;并根据光源、摄像机得到生物大分子的三维图。分辨率最大可达128undefined1024。
同类软件:molmol 2.5.1该软件能将pdb等格式的蛋白文件通过转换,存成普通的图形文件。
2、向数据库递交序列 推荐软件:Sequin 2.90 Sequin 2.90能向GenBank、EMBL、DDBJ三大核酸序列库递交序列。可以不用仔细考虑各数据库文件的具体格式,以问答填表格的形式,将需递交的序列和相关资料填好。可以在线直接发送,也可以生成相应格式文件,以e-mail形式发送。
Annhyb AnnHyb2.22是设计PCR引物和DNA探针的辅助软件。主要功能有: 1.对寡核苷酸序列,计算溶解温度(nearest-neighbour algorithm,可以设定盐浓度和引物浓度), 序列长度、GC百分比,分子量, 摩尔消光系数,IUB/IUPAC序列、反向序列、互补序列。 2.对50个bp以上的序列, 计算长度,GC百分比、不同盐浓度下的溶解温度 3.对长序列可以进行反向互补序列。 4.可以读出序列
Cervus CERVUS 是一个根据共显性资料来推测家系似然性的WINDOWS95程序,它可以用来计算等位基因发生概率,运行模拟系统来判定似然比率的临界值和分析动植物种群中的家系。这种模拟系统还可能应用于评估家系推测中所使用的系列单位点标记物的分辨能力。
Clonemap Clonemap是由CGC公司设计的用于辅助克隆设计的质粒图谱绘制软件。该软件除了能实现质粒图谱的绘制功能以外,还有其他一些辅助功能,如对序列进行翻转,按六个阅读框进行蛋白翻译,寻找开读框,搜索特定序列。 Dicroprot DICROPROT software for Windows Release 2.0 beta 是法国蛋白生化研究所蛋白构象研究室Gilbert Deleage博士编制的用于观察蛋白质圆二色谱的软件,是一个较为小巧、相对较为专业、操作简单的研究辅助工具。
Digest DIGEST 能够扫描DNA序列并查找限制性内切酶位点.它支持使用者限定搜索特定的酶,如果此酶在限制性酶切酶库文件WISCONSI.920中,它会列出酶切位置并对酶切片段按长度排序.它使用Don Gilbert的读序模块UREADSEQ,所以读出大部分的序列文件格式。
DNAMend DNAmend是一名德国人编写的用于对DNA序列进行分析,编辑的专业软件,主要的用途是在基因克隆过程中,利用其提供的对DNA序列的限制性酶切分析,开读框搜索,序列转译,末端修剪等功能对DNA序列进行酶切、连接、末端补平等模拟操作,为克隆流程设计及克隆过程监视提供直观的控制工具,便于对克隆中可能出现的问题进行分析,并可将结果输出用于发表文章。
ISIS DRAW 非常非常棒的化学结构式绘制软件,能绘制各种复杂的化学分子(包括有机大分子)的平面及立体结构,还能绘制复杂的化学反应方程式,是写论文和考试试卷的常备工具。强烈推荐给有关生物化学研究及教学的工作人员。 MatInd 使用MatInd对一组DNA序列或一个核酸分布矩阵进行分析后,能够产生描述转录因子结合位点的矩阵型来。这些矩阵型又可以被MatInspector用来扫描核甘酸序列并找到与之匹配的结合位点。
MatInspector 利用MatInd所生成的矩阵型对核甘酸序列进行扫描并使之与结合位点匹配。在输出文件中包括匹配位置的核心序列、矩阵相似性、具体位置及序列等。
Molgen OLGEN软件包可以根据分子式计算并估计其所有的异构体,MOLGEN为科学和教育在阐明分子的结构组成方面提供了有效方便而廉价的分析工具。MOLGEN结合MOLED和MOLVIEW两个软件,通过描述目的分子包含的亚结构的限制,减少异构体的数量,以便更快的获得目的分子的空间的结构的具体构像;通过MOLVIEW可以计算分子的最稳定合理的立体的结构,并且通过旋转可以从个个角度观察分子的结构情况,和每一个原子的空间的位置。
Mvsp MVSP是一个多变量统计分析软件包。它能进行多种等级特征分析:1,主成分分析(PCA), 2, 主坐标分析(PCO), 对应/对应分离趋势分析(CA/DCA)和典型对应分析(CCA)等。 另外,它还可以通过使用20种不同的距离测量或相似测量及7种分族策略进行族分析。分析得到的多样性指数就可以用于生态学数据的计算,这些指数包括辛普森指数,香农信息指数和Brillouin指数。目前这一版本是一个评估版(限时30天,限次15次),如果你想继续使用就必须付钱买正式版,价格为$140。
PCMolecule PCMolecule 是观察三维分子结构的软件,能够观察以.mcm格式保存的三维分子。
PCrare PC-Rare是应用八聚体频率出现的不一致性的方法(OFD)来设计PCR的引物,此程序简单而有效。
Promsed ProMSED是一个在Windows 3.11/95下运行的简单易用的程序,通过它能够自动或手动进行蛋白质多序列的比较和编辑。该程序能自动识别以下文件格式:NBRF/PIR、Pearson(Fasta)、EMBL/SwissProt、CLUSTAL和GCG/MSF等,同时它的界面及主要功能与通用的文本编辑语言相似。其中自动比较只对Clustal V 运算法则有效,当使用群体操作或用不同颜色表示不同性质及功能氨基酸时,人工比较和序列可视分析就显得更方便一些。随着目前计算机内存的日益扩大,程序中使用序列的大小及个数都没有什么限制了。更可贵的是ProMSED能够比较全部序列、全部序列中的任何子集和序列中选定的某一区域,并且提供一些用来进行序列分析、可视编辑及以图像展示结果的工具。
Redsoft Plasmid Redasoft Plasmid 的设计是用来帮助生命科学家快速而轻松地绘制专业级质量的质粒载体图,Redasoft Plasmid 主要的性能包括:快速和轻松地生成一个清晰,色彩鲜明的环行或线性的载体图。自动识别和解析序列文件。新增的web浏览功能允许你链接到数据库站点,并支持下载和自动将序列文件转换成载体图。强大的限制性内切酶分析功能和拥有将近1000种来自REBASE的限制性内切酶。片段的删除和插入完全模拟克隆实验并和其他图形兼容。所看即所得 (What You See Is What You Get)的编辑环境使得你的打印结果和你在屏幕上看到的保持一致。自动标记各种区段的碱基位置以避免重叠。可选择的图形比例尺使几个构造图间很容易比较。允许质粒图拷贝到剪贴板并粘贴到其他Windows应用程序。可以用e-mail的方式传递载体图。
Simplot本软件与Phylip95协同应用, 它基于Don Gilbert's ReadSeq的编码的形式能够识别多种序列的格式。首先,将序列对齐,将它们以标准的格式保存,如FASTA/Pearson的格式。SimPlot同时可做26组的序列或26个单独的序列,当然,可以只选择感兴趣的序列。 TreeView Tree View是用来生成与打印进化树的软件,它可以读取NEXUS与PHYLIP生成的进化树格式文件,生成进化树,并输出到打印机。
10、格式转换 各种软件为了自己软件的需要有不同的格式,对我们使用数据带来了困难;格式转换软件能帮助用户解决这一问题。推荐软件:Seqverter 1.3
Seqverter 1.3使用简单,填写输入文件和输出文件名就可以完成格式转换。而且能够转换的格式非常之多是其它软件所无法比拟的。另外,它可在线升级以读写更多格式文件。
同类软件:Visual Sequence Editor 1.1 1.可以同时显示编辑多达200个核酸蛋白序列 2.将所有程序打包成一个库文件 3.可以输入输出到很多格式序列: IG/Stanford;GenBank/GB;NBRF;EMBL;GCG;DNA Strider;Fitch;Pieron/FASTA;PIR/CODATA以及普通文本 4.可以直接读取MACAW的文件,并能将各序列中的同源顺序标出 5.根据遗传密码(可以自定义)读出一种或六种阅读框 6.查看的字体大小,字母间隔等灵活可调 7.模糊查找特定序列。
11、电泳图谱分析 推荐软件:band leader 3.0
提供处理DNA或蛋白分子凝胶电泳图象和从凝胶电泳图象获得相关数据的工具。它可以对电泳图谱进行半定量分析,识别扫描得到的WINDOWS图象格式 .BMP,是一个难得的好软件。
三、 实验结果输出阶段
1、生成出版质量的图片 论文发表时,一般分子结构图的质量要求较高,如果直接从RasMol等软件中抓取图片,往往都不够精美。 推荐软件:Pov-Ray for Windows 3.1
Pov-Ray for Windows 3.1 相当于一种语言,它可以修改pov格式文件,用户还可以自己设定光源、摄像机、材质、阴影等因素,把生物分子的表面用材质填充;并根据光源、摄像机得到生物大分子的三维图。分辨率最大可达128undefined1024。
同类软件:molmol 2.5.1该软件能将pdb等格式的蛋白文件通过转换,存成普通的图形文件。
2、向数据库递交序列 推荐软件:Sequin 2.90
Sequin 2.90能向GenBank、EMBL、DDBJ三大核酸序列库递交序列。可以不用仔细考虑各数据库文件的具体格式,以问答填表格的形式,将需递交的序列和相关资料填好。可以在线直接发送,也可以生成相应格式文件,以e-mail形式发送。
Annhyb AnnHyb2.22是设计PCR引物和DNA探针的辅助软件。主要功能有: 1.对寡核苷酸序列,计算溶解温度(nearest-neighbour algorithm,可以设定盐浓度和引物浓度), 序列长度、GC百分比,分子量, 摩尔消光系数,IUB/IUPAC序列、反向序列、互补序列。 2.对50个bp以上的序列, 计算长度,GC百分比、不同盐浓度下的溶解温度 3.对长序列可以进行反向互补序列。 4.可以读出序列
Cervus CERVUS 是一个根据共显性资料来推测家系似然性的WINDOWS95程序,它可以用来计算等位基因发生概率,运行模拟系统来判定似然比率的临界值和分析动植物种群中的家系。这种模拟系统还可能应用于评估家系推测中所使用的系列单位点标记物的分辨能力。
Clonemap Clonemap是由CGC公司设计的用于辅助克隆设计的质粒图谱绘制软件。该软件除了能实现质粒图谱的绘制功能以外,还有其他一些辅助功能,如对序列进行翻转,按六个阅读框进行蛋白翻译,寻找开读框,搜索特定序列。
Dicroprot DICROPROT software for Windows Release 2.0 beta 是法国蛋白生化研究所蛋白构象研究室Gilbert Deleage博士编制的用于观察蛋白质圆二色谱的软件,是一个较为小巧、相对较为专业、操作简单的研究辅助工具。
Digest DIGEST 能够扫描DNA序列并查找限制性内切酶位点.它支持使用者限定搜索特定的酶,如果此酶在限制性酶切酶库文件WISCONSI.920中,它会列出酶切位置并对酶切片段按长度排序.它使用Don Gilbert的读序模块UREADSEQ,所以读出大部分的序列文件格式。
DNAMend DNAmend是一名德国人编写的用于对DNA序列进行分析,编辑的专业软件,主要的用途是在基因克隆过程中,利用其提供的对DNA序列的限制性酶切分析,开读框搜索,序列转译,末端修剪等功能对DNA序列进行酶切、连接、末端补平等模拟操作,为克隆流程设计及克隆过程监视提供直观的控制工具,便于对克隆中可能出现的问题进行分析,并可将结果输出用于发表文章。
ISIS DRAW 非常非常棒的化学结构式绘制软件,能绘制各种复杂的化学分子(包括有机大分子)的平面及立体结构,还能绘制复杂的化学反应方程式,是写论文和考试试卷的常备工具。强烈推荐给有关生物化学研究及教学的工作人员。
MatInd 使用MatInd对一组DNA序列或一个核酸分布矩阵进行分析后,能够产生描述转录因子结合位点的矩阵型来。这些矩阵型又可以被MatInspector用来扫描核甘酸序列并找到与之匹配的结合位点。MatInspector 利用MatInd所生成的矩阵型对核甘酸序列进行扫描并使之与结合位点匹配。在输出文件中包括匹配位置的核心序列、矩阵相似性、具体位置及序列等。
Molgen OLGEN软件包可以根据分子式计算并估计其所有的异构体,MOLGEN为科学和教育在阐明分子的结构组成方面提供了有效方便而廉价的分析工具。MOLGEN结合MOLED和MOLVIEW两个软件,通过描述目的分子包含的亚结构的限制,减少异构体的数量,以便更快的获得目的分子的空间的结构的具体构像;通过MOLVIEW可以计算分子的最稳定合理的立体的结构,并且通过旋转可以从个个角度观察分子的结构情况,和每一个原子的空间的位置。
Mvsp MVSP是一个多变量统计分析软件包。它能进行多种等级特征分析:1,主成分分析(PCA), 2, 主坐标分析(PCO), 对应/对应分离趋势分析(CA/DCA)和典型对应分析(CCA)等。 另外,它还可以通过使用20种不同的距离测量或相似测量及7种分族策略进行族分析。分析得到的多样性指数就可以用于生态学数据的计算,这些指数包括辛普森指数,香农信息指数和Brillouin指数。目前这一版本是一个评估版(限时30天,限次15次),如果你想继续使用就必须付钱买正式版,价格为$140。
PCMolecule PCMolecule 是观察三维分子结构的软件,能够观察以.mcm格式保存的三维分子。
PCrare PC-Rare是应用八聚体频率出现的不一致性的方法(OFD)来设计PCR的引物,此程序简单而有效。
Promsed ProMSED是一个在Windows 3.11/95下运行的简单易用的程序,通过它能够自动或手动进行蛋白质多序列的比较和编辑。该程序能自动识别以下文件格式:NBRF/PIR、Pearson(Fasta)、EMBL/SwissProt、CLUSTAL和GCG/MSF等,同时它的界面及主要功能与通用的文本编辑语言相似。其中自动比较只对Clustal V 运算法则有效,当使用群体操作或用不同颜色表示不同性质及功能氨基酸时,人工比较和序列可视分析就显得更方便一些。随着目前计算机内存的日益扩大,程序中使用序列的大小及个数都没有什么限制了。更可贵的是ProMSED能够比较全部序列、全部序列中的任何子集和序列中选定的某一区域,并且提供一些用来进行序列分析、可视编辑及以图像展示结果的工具。
Redsoft Plasmid Redasoft Plasmid 的设计是用来帮助生命科学家快速而轻松地绘制专业级质量的质粒载体图,Redasoft Plasmid 主要的性能包括:快速和轻松地生成一个清晰,色彩鲜明的环行或线性的载体图。自动识别和解析序列文件。新增的web浏览功能允许你链接到数据库站点,并支持下载和自动将序列文件转换成载体图。强大的限制性内切酶分析功能和拥有将近1000种来自REBASE的限制性内切酶。片段的删除和插入完全模拟克隆实验并和其他图形兼容。所看即所得 (What You See Is What You Get)的编辑环境使得你的打印结果和你在屏幕上看到的保持一致。自动标记各种区段的碱基位置以避免重叠。可选择的图形比例尺使几个构造图间很容易比较。允许质粒图拷贝到剪贴板并粘贴到其他Windows应用程序。可以用e-mail的方式传递载体图。
Simplot本软件与Phylip95协同应用, 它基于Don Gilbert's ReadSeq的编码的形式能够识别多种序列的格式。首先,将序列对齐,将它们以标准的格式保存,如FASTA/Pearson的格式。SimPlot同时可做26组的序列或26个单独的序列,当然,可以只选择感兴趣的序列。
TreeView Tree View是用来生成与打印进化树的软件,它可以读取NEXUS与PHYLIP生成的进化树格式文件,生成进化树,并输出到打印机。