综述:脑组织透明成像技术
丁香学术
4854
大脑是神经系统的最高级部分,由左、右两个大脑半球组成,两半球间有横行的神经纤维相联系,结构和功能十分复杂。从大脑这个复杂的系统中获得清晰的、高分辨率的图像信息,同时保持理解系统功能所需的全局视角,是生物学的一项挑战 [1]。
近年来,国内外对于脑功能结构的研究日渐增多,脑科学成为研究热点。但由于大脑结构复杂,神经元众多且交联复杂,无法直观明确地看到大脑完整的结构。科学家们要构建单细胞分辨率的脑图集,必须准确识别整个大脑中的细胞 [2]。
研究者通过将脑组织染色,显微镜下观察从而得到脑组织中神经元、胶质细胞之间的网络结构图,但脑组织中的脂质、血细胞等成分阻碍了对于神经系统和大脑结构的观察,所以透明化是十分有必要的。
但传统的方法,如冷冻切片存在明显的缺点,无法观察连续的 3D 层面上的结构网络,工作量大且破坏神经组织,拍摄成像速度慢,一般至少需要数周,所以需要建立完整的大脑透明技术。
BABB, DISCO, CLARITY, PACT/PARS, CUBIC 和 Scale 等在内的有效的组织清除方法的最新发展 [3-8],可以与光片荧光显微镜结合使用对成年小鼠大脑进行快速高分辨率成像 [9-16]。建立完整的大脑透明化、染色、成像体系对于研究神经系统疾病等有重要作用。
本文将为大家重点介绍几项常用的透明化方法。
1. CLARITY
CLARITY 是一种将完整组织浸泡在纳米多孔水凝胶中,与凝胶形成网络杂交(交联至亲水性聚合物的三维网络)的方法。这种水凝胶与组织的杂交从物理上支持组织结构,使组织交联,将水凝胶单体与包括蛋白质,核酸和小分子在内的生物分子共价连接。清除时由于组织与水凝胶共轭连接,故不会破坏结构,SDS 去除脂质,使组织透明(图 1)。
该方法使完整的脑组织呈现光学透明且具有大分子可渗透性。小鼠的大脑经过透明和染色,可以实现局部神经网络延伸、细胞间联系、亚细胞结构、蛋白质复合物、核酸和神经递质的完整组织成像 [1]。
CLARITY 利用带电胶束的特性进行电泳,加速组织清除的速度(图 2)。利用离子的高度带电性质,将提取速度提高了几个数量级。
水凝胶确保生物分子和精细结构特征(如膜局部蛋白,突触),而会引起光散射并阻止大分子渗透的膜状脂质将被主动清除,从而留下了原本组装的生物组织,适合完整地标记和成像。
CLARITY 还可以实现完整的组织原位杂交:对大脑透明化后,在未切片的组织中进行多轮免疫组化,以及可以在完整的成年小鼠大脑中进行抗体标记。
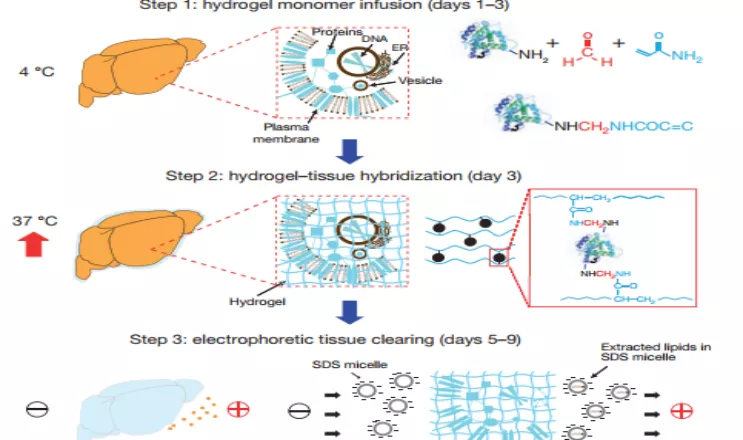
图 1:CLARITY 原理步骤
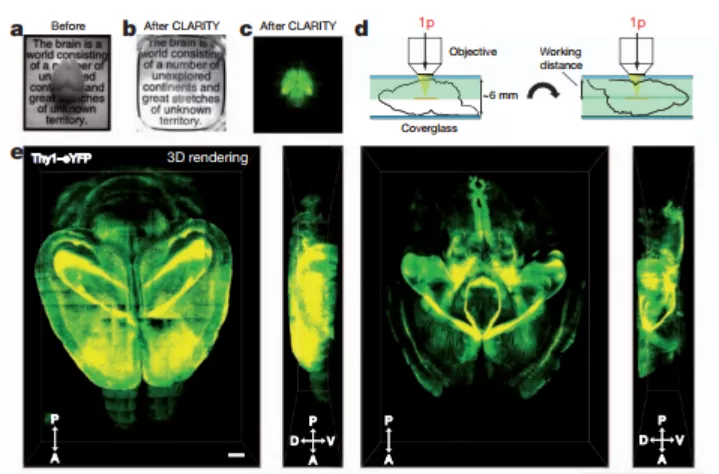
图 2:电泳清除大脑及成像图
CLARITY 的优点:
(1)可以对临床样品进行精细的结构分析。
(2)在水凝胶-组织杂交物中,荧光不易淬灭,保留更完整。
但是 CLARITY 最初的形式是使用电泳进行组织清除(ETC)来从大样品中提取脂质,但这对于易碎样品来说,可能会导致组织质量的变化,包括加热导致的组织褐变。
2. CUBIC
CUBIC 是一种用多组分配方,使得组织经过长时间的浸泡达到透明状态,得以在光镜下进行观察。此种方法简单有效,脑样本浸入含有氨基醇的混合物中,通过单光子激发显微镜便可快速进行全脑成像(图 3)。
经灌注固定后的组织器官按顺序浸泡到 1/2 Reagent-1、Reagent-1、1/2 Reagent-2、Reagent-2 中(表 1),37℃ 水浴,以 60 rpm 的速度轻微震荡即可透明。透明后的样品转移到 Imaging Oil 中固定 1 小时后可在光镜下观察。
CUBIC 适用于成人大脑中荧光蛋白或免疫染色样品的多色成像,并且与全脑细胞核复染方案和计算图像分析管道一起,可视化和量化环境刺激引起的神经活动 [17]。
CUBIC 可以通过单细胞分辨率对整个成人大脑进行时程表达分析。氨基醇成分的加入解决了之前透明方法中使用过氧化物去除血色素带来的一个缺点,即引起处理后组织的外部损伤和荧光信号的淬灭。
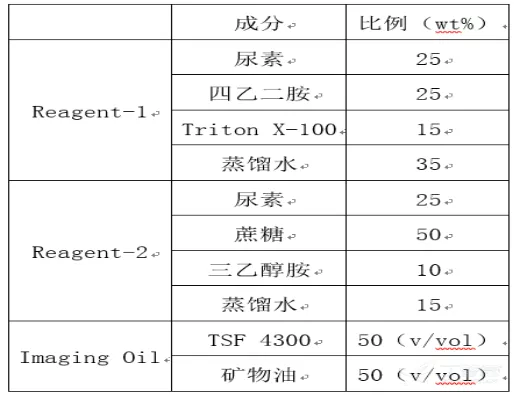
表 1:CUBIC 成分

 图 3:经 CUBIC 试剂处理后的小鼠大脑和成像图
图 3:经 CUBIC 试剂处理后的小鼠大脑和成像图
3. ScaleA2/ScaleS
ScaleA2 是利用化学溶剂清除的一种方法,使用 ScaleA2(水溶液试剂)对固定的大脑组织进行成像,该试剂使生物样品具有光学透明性,并完全保留了荧光信号。将组织浸泡在溶剂中一周,就可以得到透明的脑组织,但是组织膨胀率高达 10%-30%。
尿素溶液处理固定的脑组织,通过水合作用使组织透明,但相应的组织略膨胀,后续用 ScaleA2 溶液继续处理,ScaleA2 溶液包含 4M 尿素,10%(wt / vol)甘油和 0.1%(wt / vol)Triton X-100 组成。
甘油可以防止过多的水合作用并使组织扩张最小化。在 ScaleA2 溶液中孵育完整的固定小鼠大脑,孵育时间 2 周以上可充分清除脑组织。在有图案的背景下或通过 532 nm 激光的穿透,透明性是明显的(图 4)。
Scale S 方法改进了传统的配方,增加了山梨醇。山梨醇对脑的透明化更佳,尿素有水合作用,使组织膨胀,但山梨醇有脱水作用,让组织缩小,通过配比可以保留样本的原本体积。
优点:操作简单,配置溶液每天更新即可。
缺点:脑组织膨胀率 10%-30% 不等,影响组织原本状态,有可能破坏组织内原本结构。孵育时间长,溶液消耗大。
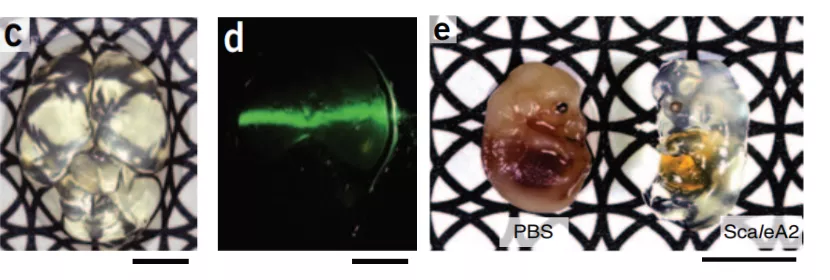
图 4:经 ScaleA2 孵育后的组织
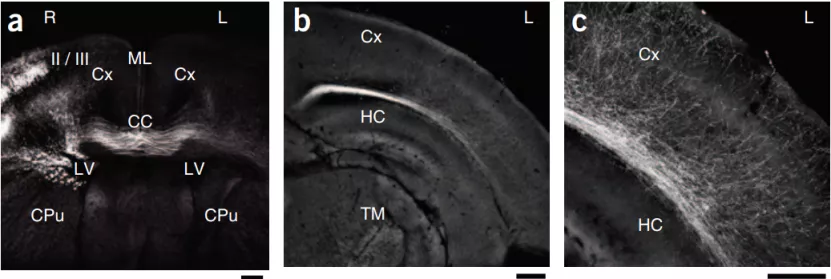
图 5: 共聚焦成像
4. BABB
BABB 是使用有机溶剂如苯甲醇/苯甲酸苄酯去除脂质,从而得到透明的组织的方法。有机溶剂可以提取脂质或减少折射率的变化 [4, 5, 16],但同时会猝灭荧光,从而限制了成像时间。
此外,BABB 中天然荧光的不稳定性限制了精细神经元投射或其他易于淬灭的信号的成像。所以 BABB 等有机法由于上述缺点如今已不常使用。
5. iDISCO+
iDISCO+是基于 DISCO 和 3DISCO 方法的改进版本(图 6)。iDISCO+也是一种用化学试剂浸泡使组织透明的方法,与之前介绍的方法不同的是,iDISCO+先对组织进行脱水和免疫染色,之后再清除组织中的脂质使组织透明(图 7)。
先用甲醇梯度脱水,之后用过氧化氢对组织进行漂白,清除溶液中含有 DCM、DBE 等,防止组织产生气泡和氧化。
此种方法操作简单,但是过氧化物清除的弊端在于破坏组织完整性,会使成像结果略模糊,DCM、DBE 也会一定程度上猝灭荧光信号。iDISCO+ 常用于 10.5-16.5 天的小鼠胚胎的透明。iDISCO+ 与开发的 clearMAP 成像分析技术结合改善了成像水平,并且软件开源性使得数据分析更加容易。

图 6:iDSCO\3DISCO 与 iDISCO+步骤区别
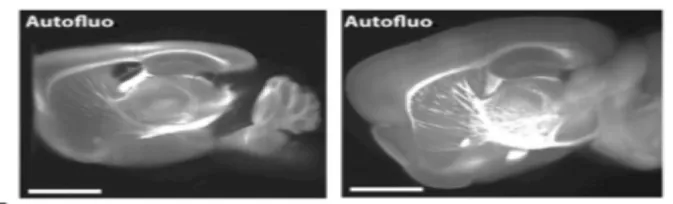
图 7:小鼠全脑经透明后 LSFM 扫描成像
6. PACT 和 PARS
PACT 是用于完整组织的组织清除和免疫染色的方法;PARS 是一种全身清除和免疫标记的方法。在啮齿动物中,PACT、PARS 与内源性荧光、免疫组织化学、FISH 相结合,可以长期保存,并和具有细胞和亚细胞分辨率的显微镜相兼容 [17]。这些方法适用于完整器官和体内需要高分辨率和表型分析的组织器官。
PACT 方法分为三步:组织与和水凝胶单体交联混合以稳定生物大分子;用离子去垢剂去除组织凝胶基质中的脂质;透明化的组织用 RIMS 包埋以成像或长期保存。
这种方法的优点是对大量组织的光学扫描成像,从而能够研究细胞间的关系和远距离神经连通性。结合荧光示踪剂,组织清除有助于识别相互作用的细胞结构,包括发散或会聚的神经和脉管系统。
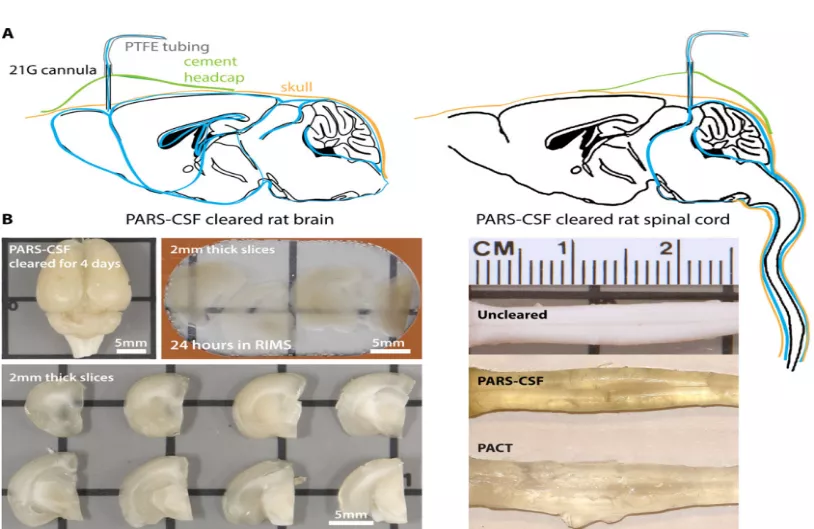
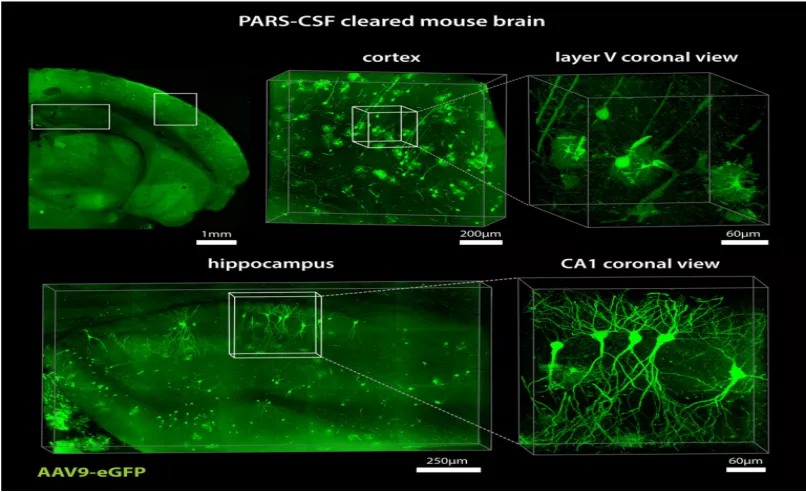
图 8:PACT 透明及扫描成像
总结与讨论
目前最为常用的方法是 CLARITY、CUBIC、ScaleS 等。CLARITY 方法自 2013 年问世以来,近年来对其电泳设备及染色方法的改进,极大地缩短了透明和染色时间 [18]。并且已有稳定的商业化透明设备,透明效果好、性能稳定,是目前应用较多的一种方法。
CUBIC 由于可同时进行透明和免疫染色,操作简单,不需要复杂的仪器即可完成,透明效果较好、成本较低,也是常用的方法之一。
ScaleS 对原始 Scale 方法进行改造,增加了山梨醇这一成分,减少组织膨胀率,并且操作简单,只需每日更换溶液即可完成透明。但有机溶剂法对荧光信号的淬灭较为严重,故而不常使用。
总的来说,透明化技术日益成熟,通过透明化可以实现大组织的透视以及多重免疫化学标记,甚至可以对神经细胞之间的相互作用立体成像,是神经科学领域研究强有力的技术手段。
参考文献:
1. Kwanghun Chung. et al. Structural and molecular interrogation of intact biological systems. Nature 497, 332-337(2013).
2. Bin Yang, Jennifer B. Treweek. Single-Cell Phenotyping within Transparent Intact Tissue Through Whole-Body Clearing.Cell 2014 158(4): 945–958.
3. Ragan, T. et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nature Methods 9, 255–258 (2012).
4. Dodt, H. U., Leischner, U. & Schierloh, A. Ultramicroscopy: three-dimensional visualization of neuronal networks in the whole mouse brain. Nature Methods 4, 331–336 (2007).
5. Ertu¨rk, A., Mauch, C., Hellal, F. & Fo¨rstner, F. Three-dimensional imaging of the unsectioned adult spinal cord to assess axon regeneration and glial responses after injury. Nature Med. 18, 166–171 (2012).
6. Hama, H. et al. Scale: a chemical approach for fluorescence imaging and
reconstruction of transparentmouse brain.Nature Neurosci. 14, 1481–1488(2011).
7. Cheong, W., Prahl, S. & Welch, A. A review of the optical properties of biological tissues. IEEE J. Quant. Electron. 26, 2166–2185 (1990).
8. Sykova, E. & Nicholson, C. Diffusion in brain extracellular space. Physiol Rev. 88, 1277–1340 (2008).
9. Denk, W., Strickler, J. H. & Webb, W. W. Two-photon laser scanning fluorescence microscopy. Science 248, 73–76 (1990).
10. Carmeliet, P. & Tessier-Lavigne, M. Common mechanisms of nerve and blood vessel wiring. Nature 436, 193–200 (2005).
11. Helmchen, F. & Denk, W. Deep tissue two-photon microscopy. Nature Methods 2, 932–940 (2005).
12. Denk, W. & Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS Biol. 2, e329 (2004).
13. Micheva, K. D.& Smith,S. J.Arraytomography:a newtoolfor imagingthemolecular architecture and ultrastructure of neural circuits. Neuron 55, 25–36 (2007).
14. Livet, J. et al. Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system. Nature 450, 56–62 (2007).
15. Li, A. et al. Micro-optical sectioning tomography to obtain a high-resolution atlas of the mouse brain. Science 330, 1404–1408 (2010).
16. Botcherby, E. J. et al. Aberration-free three-dimensional multiphoton imaging of neuronal activity at kHz rates. Proc. Natl Acad. Sci. USA 109, 2919–2924 (2012).
17. Etsuo A. Susaki,Kazuki Tainaka, Dimitri Perrin. Whole-Brain Imaging with Single-Cell Resolution Using Chemical Cocktailsand Computational Analysis. Cell 157, 726–739(2014).
18. Tatsuya C. Murakami1. A three-dimensional single-cell-resolution whole-brain atlas using CUBIC-X expansion microscopy and tissue clearing. Nature neuroscience (625–637), 2018.