下载别人的数据,发表自己的论文 | 论文
丁香园
对于做肿瘤研究的科技工作者来说,肿瘤基因芯片数据库的熟练使用已成为必备的技能。
TCGA 是目前最大的癌症基因信息的数据库,然而并不提供相关分析,这对于小白来说有点麻烦了。
相比之下,Oncomine(目前世界上最大的癌基因芯片数据库和整合数据挖掘平台)就要友好多了。
其相对简单的操作,相信大家一定有所了解了;然而,Oncomine 并不提供免费的午餐——在用学术邮箱注册登陆后仍然有诸多限制。
其中,最大的限制是不能下载数据。
什么?好不容易找到一个满意的数据,不能下载原始数据?也不能保存可编辑图片!
你能做的只能截个图了… …(然后你就在纠结用 QQ 还是搜狗截屏像素更好了吧?一脸斜线…)
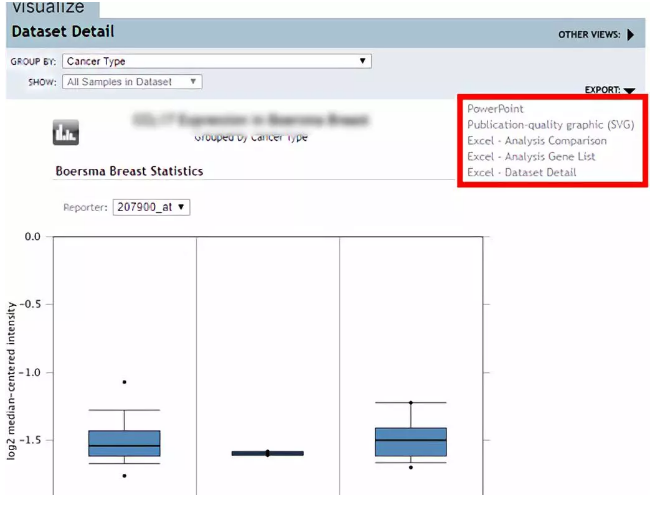
于是网上就有了如何用 photoshop 或 illustrator 描图的教程了… 再次满脸斜线… (如果想统计差异怎么办?)

基于以上原因,特写以下教程分享给大家。原汁原创,全网首发。
欧耶~ 下面上干货!
1 在找到满意的数据后,先选择以 bar 图显示;再在图表上右键选择审查元素(如下图,英文版浏览器选择 Inspect,建议使用 chrome 内核浏览器);

2 然后,再次在图表上右键选择审查元素即可自动跳到 map 这一项,再右键复制元素;并将复制的元素粘贴到 txt 文本中;
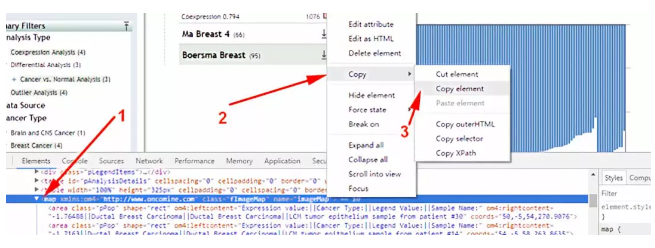
3 接下来就用 R 语言提取刚才文本中的数据(只需要把以下代码粘入 R 软件中即可):
x=read.table(choose.files(),header=F,sep="\n",quote = "" ,comment.char="")
#在粘贴上面一句后会弹出窗口让你选择打开刚才保存的 txt 文本
x <- as.character(x[1,1])
x=strsplit(x,split=" ")
y=unlist(x)
z=y[grep("om4:rightcontent",y)]
a=unlist(strsplit(z,"\\=\""))
b=matrix(a,2)[2,]
#以上代码在 oncomine 中下载基因表达数据是通用的,运行后的结果如下图

可以看到,我们想要的数据基本已经摘出来了(前面为表达值,后面为肿瘤样本类型)。
4 接下来可以用以下代码将数据保存为 csv 文件后在 excel 中分割后获得数据(如下图)
setwd(choose.dir())# 选择存储路径
write.csv(b,"result.csv")
如下图,对数据分列后即可得到想要的数据,然后就可以在 graphpad 等软件中作图了。

5 除上述方法外,也可以直接在 R 中运行以下代码清洗数据(注意根据自己的数据修改肿瘤类型)
ductal=b[grep("Ductal",b)];
Ductal=t(matrix(unlist(strsplit(ductal,"\\|")),3)[c(1,3),]);
lobular=b[grep("Lobular",b)];
Lobular=t(matrix(unlist(strsplit(lobular,"\\|")),3)[c(1,3),]);
high=b[grep("High",b)];
High=t(matrix(unlist(strsplit(high,"\\|")),3)[c(1,3),]);
# 将 Ductal、Lobular 等相应的替换为你的样本分类名字,如 Normal 或 cancer
dataset <-rbind(Ductal,Lobular,High);# 合并数据
运行后即可得到如下的数据结果:

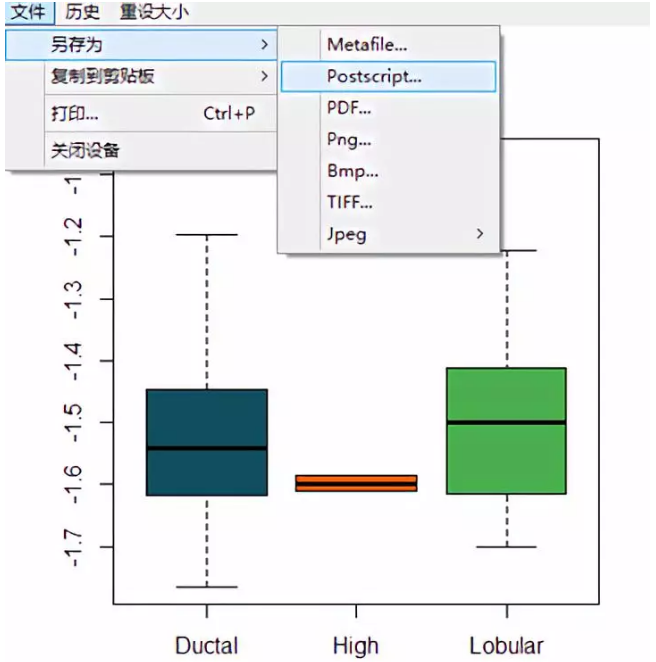
然后运行以下代码直接绘图
保存为你想要的格式的图即可(Metafile 为矢量可编辑格式,可在 illustrator 里编辑哦)
colnames(dataset)=c("value","group");
boxplot(as.numeric(dataset[,1]) ~ group,dataset, col = c("#124D60", "#EB6017","#4CAE50"))
6 几点补充:代码可能还可以进一步优化,还可以用 R 语言中的 Rselenium 软件包更加自动化的获取数据。
本教程旨在提供一种免费获取 oncomine 数据的思路,在应用以上代码时有可能需要稍微调整。
有问题可留言讨论,也可 VX 或邮箱交流,谢谢!