实验时间 | 筛选基因总不好?这个网站不可少
丁香园
基因功能富集分析已经成为生物信息学中几乎每篇文章的必备套路。
差异表达得到的显著的基因做一个基因功能富集分析,看看是否富集在一些感兴趣的功能上;共表达分析得到的基因模块,做一个基因功能富集分析,看看这个模块主要跟哪些功能相关;相关分析得到的候选基因,做一个基因功能富集分析,看看这些与性状相关的基因的有一些什么功能。
此外基因功能富集分析还可以用来帮助筛选基因,确定未知功能的基因簇的潜在功能等作用。
今天就给大家安利一个好用的基因功能富集分析网站 WebGestalt 。
相比应用较为广泛的 DAVID (https://david.ncifcrf.gov/ )WebGestalt 具有以下优势:
1. 结果可视化更漂亮,比如可以得到 GO 注释的有向无环图,直接放在文章中,高大上又直观;疾病注释的结构图等;
2. 操作更方便,点一点鼠标,就可以下载下来整理好的数据,而 DAVID 里面下载出来的东西还需要转化之后用其他的软件进行绘图,甚是麻烦;
3. 输入更优化,表现在对输入的 gene symbol 数目没有限制,DAVID 的数据库目前不能接受大于 3 000 个 gene symbol 的输入,基因数目一大就麻烦,还需要输入几次,还能够识别的输入类型更多样;
4. 2017 年的更新版本还嵌入了第二代的功能富集分析方法 GSEA,拓扑网络分析算法等。
接下来是手把手进行一次功能富集分析的操作教程,废话不多说,上图。
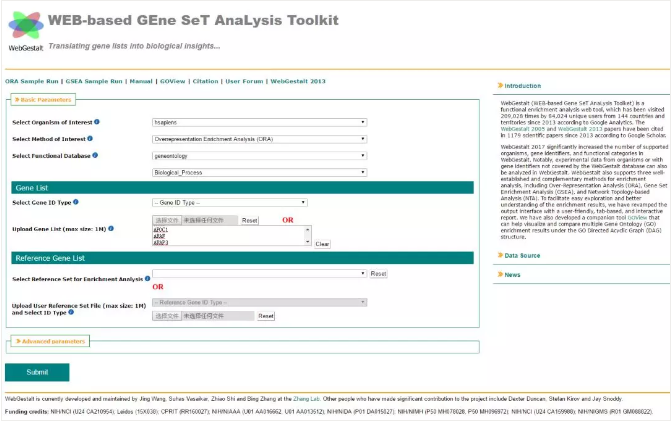
01 打开网站
界面介样 ↓
02 选择目标物种
目前 WebGestalt 网站支持 12 个不同的物种,具体包括 athaliana(拟南芥), btaurus(牛), celegans(线虫), cfamiliaris(狗), dmelangaster(黑腹果蝇),drerlo(斑马鱼), ggallus(原鸡), hsapiens(人类), mmusculus(小鼠), morvegicus(褐鼠),scerevisiae(酿酒酵母), sscrofa(野猪)。

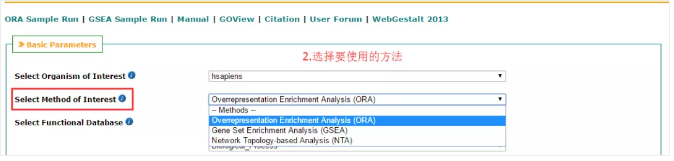
03 选择要使用的方法
2017 年 WebGestalt 进行了更新后,可以进行 overrepresentation enrichment analysis(ORA),Gene Set Enrichment Analysis(GSEA)和 Network Topology based analysis(NTA),其中 ORA 即我们常说的一代的功能富集分析,基于超几何分布的思想,进行感兴趣的基因列表和参考总集合的富集分析,也是今天我们主要操作的内容。
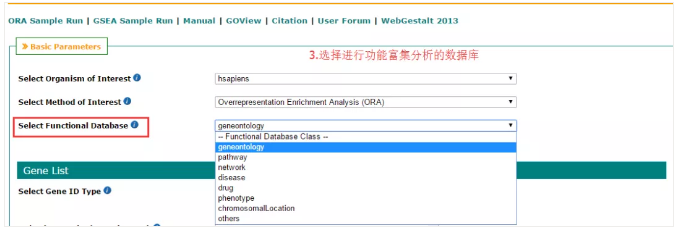
04 选择进行功能富集分析的数据库
在 ORA 的方法下,可以进行 gene ontology 基因本体即 GO 分析,pathway 通路分析包括 KEGG pathway,Wiki pathway, pathway common;network 网络分析包括共表达分析,PPI 分析,micoRNA 靶基因分析,TF 的靶基因分析;disease 疾病的候选基因富集分析包括 DisgeNET 和 PharmGKB 数据库提供的数据;phenotype 表型分析包括 Human Phenotype Ontology 和 Mammalian Phenotype Ontology 的数据;
durg 药物靶标的富集分析等。
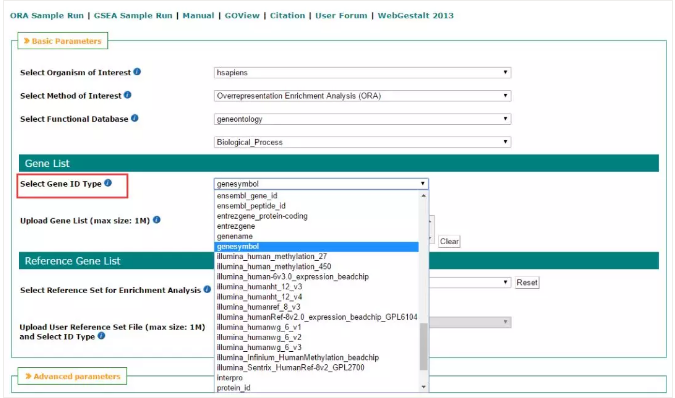
05 输入基因列表
1. 选择上传的基因 ID 类型:
目前 WebGestalt 可以识别 324 中 ID 类型,你想要的都在这里 ^_^
2. 输入 ID
输入方式,可以直接复制粘贴,基因之间以换行符间隔,也可以直接上传文件。
06 选择参考集
根据自己的需求不同,选择上传参考集,或者选择已经有的参考集。
这个时候,你就可以 Submit 啦……
结果是介样的↓,点击 Result Download 即可下载所有的结果啦。

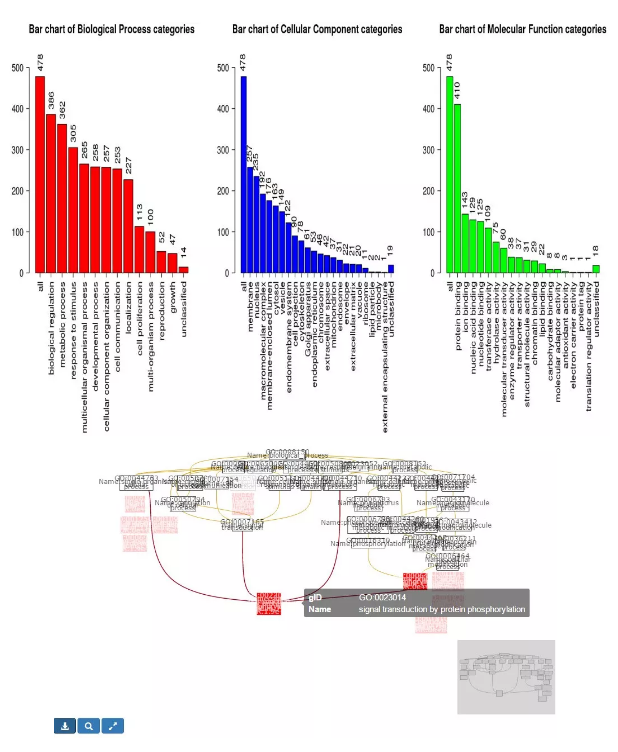
这个鬼畜的模块,其实就是 GO 的有向无环图,可以把 GO term 之间的关系展现处理,现在网站用 Java Script 进行了在线交互,当你把鼠标放在上面的时候,可以看到每个里面的详细参数。
另外啰嗦两句在 advanced parameters 里面的参数设置问题,简单说就是利用统计值进行筛选的参数,这在结果试图也有体现,详解如下:
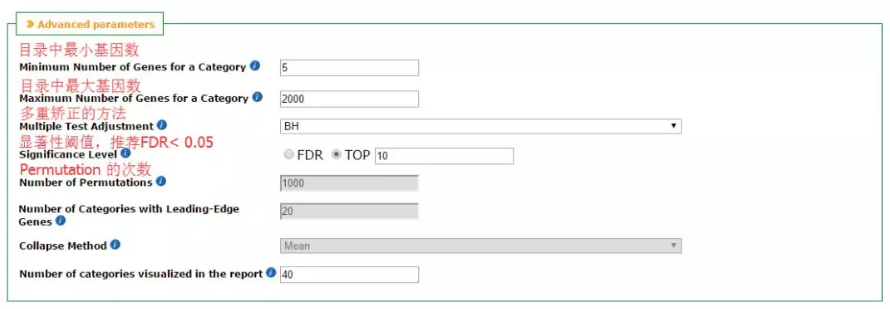
好了,你也快去试一试吧!