轻松学会寻找 crisprcas9 目的基因 CDS 序列
丁香园
最近 CRISPR/Cas9 基因敲除技术可谓风靡全世界的实验室,CRISPR 被称为规律成簇间隔短回文重复,其实际上就是一种基因编辑器,是细菌用以保护自身对抗病毒的一个系统。
后来,研究人员发现,它似乎是一种精确的万能基因武器,可以用来删除、添加、激活或抑制其他生物体的目标基因。由于其相对于前几代基因敲除技术而言更为简便高效。所以一经问世便受到科研界的热捧。利用 Cas9 质粒建立 knock-out 细胞系实验过程主要有以下几步:
1. 确定待敲除基因的靶位点;
2. 设计识别靶位点识别的一对 DNA Oligo(引物);
3. 构建表达 sgRNA 的质粒 ;
4. sgRNA 活性检测;
5. 利用 Cas9 质粒建立 knock-out 细胞系 。
本实验室老师也想跟跟潮流,弄个敲除细胞系出来,于是本小硕就被打发过来进行 CRISPR/Cas9 技术的学习,刚开始是一头雾水,后来在咨询有过分子生物学基础的博士师姐及科室其他老师后,终于是摸到一点门道了。
我们都知道 gRNA 的设计对于 CRISPR/Cas9 基因敲除至关重要,而 gRNA 设计的关键是寻找正确的 CDS 序列。所以在这里首先给大家介绍的是 Cas9 质粒构建的第一个拦路虎,如何寻找目的基因的 CDS 序列。
1. 在 NCBI 的 Gene 数据库中输入目的基因名称(以 p53 为例)
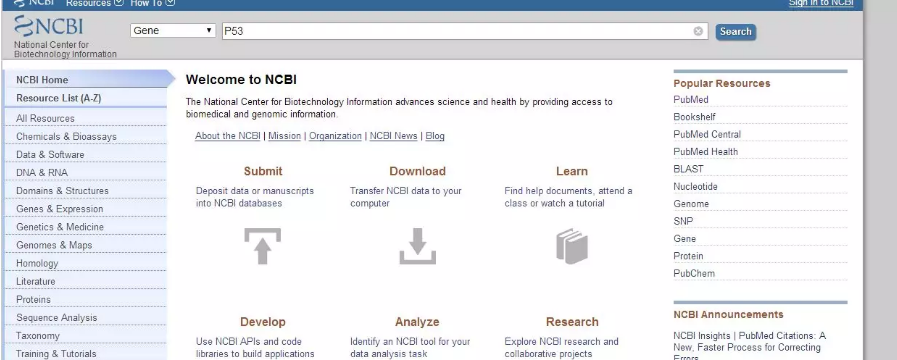
2. 找到对应种属的目的基因,这里选择人源的话是第二个、点击(方框内)
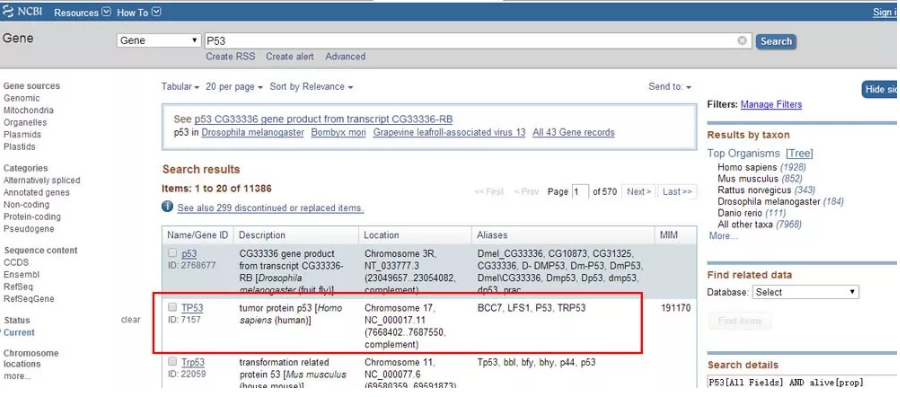
3. 进入后找到 Genebank 选项、点击(往下拉慢慢看)
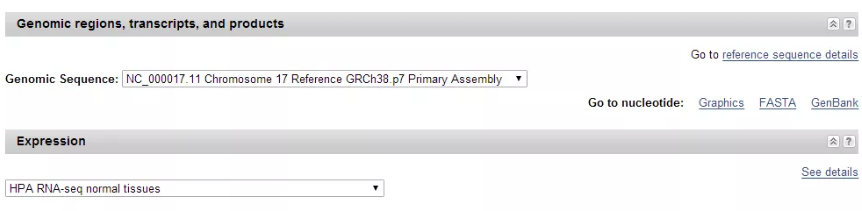
4. 进入后找到 CDS 区中的 CCDS(方框中),点击

5. 找到对应的 CDS 区

6. 将红色以及蓝色的部分间隔的输入麻省理工设计 gRNA 评分网址后根据评分选择分数较高的 3-5 对 gRNA 即可。
这里需要区分解释的几个名称是 mRNA 与 CDS 的区别于联系,只有一部分 mRNA 可以将来翻译为蛋白,这部分叫 CDS(编码序列),就是起始密码子和终止密码子之间那一段。其余的 mRNA 叫 UTR(非翻译区),那么 mRNA 就由 5'UTR,CDS 和 3'UTR 组成。而 CDS 区也包含了内含子与外显子。需要注意的是第 4 点图中红蓝区分的是两段外显子,有可能是跨内含子的。所以网站用不同的颜色将之区分开来。大家设计 gRNA 时也要注意。
好了,这期的 CRISPR/Cas9 基因敲除技术就讲到这里。有兴趣的童鞋请继续关注生物学霸后期关于 CRISPR/Cas9 基因敲除技术的微信文章。