献给初学者:一款超好用的 DNA 序列比对软件
丁香园
对于每一个进入生物领域的人,基本都避免不了分子克隆(认知的同胞除外),当然就需要一个称手的 DNA 比对软件。今天给大家强烈推荐一款 DNA 比对软件,APE(a plasmid editor)。
这款软件不仅能做 DNA 序列比对,DNA 序列翻译,还能够做引物设计,酶切位点设计,质粒图谱构建(这个还是 snapgene 更好用,不过人家是收费的),ORF 查找等非常多的实用功能,下面我来详细介绍一下这几个常用的功能。
先发个软件的链接,需要的小伙伴可以去下载使用☟
http://biologylabs.utah.edu/jorgensen/wayned/ape/
1. DNA 序列比对
在对话框中粘贴序列,工具栏有反向互补的工具,图标为双向箭头,当反向测序时可用。再新建另一个文件,将所需序列粘贴,即可进行比对,如下图。
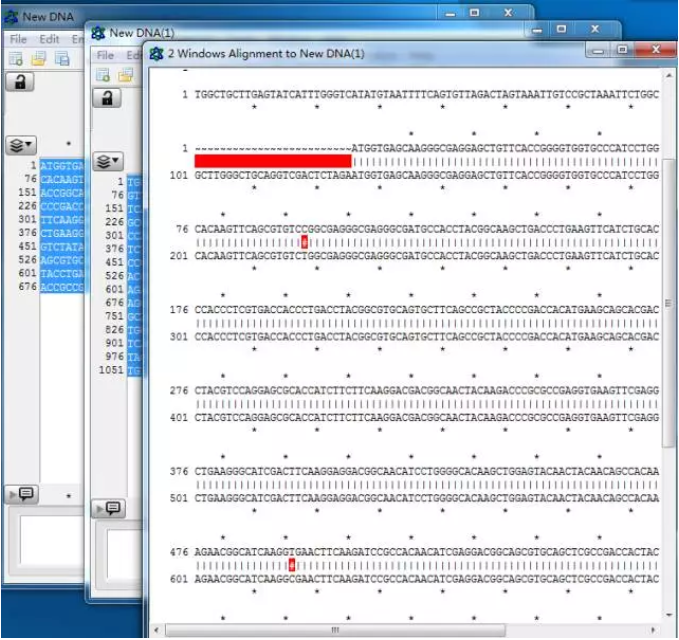
在 Tools 中选择 Align Two Sequence 进行双序列的比对,结果一目了然,mismatch 都被红色标注清楚,双击想看的位置可进入到对话框中,方便找到突变的位置。
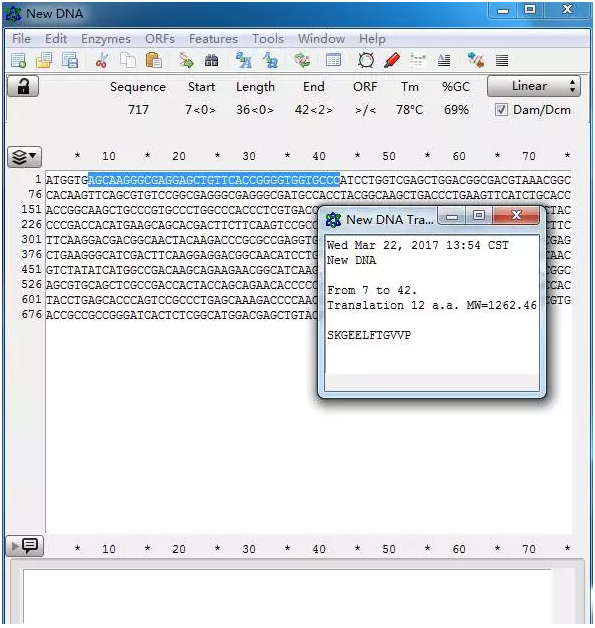
2. 对 DNA 序列进行翻译
选中你想翻译的序列,ctrl+T 即可进行对 DNA 序列翻译成蛋白序列,还可自己进行设计每行显示多长以及蛋白简写的方式。
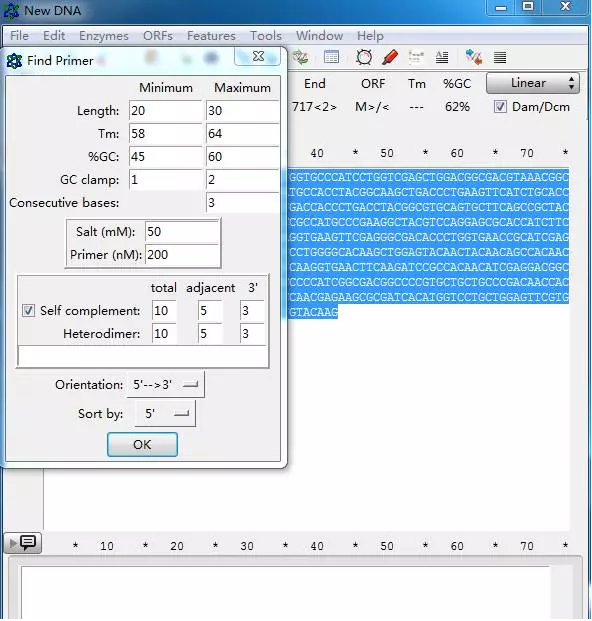
3. 引物设计
输入序列,在 Tools 里找到 find primers,弹出对话框,包含有长度、Tm 值、GC 含量、GC clamp、二级结构等参数可以自行设计。
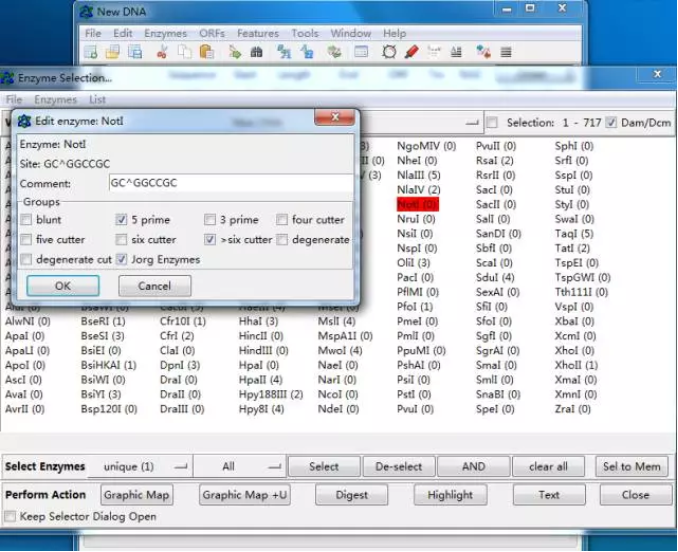
4. 酶切位点设计
在做分子克隆的时候,要将片段插入到质粒中常用的方法就是酶切连接,这时候就要考虑目的片段选用什么酶切位点合适,同样将序列输入后,单击 Enzymes 选择 Enzyme Seletion 或者快捷方式 ctrl+E,即可弹出一个限制性内切酶列表,里面有常用的酶。单击酶可以对需要的内切酶进行标记,双击酶的名称,即可弹出酶的酶切位点信息。

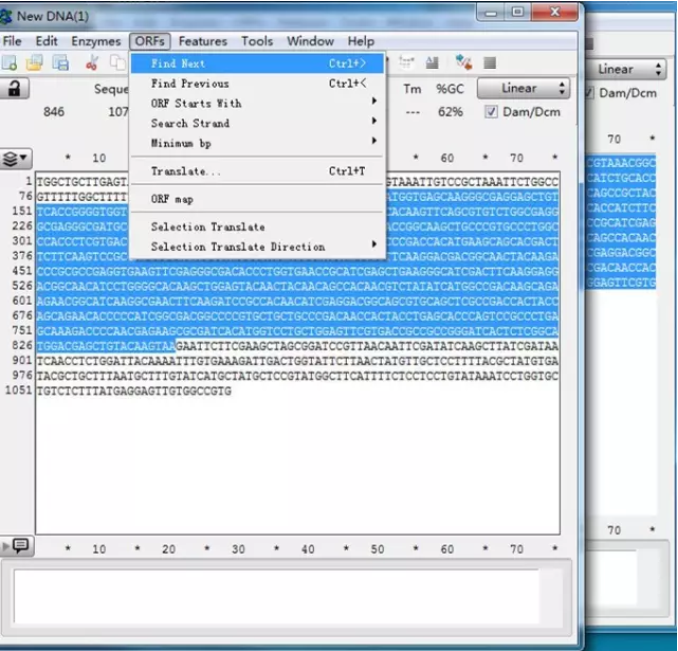
5. ORF 查找
在将鼠标单击序列的任意位置,然后从该位置开始往后 (Find Next) 或往前 (Find Previous)查找 ORF 的序列,单击 Find Next,软件给出查找的结果。
APE 的 Tools 中还有许多功能,如质粒图谱构建、sgRNA 设计、Golden Gate Reaction 等强大的设计功能,满足实验室大部分常规的使用,大家可就自己的需求进行学习。