如何快速得到芯片探针与gene的对应关系,并将探针转换成基因?
ZSCI
现有的基因芯片种类很多,但重要而且常用的芯片并不多,一般分析芯片数据都需要把探针的 ID 切换成基因的 ID。
一般有三种方法可以得到芯片探针与 gene 的对应关系:
① 金标准是去基因芯片的厂商的官网直接下载
② 从 NCBI 里面下载文件来解析
③ 直接用 bioconductor 包。
其中前两种方法都比较麻烦,所以接下来要讲的是:
如何用 R 的 bioconductor 包来批量得到芯片探针与 gene 的对应关系。
一、说明
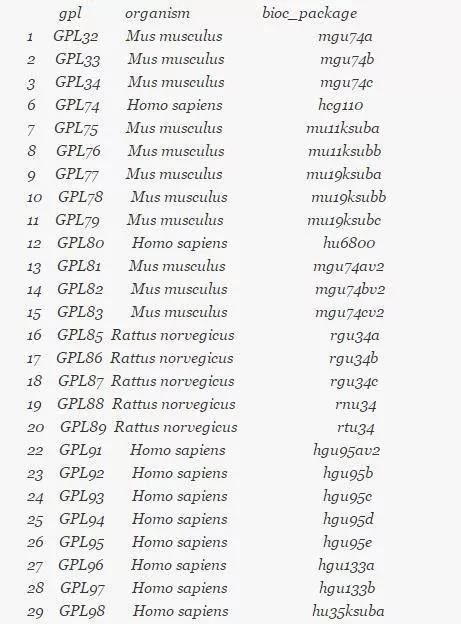
1. 一般重要的芯片在 R 的 bioconductor 里面都是有包的,用一个 R 包可以批量获取有注释信息的芯片平台,选取了常见的物种。
如下:
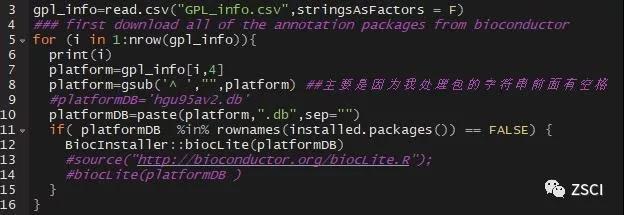
2. 这些包首先需要都下载。
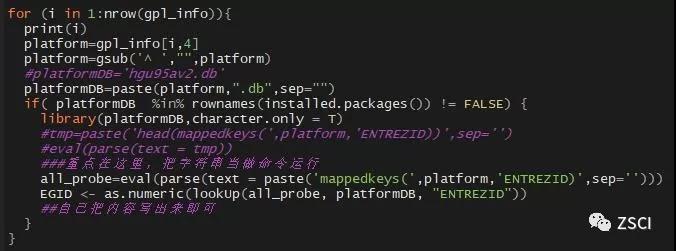
3. 下载完了所有的包, 就可以进行批量导出芯片探针与 gene 的对应关系。
二、 实操
1. 通过 GEO 数据库下载探针矩。
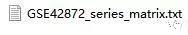
2. 通过探针矩阵查找注释平台信息(GPL6244),根据平台信息在 jimmy 博客中搜索 bioconductor 中包含关系所对应的包(http://www.bio-info-trainee.com/ 1399.html),可知 GPL6244 -->hugene10sttranscriptcluster。
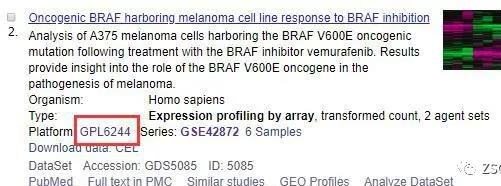
3. 对探针矩阵预处理把基因 id 换成行名。

4. 加载 hugene10sttranscriptcluster 包,并查看里面对象,获取想要的对象。
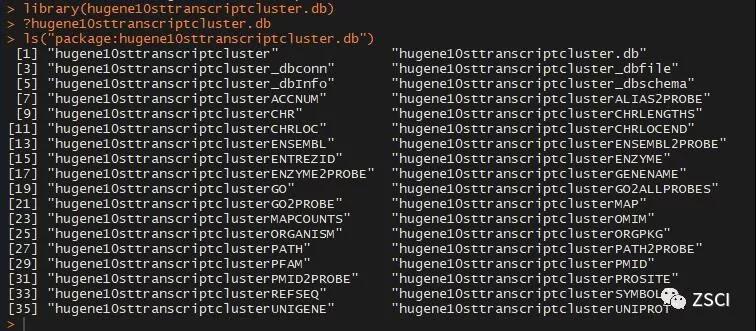
注:要将基因 id 转换成基因名所以选择 hugene10sttranscriptclusterSYMBOL。
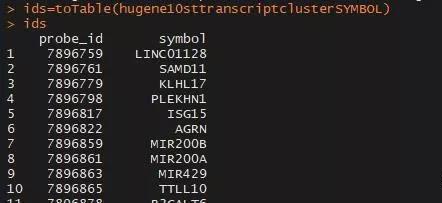
5. 选择「hugene10sttranscriptclusterSYMBOL」对象,获取探针对应基因名。
6. 过滤表达矩阵中基因 id 没有在包中的数据,并将探针 id 改一下顺序,使其与表达矩阵的顺序一致。
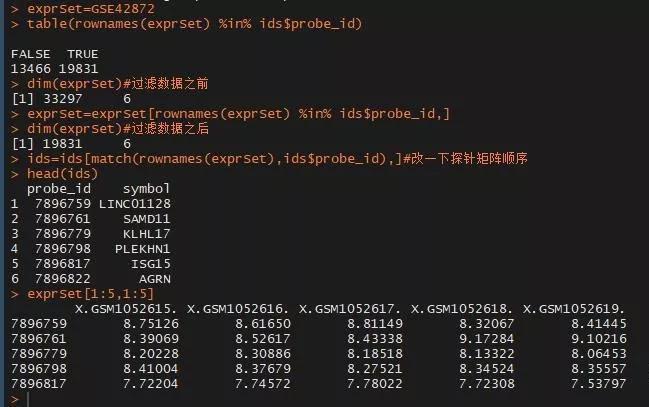
7. 根据包中的探针 id 和基因名的对应关系,将表达矩阵中的 id 转换成基因名,并保存修改文件。






