Pymol 展示氢键
ZeroDesigner
材料
蛋白:1STP
软件:PyMOL (TM) 2.3.0(Edu)
目的
使用 pymol 展示蛋白中的氢键
步骤
1. 蛋白导入
此次选用的蛋白为 1STP 可以直接从 PDB 官网下载文件,或者直接在命令行框中输入fetch 1stp
2. 展示蛋白的 technical 结构
按照步骤,首先点击蛋白右侧的 A 字符
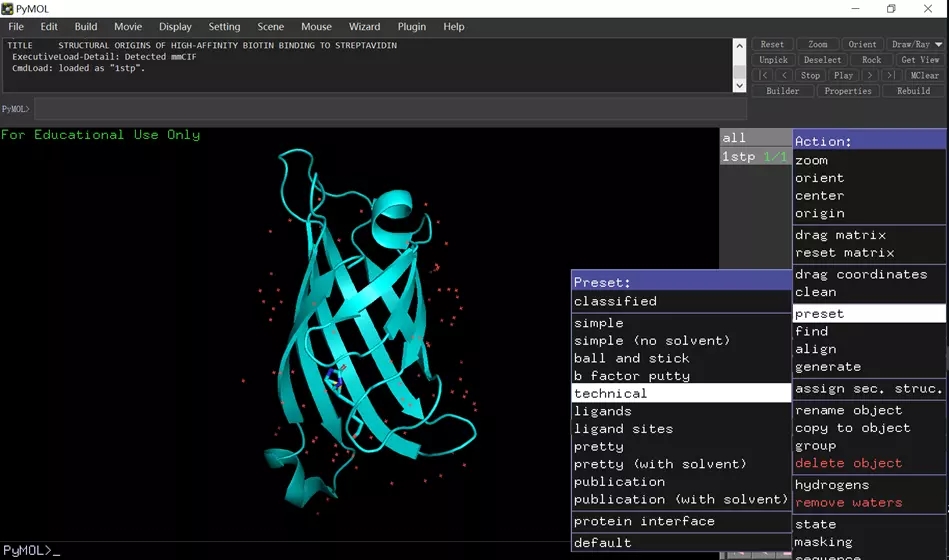
下拉菜单中,点击 preset,然后点击 technical
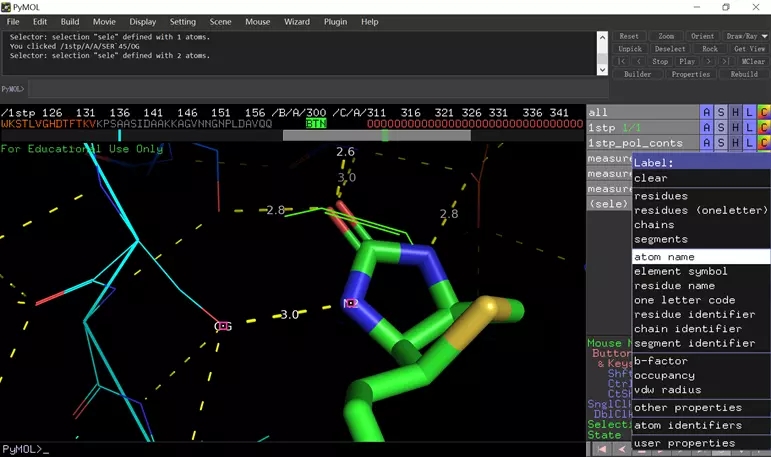
出现图即为氢键相互作用的结构,黄色短线即为氢键,氢键两头分别为供体与受体

3. 查看蛋白的配体氢键
点击右下角字母 S

出现蛋白序列,向右侧拉,红色「0」代表的是水分子。水的前面,蛋白序列的最后会出现蛋白配体。

4. 选中 BTN,点击 sele 右侧的 A,下拉菜单中点击 zoom

5. 按住鼠标左键进行拖动,旋转到合适的角度。

6. 点击 wizard,下拉菜单中选择 measurement

左键选择黄线两端的原子,进行测量,3.0A,氢键距离一般为 3.5A 以内,复合要求,点击 Done。
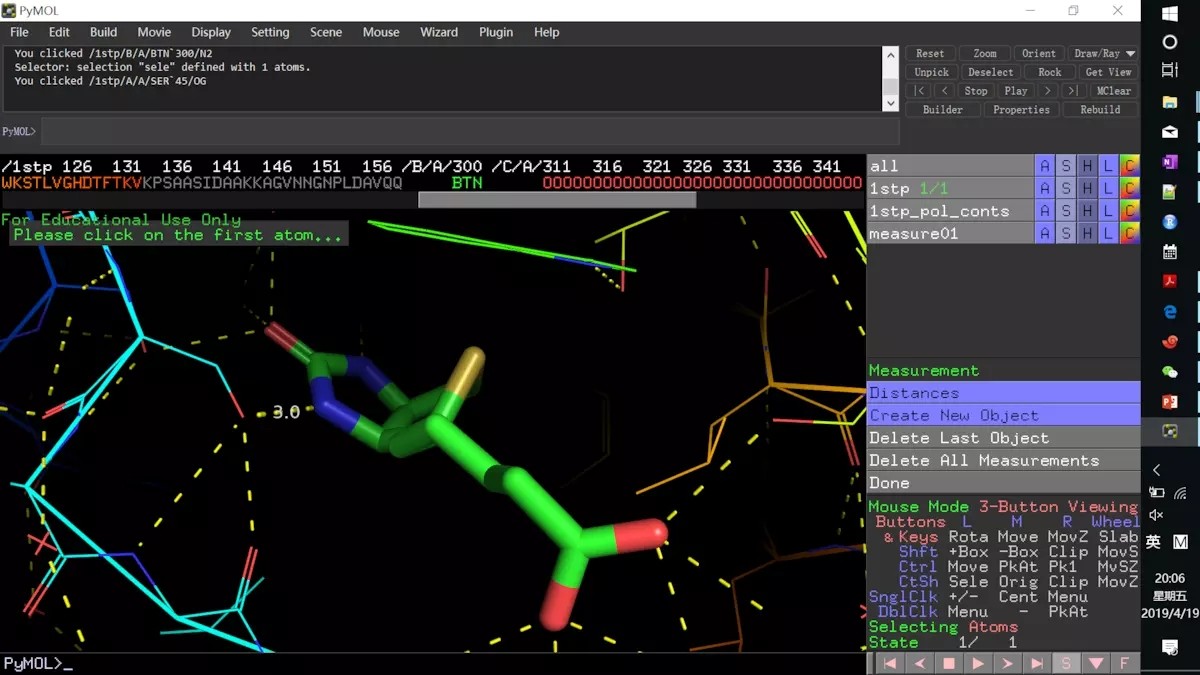
7. 不断点击红框处,直到出现 ATOM。选择测量好的黄线两端的原子。

点击 sele 右端的 label,选择 atom name。此时出现的是两端的名字,OG 为氧原子为氢键供体,N 为氢键受体。