单株 GWAS 与 BSA 结合快速鉴定株高候选 SNP
南京集思慧远生物

GWAS 是定位 QTL 和多种作物性状相关的 SNPs/gene 的重要工具。典型的 GWAS 多用在自然群体中。本研究提出了一种单株 GWAS(sp-GWAS)分析的策略,不需要提供一系列其他不同品种或品系(听起来是不是有些不可思议)。但是 sp-GWAS 依赖于从随机交配群体中取样的单个植物的表型和基因型。更重要的是本文证实了 sp-GWAS 能够有效的和 BSA 策略结合快速确认显著关联的 SNPs。
sp-GWAS+BSA 最大的特点就是群体材料的准备不需要庞大的资源材料,只需要合适的一种即可!是的,你没有看错,只要一种!
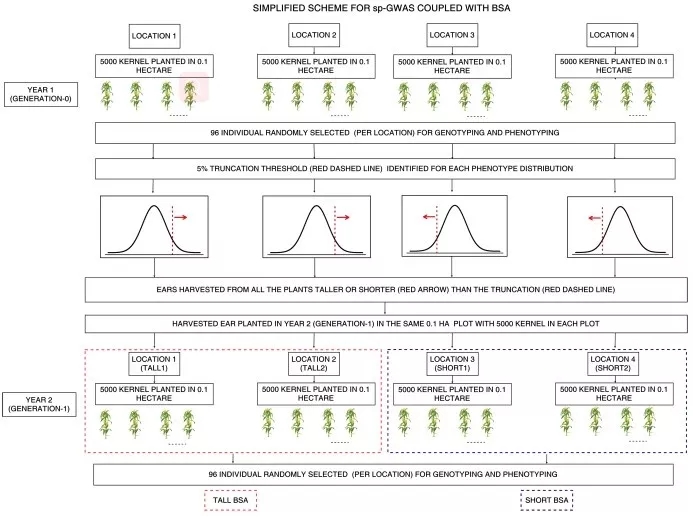
方案概述:
第一年 4 个地方,每个地方分别种植 5000 株 Shoepeg(玉米的一个地方品种),每个地方随机挑选 96 株进行基因型和表型分析。按照表型分别挑选两个地方 5% 极端高和矮的单株。
第二年将 4 个地方分别挑选的极端个体在相同的地方再分别种 5000 株。然后分别从高和矮的两个种植点分别随机各挑选 96 个极端个体进行表型鉴定和 BSA 测序分析。
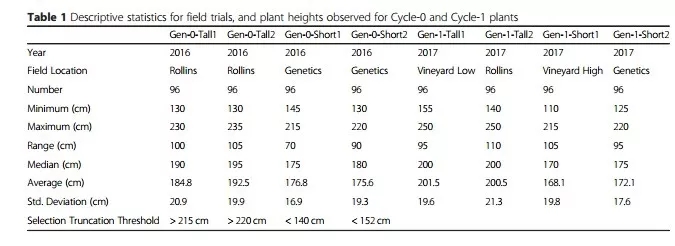
GWAS+BSA 定位玉米株高 QTL
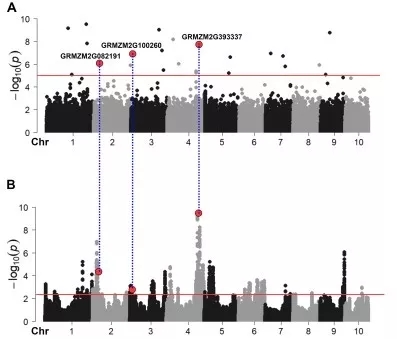
GWAS 和 BSA 对玉米株高定位结果(其中 GWAS 显著相关位点有 25 个,其中 3 个与 BSA 关联区域重叠)
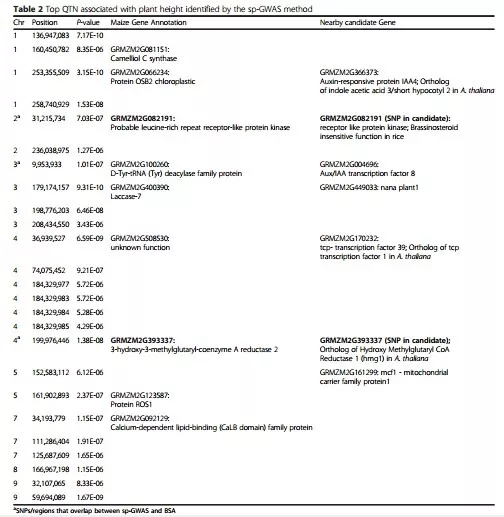
GWAS 关联位点注释信息
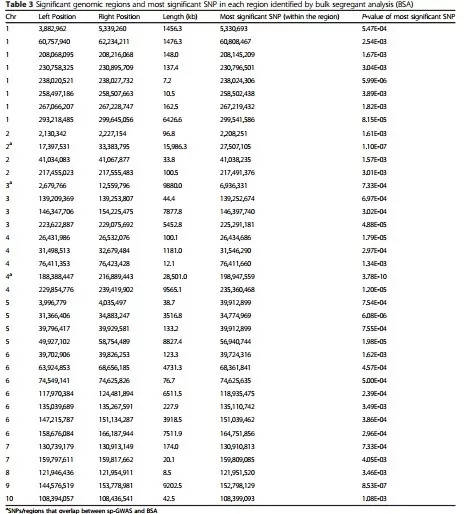
GWAS 关联区域及注释信息
主要结论
本研究用到 shoepeg 玉米地方品种(一种作为开放授粉的品种),用来评估 sp-GWAS 与 BSA 联用是否能高效的检测到与株高显著关联的 SNPs。共有 768 个单株种植在两年的 8 个不同环境下,sp-GWAS 分析。在 768 个单株中共有 306k 个多态标记用于关联分析,最终获得 25 个与株高显著相关的 SNPs(p<0.00001)。相同群体分别挑选高和矮的个体进行混池测序,BSA 关联到 37 个与主要相关的区域。25 个 GWAS 关联的位点中有 3 个是和 BSA 定位区域是重叠的。







