这款建树速度超快的工具,盘它!
随着基因组数目的增加,挑选「同源基因」和「序列联配计算」的难度都在增加。如何更加充分地利用全基因组信息并且使分类变得更加准确一直困扰着许多科研工作者。
今天介绍的这款工具,是由复旦大学基于组分矢量构树法开发的一款软件,组分矢量构树法(Composition vector tree,简称 CVTree)。
CVTree 的特点
首先统计基因组中特定长度短串组,为每个物种构造了一个「高维代表矢量」,然后用矢量之间的余角余弦计算物种间的遗传距离,最后使用邻接法进行构树。
使用起来方便简单,不需要自己来挑选同源基因,也不进行序列比对,所以从根本上避开了人为干预对结果可能产生的影响.
具有比传统方法更高的分辨能力,为了更加方便用户使用以及适应基因组数据的增加,该研究组又开发并更新了现如今的 CVTree3 网络服务器,让我们一起去探索吧。
如何建树?
1. 首先打开网站(http://tlife.fudan.edu.cn/cvtree/cvtree/)。单击「Load-Create Project」,新建自己的项目,然后选择和设置「基本参数」。
CVTree3 为大家提供了 338 种古菌、2850 种细菌以及 8 个真核生物作为参比基因组备选,大家可以选择类别范围,也可以点击「See Detail」进行逐条选择。
同时可以上传分析自己的(多个)基因组,注意上传时序列文件的扩展名要改成 .ffn(核苷酸)或 .faa(蛋白质)。
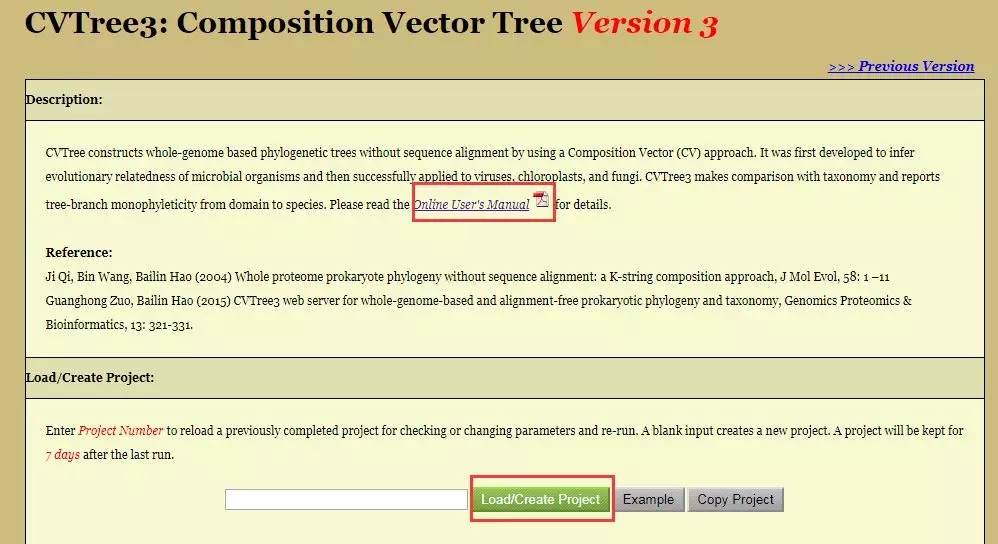

2. 上传好了之后检查一下参数,也可以留下自己的邮箱,项目完成后会发送邮件通知你。点击右上角的「All parameters are fine, Run Project」,服务器就可以开始计算了。
不过需要注意,每个项目完成后只在服务器保存 7 天,所以对于结果及时进行下载和保存。
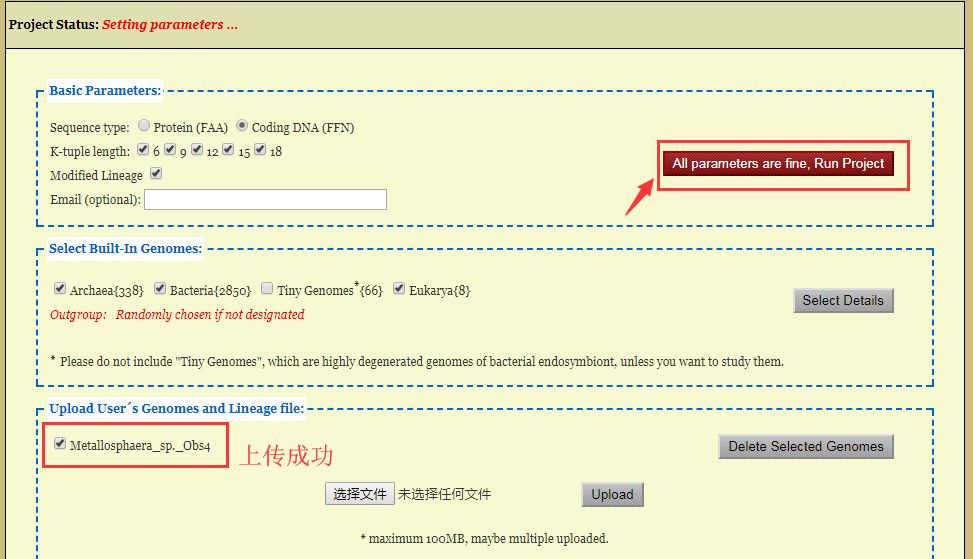
3. 计算结束后,点击右上角出现的「See Result」,进入结果页面。
首先该页面展示的是一个按照菌株的分类阶梯显示的亲缘关系与分类系统的对比结果,依靠的关键概念是「单源枝」,即分类单元内所包含的菌株对应于该分支下的所有枝叶所代表的菌株。
当某个枝为单源时,则说明亲缘关系与分类系统对于当前的数据集合是一致的。单源枝的数目按分类级别,统计在后面的「Monophyly」与「None」里,「Unclassified」中列出了一些分类关系不确定的菌株。
除此之外,CVTree 还有一大亮点,就是自动建立进化树,并可以在线进行修改美化,这个功能非常方便强大。在结果页点击右上角的「See Tree」就可以看到这棵进化树了。
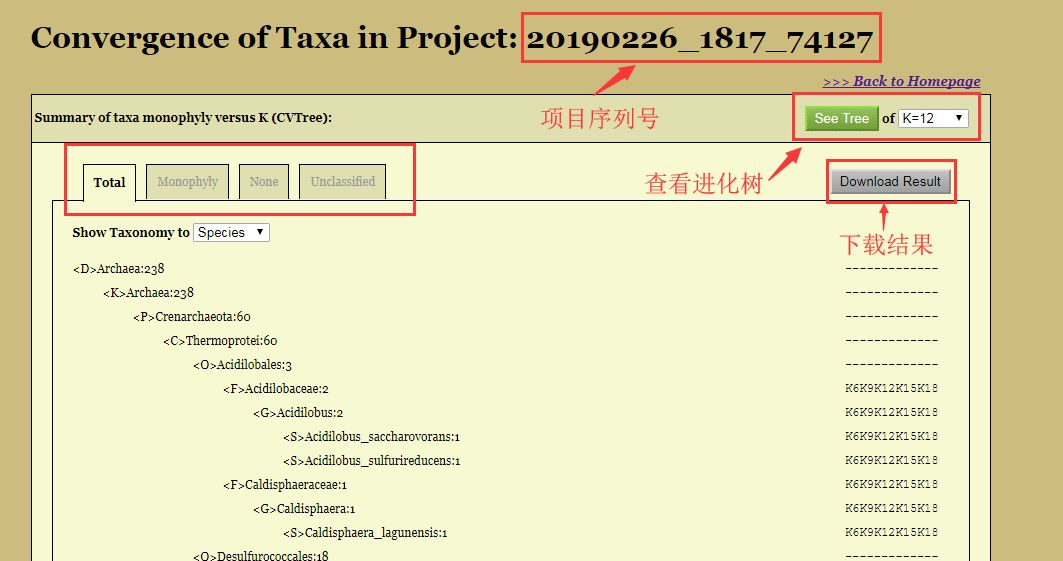
如何美化进化树?
1. 在网页中,这是一棵动态的进化树,每个节点都可以展开或者收缩。
也可以根据自己的需要设置「K 值」,选择展示自己上传的序列,调整好之后点击「Output」,下一步出现的界面还可以对字体以及间距等进行美化和调整。
不过展开和收缩要在这一步进行之后再进行导出,下一步无法调整序列的展开和收缩。
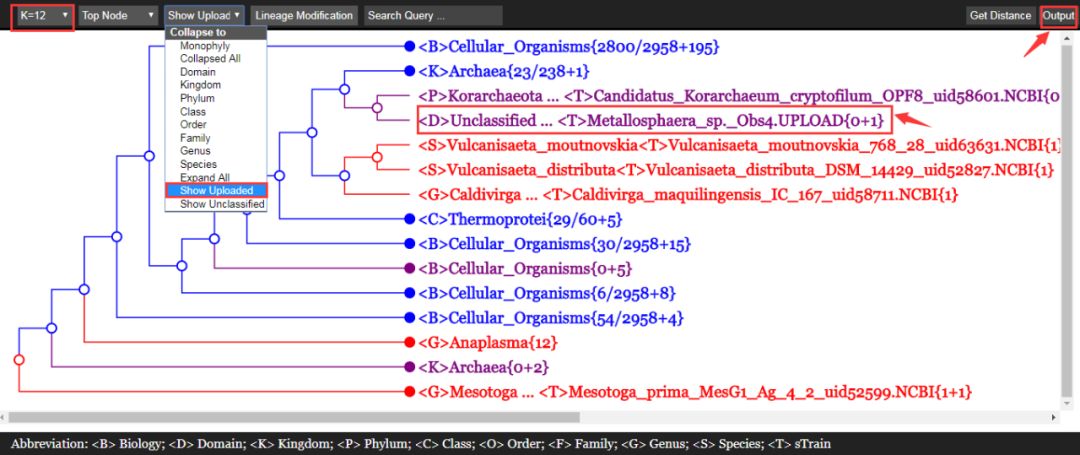
2. 点击之后可以对里面的信息进行修改,枝干和字体都可以变成自己喜欢的颜色和样式。
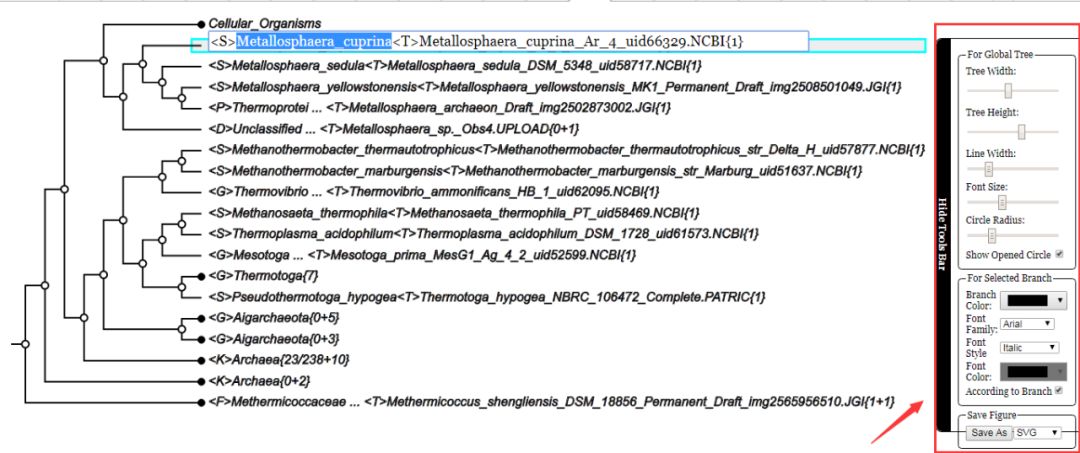
3. 最后设置自己需要的格式导出图片。一棵美丽的进化树就建好了!
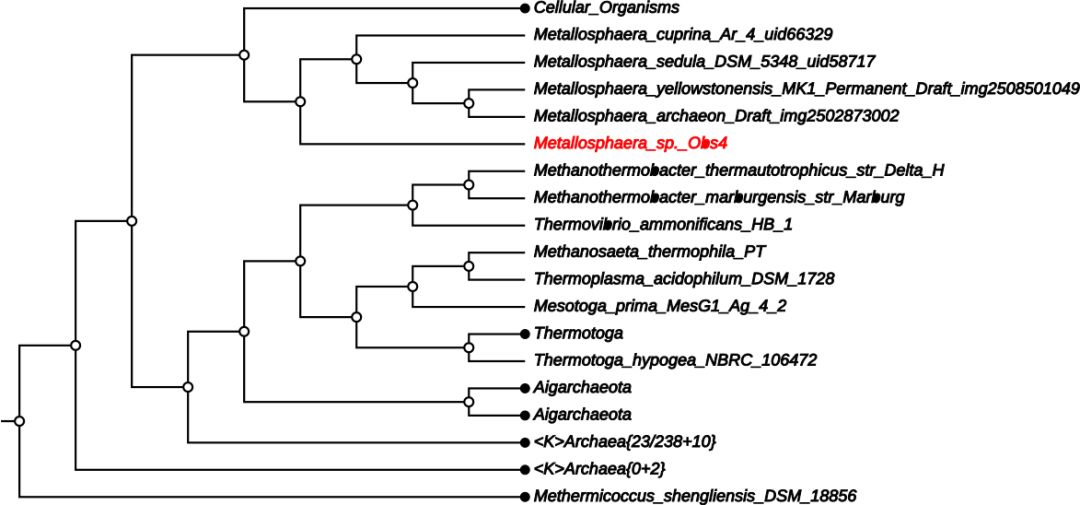
CVTree3 的使用非常简单,无需下载安装,页面很友好,并且充分利用了全基因组的信息,尤其是对于一些需要高要求分类标准的菌株是非常好的一个工具。
最后祝愿大家都能「画漂亮图,发高分文」!
参考文献:
[1] Zuo G , Hao B . CVTree3 Web Server for Whole-genome-based and Alignment-free Prokaryotic Phylogeny and Taxonomy[J]. Genomics, Proteomics & Bioinformatics, 2015, 13(5):321 - 331.
[2] 左光宏, 郝柏林. 基于全基因组的微生物亲缘关系与分类系统研究工具——CVTree[J]. 生物技术通报, 2015, 31(11):60 - 67.
图片来源:作者提供




