这款免费的网页版工具,轻松搞定你的 qPCR 数据统计分析
众所周知,qPCR 从来就不是让人省心的实验。小心翼翼的提 RNA,宝贝一样的呵护起来;兢兢业业的摸索最佳温度梯度,生怕引物探针一言不合就「罢工」;
好不容易跋山涉水来到最后一关,拿到了一大堆 Cq 值,可大神级的 SPSS、R 语言听起来就头大,更惶论轻松掌握了,不擅长统计分析的你是不是欲哭无泪?
终于,福音出现了 —— SATQPCR 1
全称:short for Statistical Analysis Tool for Quantitative PCR Data
它是免费的网页版工具,无需安装软件,简单易用;完全遵循 MIQE 指南设计2(扩增效率、内参基因的选择等);支持图片生成、t 检验和方差分析(事后检验采用 Tukey Test)。
闲话少说,直接上干货。
图文教程
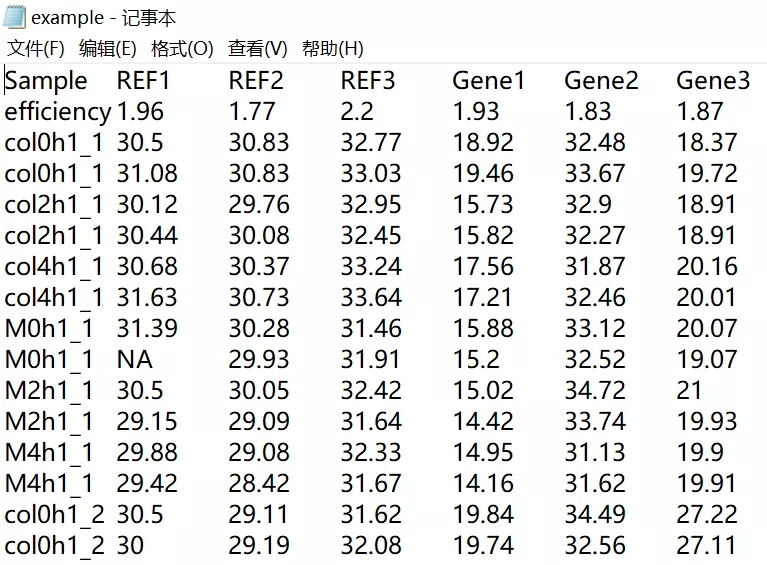
1. 比如要研究处理后小鼠的三个靶标基因(Gene1、Gene2、Gene3)随时间(0 h、2 h、4 h)的表达水平变化,有三个基因(Ref1、Ref2、Ref3)作为参照基因。
设置 Control 组(col)和处理组(M),有两个技术学重复(Sample Name 完全相同)和三个生物学重复(即 Sample Name 的后缀,小鼠 1、2 和 3)。
按照下图所示导出 Cq 值到 Excel 或者 txt,其中第二行为扩增效率,没有数值用 NA 表示。
2. 进入 SATQPCR 网页,点击「SATQPCR Tool」按钮即可到达数据上传页面。点击「选择文件」按钮将 example 数据文件上传。
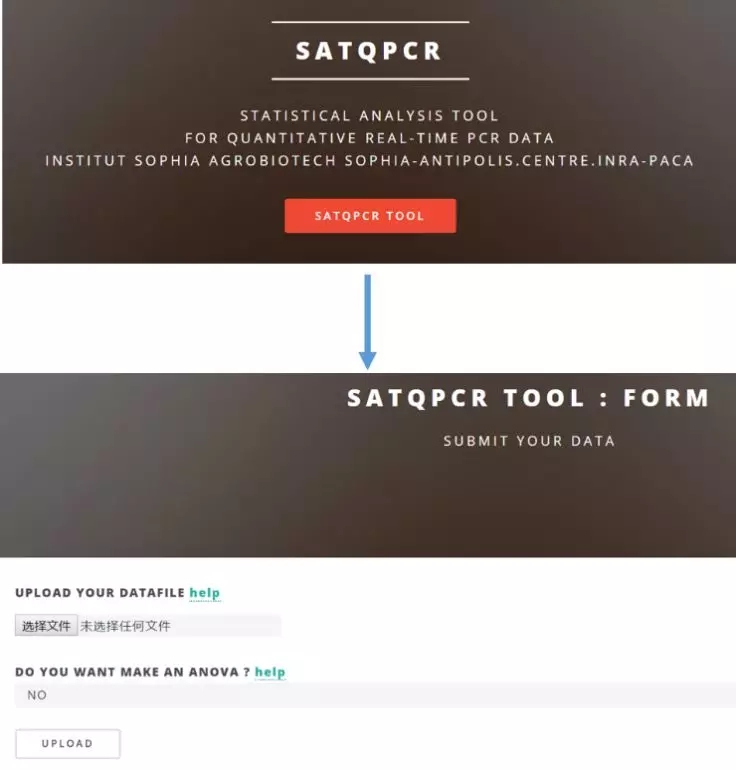
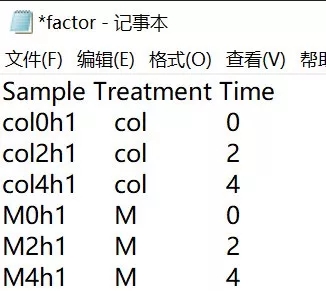
3. 如果要做 ANOVA 分析,则需要选择 Yes 选项,同时上传 Factor 文件。
该文件含有变量的数量等信息,如下图所示,该实验包含处理因素和时间两个考察维度,处理因素有两个变量(处理 M 和不处理 col),时间有三个变量。上传后点击 upload。
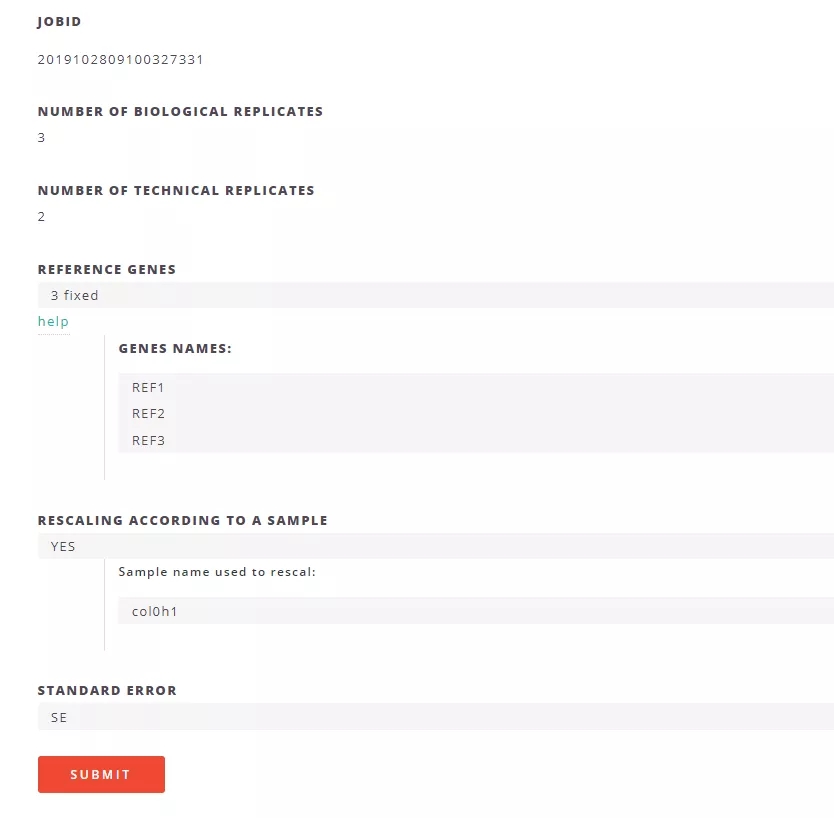
4. 数据上传成功后会自动识别重复的数量。
内参基因可以由用户自己指定(fixed)或者由程序根据算法3自动寻找最稳定表达的基因(calculated),数量可以选择 2 个或者 3 个。
同时可以选择以某一个样本(如 col0 h1)为 control 来建图,误差线可选择标准误 SE 或者标准差 SD。点击 Submit 提交。
5. 数据文件中包括表达量数据、柱状图和统计检验三部分,可以直接点击下载压缩的结果文件。
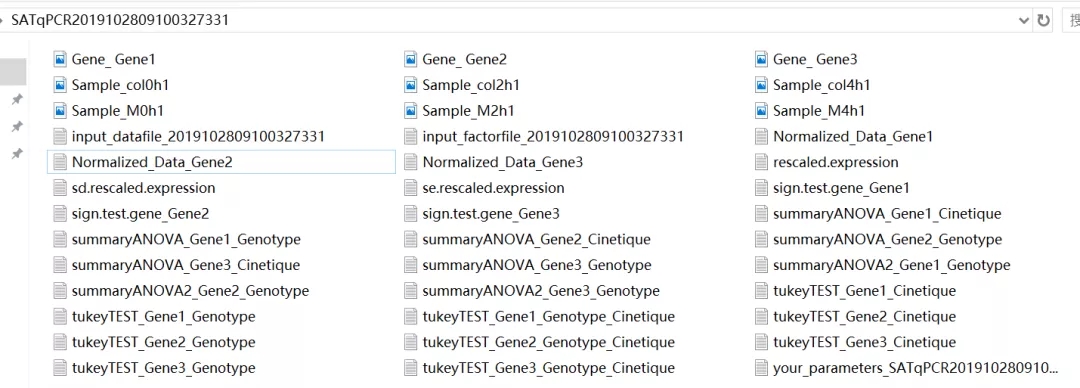
6. Normalize_Data 文件中包含每个靶标基因的均一化表达量。
Gene_柱状图展示了不同的 sample 中特定靶标的表达量(以 col0 h1 为 control),Sample_柱状图展示了同一 sample 中不同靶标的表达量。

7. Sign.test 文件展示了特定靶标在不同样本中表达量的 t-test 检验的 p 值矩阵,下图以 Gene1 为例,可以看到 col0 h1 样本与其他样本均有极显著的统计学差异(p<0.01)。
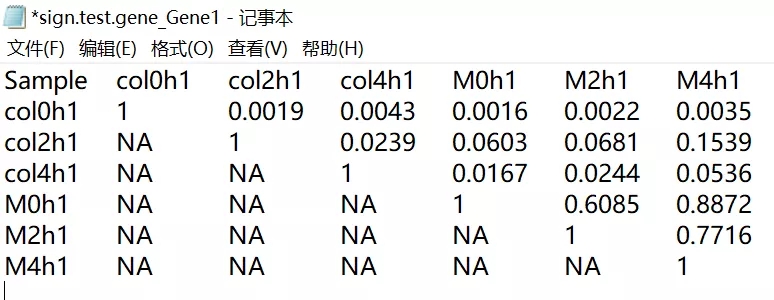
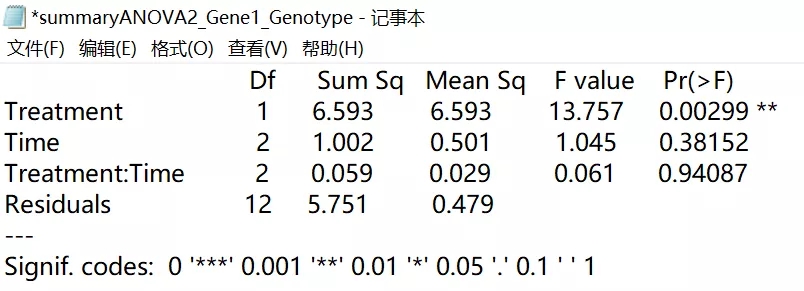
8. summaryANOVA 及 summaryANOVA2 分别展示了单因素和双因素的 ANOVA 检验结果。
该实验中我们需要参考双因素的检验结果,可以看到 Treatment 因素具有极显著的统计学差异(双星号表示,最后一行有标注说明),这样需要对 Treatment 因素进行 Tukey Test。
9. tukeyTEST 给出了不同基因的检验结果,包括差值(diff)、95% family-wise 置信区间(lwr 和 upr)及 p 值。

是不是够 easy?
简化流程而又不出错是我们做数据分析的目标,这个网站都帮我们想到了。
可能很多人对最后的统计分析的解读无所适从,接下来我会推送一篇详细的关于 qPCR 数据统计分析的文章,对其中可能犯的错误一一列出,希望能够帮到大家。
工具网址:
http://satqpcr.sophia.inra.fr/cgi/home.cgi
参考文献:
1. Rancurel, Corinne, et al. "SATQPCR: Website for statistical analysis of real-time quantitative PCR data." Molecular and cellular probes 46 (2019): 101418.
2. Bustin, Stephen A., et al. "The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments." Clinical chemistry 55.4 (2009): 611 - 622.
3. Vandesompele, Jo, et al. "Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes." Genome biology 3.7 (2002): research0034 - 1.
图片来源:作者提供