microRNA实验方法小结
互联网
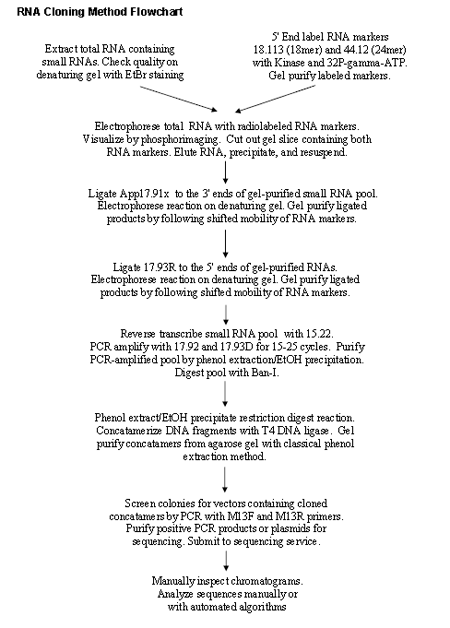
Stem-loop实时定量RT PCR
传统实时定量RT PCR只能检测到miRNA前体,而Stem-loop实时定量RT PCR技术可以解决这一问题。
Stem loop实时定量RT PCR是一项高特异度、敏感度的检测miRNA表达的实验技术,包括设计具有茎环(stem loop)结构的反转录引物和用miRNA荧光标记的特异分子探针进行实时PCR 二个关键步骤。
该技术具有以下优点:高度特异性,对序列高度同源的miRNA也可精确区分;超宽的定量线性范围和高度的检测灵敏度;样品消耗少,仅需 1~10 ng的总RNA;适用范围广,总RNA 、细胞裂解物以及纯化的RNA都可用于miRNA的定量检测。
Stem- loop实时定量RT PCR基本原理: microRNA的RT-PCR一般使用茎环结构的引物,这种具有茎环结构的引物针对所需检测的目的microRNA设计,具有特异的序列,结构示意图如 下所示。内参使用的是小RNA U6,U6的反转录使用的是随机引物。将反转录的产物作为real-time PCR的模板,检测目的microRNA的引物是针对目的microRNA的特异的上下游引物,检测内参使用的是针对U6的特异的上下游引物。
实验过程
用Trizol抽提total RNA,抽提方法同普通的RNA的Trizol抽提,只是在用异丙醇处理时需要4 ℃ 过夜。将抽提得到的RNA置于-80 ℃保存。
将抽提的RNA进行反转录。反转录的体系一般使用12.5 ul的体系。具体的体系如下:
RNase free water
Total RNA: 0.625 ug
Impron buffer: 2.5 ul
dNTPs: 2.5 ul
RNase inhibitor: 0.625 ul
Impron Mgcl2: 1.5 ul
Primer: 0.5 ul
Reverse transcriptase: 0.625 ul
具 体的操作是事先计算好需要的模板量,以及所要的RNase free water的量,在PCR小管中加好需要的模板量以及RNase free water的量,并将剩下的模板立即置于-80 ℃保存。再按照反转录的体系做好总的MIX,再分装到每个PCR小管内。将PCR小管置于PCR仪上反转,反转的具体程序如下: 25 ℃ 5 min ,42 ℃ 60 min ,70 ℃ 15 min. 4 ℃储藏。
反转录的cDNA作为real-time PCR检测的模板,由于real-time PCR检测灵敏性,要求每个样需要三个重复。每一个反应的用量如下:
SYBR 5.0 ul
Primer mix 0.2 ul
cDNA 1.0 ul
water 3.8 ul
在八连管中先加入模板,加入3.4 ul的cDNA模板。
根据需要检测样本的个数,计算所需要的SYBR、Primer mix、water量,具体的计算过程为:试剂单反应量X检测样本个数X3.4 ,根据计算需要的量制作MIX 。将MIX加入每个八连管的小管中。
将对应的每个八连管的小管取出10ul加入96孔板,每个小管对应96孔板的3个孔,即3个重复孔。
加完样品后,用专用的封膜覆盖96孔板,用专用的刮板覆盖严实。
上机检测。具体PCR程序如下:
50 ℃ 2 min ;
95 ℃ 5 min ;
95 ℃ 30 s ;
60 ℃ 40 s ;
72 ℃ 30 s ;
95 ℃ 15 s ; 60 ℃ 30s ;95 ℃ 15 s .
实验注意事项
由于real-time PCR的检测十分灵敏,因此它对RNA定量的准确性要求较高,一般RNA的定量多采用3复孔定量。
无论在反转录或real-time PCR加样时要求加样一定要准确。
在将八连管的mix加入96孔板前一定要充分混匀,一般采取的策略是将八连管一剪为二,置于Vortex上振荡混匀,Vortex的转速不要太高,以免将管内的液体溅到盖子内。
miRNA靶基因的寻找
miRNA 的靶基因的寻找主要通过利用生物信息学方法和生物学实验方法。将生物信息学与生物实验相结合来寻找靶基因可以很大提高准确率。 生物信息学方法主要是利用某种算法对靶基因样本进行评分及筛选。虽然预测方法各有不同, 但由于miRNA和靶基因间的作用具有一定规律性:
miRNA与其靶位点的互补性;
miRNA靶位点在不同物种之间的保守性;
miRNA-mRNA双链之间的热稳定性;
miRNA靶位点处不应有复杂二级结构;
miRNA 5 端与靶基因的结合能力强于3 端.
除此以外,不同的预测方法还会根据各自总结的规律对算法进行不同的限制与优化。为了得到更为可靠的靶基因预测,通常综合多个预测方法,取其共同预测的基因作为研究重点。目前常用的预测方法主要是以下三种:
miRanda.
miRanda 是最早的一个利用生物信息学对miRNA靶基因进行预测的软件, 由Enright等人于2003年设计开发. 作为最早的miRNA靶基因预测软件, miRanda对3-UTR的筛选依据主要是从序列匹配、miRNA与mRNA双链的热稳定性以及靶位点的保守性三个方面进行分析。基于以上三个方 面,miRanda选取每条miRNA相对的3-UTR中排名前10位的基因, 作为miRNA的候选靶基因, 对于多个miRNA对应于同一靶位点的情况, miRanda则使用贪心算法选取其中得分最高且自由能最低的那一对。
TargetScan.
TargetScan 是Lewis等在2003年开发的一款用于预测哺乳动物miRNA靶基因的软件, 该软件将RNA间相互作用的热力学模型与序列比对分析相结合, 预测不同物种间保守的miRNA结合位点. 与miRanda不同,TargetScan提出了“miRNA种子区”的概念。“miRNA种子区”是指miRNA5 端第2-8位碱基与mRNA的3-UTR完全互补所在序列。此外,它也引用了信噪比来评估预测结果的准确度. 所谓信号噪声比即用已知(信号组)和随机生成的miRNA(噪声组)分别对mRNA的3?UTR进行预测, 所得靶基因数目的比值.
PicTar.
PicTarKrek 等人在2005年开发的一款名叫组合靶位点的概率识别(probabilistic identification of combinations of target sites)的预测软件.它采用一种更先进的算法来预测脊椎动物、线虫和果蝇中miRNA的靶基因, 并通过实验方法进行了验证。与TargetScan一样, PicTar也强调“种子区”在靶位点识别及在转录后调控中的关键作用, 也注重miRNA和靶基因结合的自由能在靶基因翻译抑制中的关键作用。
不同的是PicTar把种子序列分为“完全匹配种子序列”和“不完全匹配种子序 列”,前者要求种子序列和靶基因完全互补配对; 后者在满足miRNA与靶基因结合自由能不增加的前提下允许种子序列出现错配, 但不允许G U 配对。
同时结合以前实验数据对两类种子序列对应的 miRNA和靶基因结合自由能进行了限制, 要求“完全 匹配种子序列”的miRNA 与靶基因结合自由能小于miRNA和靶基因最优结合能的33%; 而“不完全匹配种子序列”的miRNA与靶基因的结合自由能要求小于miRNA和靶基因最优结合能的66%, 这有效地降低了假阳性率.
miRNA靶基因的鉴定
由 于计算机模拟在预测miRNA靶基因时存在一定的局限性,利用生物学实验方法可以更加直观地寻找miRNA靶基因.目前主要是从mRNA水平与蛋白质水平 来寻找靶基因。从mRAN水平来寻找靶基因主要是通来在细胞内过表达miRNA后,利用基因芯片分析mRNA的变化以找出相应miRNA的靶基因。
这种方法由于miRNA所介导的转录后翻译抑制过程不引起mRNA水平的改变, 因此依据mRNA水平的变化寻找miRNA靶基因的方法在检出率上存在一定问题. 故采用蛋白质质谱的方法可以有效弥补这个不足.同时再结合miRNA靶基因的预测方法,可以大大提高了靶基因的检出率及可靠性。
目前鉴定靶基因最直接的方法是, 利用荧光定量PCR及Western blot方法分别检测转染或敲低miRNA后细胞中mRNA水平及蛋白水平的变化, 从而确定miRNA与靶基因的对应关系.
这种方法可以大大提高准确率,但最终确定靶基因,还需要鉴定miRNA的靶位点. 而miRNA的靶位点的鉴定最常用的方法是荧光素酶报告基因法. 其基本原理是首先构建荧光素酶表达载体, 将希望鉴定的miRNA靶基因的3 UTR构建到荧光素酶基因的3 UTR中, 然后将荧光素酶基因表达载体转染细胞并改变细胞中相应miRNA的表达水平, 最后检测荧光素酶的表达情况以分析转染3 UTR中是否含有miRNA的靶位点。
Stem-loop实时定量RT PCR
传 统实时定量RT PCR只能检测到miRNA前体,而Stem-loop实时定量RT PCR技术可以解决这一问题。Stem loop实时定量RT PCR是一项高特异度、敏感度的检测miRNA表达的实验技术,包括设计具有茎环(stem loop)结构的反转录引物和用miRNA荧光标记的特异分子探针进行实时PCR 二个关键步骤。该技术具有以下优点:高度特异性,对序列高度同源的miRNA也可精确区分;超宽的定量线性范围和高度的检测灵敏度;样品消耗少,仅需 1~10 ng的总RNA;适用范围广,总RNA 、细胞裂解物以及纯化的RNA都可用于miRNA的定量检测。
Stem- loop实时定量RT PCR基本原理: microRNA的RT-PCR一般使用茎环结构的引物,这种具有茎环结构的引物针对所需检测的目的microRNA设计,具有特异的序列,结构示意图如 下所示。内参使用的是小RNA U6,U6的反转录使用的是随机引物。将反转录的产物作为real-time PCR的模板,检测目的microRNA的引物是针对目的microRNA的特异的上下游引物,检测内参使用的是针对U6的特异的上下游引物。
实验过程
用Trizol抽提total RNA,抽提方法同普通的RNA的Trizol抽提,只是在用异丙醇处理时需要4 ℃ 过夜。将抽提得到的RNA置于-80 ℃保存。
将抽提的RNA进行反转录。反转录的体系一般使用12.5 ul的体系。具体的体系如下:
RNase free water
Total RNA: 0.625 ug
Impron buffer: 2.5 ul
dNTPs: 2.5 ul
RNase inhibitor: 0.625 ul
Impron Mgcl2: 1.5 ul
Primer: 0.5 ul
Reverse transcriptase: 0.625 ul
具 体的操作是事先计算好需要的模板量,以及所要的RNase free water的量,在PCR小管中加好需要的模板量以及RNase free water的量,并将剩下的模板立即置于-80 ℃保存。再按照反转录的体系做好总的MIX,再分装到每个PCR小管内。将PCR小管置于PCR仪上反转,反转的具体程序如下: 25 ℃ 5 min ,42 ℃ 60 min ,70 ℃ 15 min. 4 ℃储藏。
反转录的cDNA作为real-time PCR检测的模板,由于real-time PCR检测灵敏性,要求每个样需要三个重复。每一个反应的用量如下:
SYBR 5.0 ul
Primer mix 0.2 ul
cDNA 1.0 ul
water 3.8 ul
在八连管中先加入模板,加入3.4 ul的cDNA模板。
根据需要检测样本的个数,计算所需要的SYBR、Primer mix、water量,具体的计算过程为:试剂单反应量X检测样本个数X3.4 ,根据计算需要的量制作MIX 。将MIX加入每个八连管的小管中。
将对应的每个八连管的小管取出10ul加入96孔板,每个小管对应96孔板的3个孔,即3个重复孔。
加完样品后,用专用的封膜覆盖96孔板,用专用的刮板覆盖严实。
上机检测。具体PCR程序如下:
50 ℃ 2 min ;
95 ℃ 5 min ;
95 ℃ 30 s ;
60 ℃ 40 s ;
72 ℃ 30 s ;
95 ℃ 15 s ; 60 ℃ 30s ;95 ℃ 15 s .
实验注意事项
由于real-time PCR的检测十分灵敏,因此它对RNA定量的准确性要求较高,一般RNA的定量多采用3复孔定量。
无论在反转录或real-time PCR加样时要求加样一定要准确。
在将八连管的mix加入96孔板前一定要充分混匀,一般采取的策略是将八连管一剪为二,置于Vortex上振荡混匀,Vortex的转速不要太高,以免将管内的液体溅到盖子内。
miRNA靶基因的寻找
miRNA 的靶基因的寻找主要通过利用生物信息学方法和生物学实验方法。将生物信息学与生物实验相结合来寻找靶基因可以很大提高准确率。 生物信息学方法主要是利用某种算法对靶基因样本进行评分及筛选。虽然预测方法各有不同, 但由于miRNA和靶基因间的作用具有一定规律性:
miRNA与其靶位点的互补性;
miRNA靶位点在不同物种之间的保守性;
miRNA-mRNA双链之间的热稳定性;
miRNA靶位点处不应有复杂二级结构;
miRNA 5 端与靶基因的结合能力强于3 端.
除此以外,不同的预测方法还会根据各自总结的规律对算法进行不同的限制与优化。为了得到更为可靠的靶基因预测,通常综合多个预测方法,取其共同预测的基因作为研究重点。目前常用的预测方法主要是以下三种:
miRanda.
miRanda 是最早的一个利用生物信息学对miRNA靶基因进行预测的软件, 由Enright等人于2003年设计开发. 作为最早的miRNA靶基因预测软件, miRanda对3-UTR的筛选依据主要是从序列匹配、miRNA与mRNA双链的热稳定性以及靶位点的保守性三个方面进行分析。基于以上三个方 面,miRanda选取每条miRNA相对的3-UTR中排名前10位的基因, 作为miRNA的候选靶基因, 对于多个miRNA对应于同一靶位点的情况, miRanda则使用贪心算法选取其中得分最高且自由能最低的那一对。
TargetScan.
TargetScan 是Lewis等在2003年开发的一款用于预测哺乳动物miRNA靶基因的软件, 该软件将RNA间相互作用的热力学模型与序列比对分析相结合, 预测不同物种间保守的miRNA结合位点. 与miRanda不同,TargetScan提出了“miRNA种子区”的概念。“miRNA种子区”是指miRNA5 端第2-8位碱基与mRNA的3-UTR完全互补所在序列。此外,它也引用了信噪比来评估预测结果的准确度. 所谓信号噪声比即用已知(信号组)和随机生成的miRNA(噪声组)分别对mRNA的3?UTR进行预测, 所得靶基因数目的比值.
PicTar.
PicTarKrek 等人在2005年开发的一款名叫组合靶位点的概率识别(probabilistic identification of combinations of target sites)的预测软件.它采用一种更先进的算法来预测脊椎动物、线虫和果蝇中miRNA的靶基因, 并通过实验方法进行了验证。与TargetScan一样, PicTar也强调“种子区”在靶位点识别及在转录后调控中的关键作用, 也注重miRNA和靶基因结合的自由能在靶基因翻译抑制中的关键作用。不同的是PicTar把种子序列分为“完全匹配种子序列”和“不完全匹配种子序 列”,前者要求种子序列和靶基因完全互补配对; 后者在满足miRNA与靶基因结合自由能不增加的前提下允许种子序列出现错配, 但不允许G U 配对.同时结合以前实验数据对两类种子序列对应的 miRNA和靶基因结合自由能进行了限制, 要求“完全 匹配种子序列”的miRNA 与靶基因结合自由能小于miRNA和靶基因最优结合能的33%; 而“不完全匹配种子序列”的miRNA与靶基因的结合自由能要求小于miRNA和靶基因最优结合能的66%, 这有效地降低了假阳性率.
miRNA靶基因的鉴定
由 于计算机模拟在预测miRNA靶基因时存在一定的局限性,利用生物学实验方法可以更加直观地寻找miRNA靶基因.目前主要是从mRNA水平与蛋白质水平 来寻找靶基因。从mRAN水平来寻找靶基因主要是通来在细胞内过表达miRNA后,利用基因芯片分析mRNA的变化以找出相应miRNA的靶基因。这种方 法由于miRNA所介导的转录后翻译抑制过程不引起mRNA水平的改变, 因此依据mRNA水平的变化寻找miRNA靶基因的方法在检出率上存在一定问题. 故采用蛋白质质谱的方法可以有效弥补这个不足.同时再结合miRNA靶基因的预测方法,可以大大提高了靶基因的检出率及可靠性。
目 前鉴定靶基因最直接的方法是, 利用荧光定量PCR及Western blot方法分别检测转染或敲低miRNA后细胞中mRNA水平及蛋白水平的变化, 从而确定miRNA与靶基因的对应关系. 这种方法可以大大提高准确率,但最终确定靶基因,还需要鉴定miRNA的靶位点. 而miRNA的靶位点的鉴定最常用的方法是荧光素酶报告基因法. 其基本原理是首先构建荧光素酶表达载体, 将希望鉴定的miRNA靶基因的3 UTR构建到荧光素酶基因的3 UTR中, 然后将荧光素酶基因表达载体转染细胞并改变细胞中相应miRNA的表达水平, 最后检测荧光素酶的表达情况以分析转染3 UTR中是否含有miRNA的靶位点。