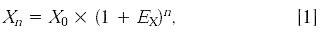利用实时定量PCR分析基因相对表达量
互联网
2575
1.4. 2-△△CT方法的数据分析
实时定量PCR所得到CT值可以很容易的输出到表格程序如Microsoft Excel中去。为了显示数据分析过程,我们在这里给出了一个基因表达定量的实验数据和样本列表。通过β-actin 均一化处理,我们对目标基因fos-glo-myc 的表达变化进行了监测。在8h 的时间范围内,在每一时间点都取3 个重复样本,每一样本在cDNA 合成之後都做定量PCR ,数据分析用到了公式9,即:
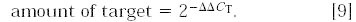
Time x 表示任意时间点,Time 0表示经β -actin 校正后1 倍量的目标基因表达。
0 时刻目标基因和内标基因的平均CT(见图2 第8 栏)被用于公式9 中。通过公式9 计算出每一个样本目标基因表达通过β -actin 均一化处理後相对于0 时刻的倍数(见图2 第9 栏)。平均SD,CV 由每一个时间点所取的三个重复样求得。用这种分析方法,在0 时刻的平均倍数变化接近于1。我们发现通过检测在 0 时刻平均倍数变化是否为1 可以很方便的验证三个重复样品之间是否有错误或者误差。如果得到的结果与1偏差很大,则表明存在计算错误或者是很高的实验误差。
在前面的例子中,在每一时间点上分别取了三个独立的 RNA 样本进行了分析。因此对每一个样本分别处理,通过计算後取结果的平均值就非常重要。如果是同一样本进行 PCR 扩增的重复,这就需要首先求出平均CT,然後再进行 计算。怎么样计算平均值就要看目标基因和内参基因是在同一个管子中扩增还是在不同的管子中扩增。表 1 给出了目标基因(c- myc )和内参基因(GAPDH )在不同管中扩增的实验数据。在这里不应该把任一单个的 c-myc 管子和 GAPDH 管子作比较,而应该分别计算出 c-myc 和 GAPDH 的平均CT 来计算△CT 。重复实验中CT值的估计偏差通过标准的指数计算转化成最後结果中相对量的变化。但其中的一个难点是 CT值与相应的拷贝数成指数关系(见第4 部分),因此,在最後的计算中,的误差通过△△CT 加上标准偏差和△△CT 减去标准偏差来评估,这就使得求得的数值相对于平均值呈不对称分布。不对称分布是因为结果经指数处理後转化成量的线性比较造成的。
通过不同荧光染料标记的探针,我们可以在同一管中同时扩增目标序列和内标序列。表2 给出了目标基因(c- myc)和内标基因(GAPDH)在同一管中扩增的实验数据。对于任意一个管子,目标基因(c- myc)和内参基因(GAPDH)扩增时加入的cDNA 量都是一样多的,所以可以分别对每个管子计算△CT 值,这些值取平均后再进行计算。 在这里估计误差值也是一个不对称的范围,反映了误差经指数处理转化为线性差异。
在表1 和表2 中,估计误差在从△CT 到△△CT 的计算中未见有增加,这是因为我们把参照基因和检测基因的误差都显示出来了。我们把△CT,cb 当作一个人为设定的常数来减去,得到△△CT 。这样得到的结果就与图 2 所显示的在求平均之前对不同重复样本分别通过各自的CT 值求实 所得结果 相当。另一种方法是将参照基因当作没有任何误差的1倍的量,在这种情况下,平均△CT,cb 的误差值被引入到每一样本的△△CT中。在表1中,肾脏中△△CT 变成- 2.50±0.20 而经过校正的c-myc 量是5.6 倍,范围从4.9 到 5.6 。而在大脑中的结果是没有误差的1倍。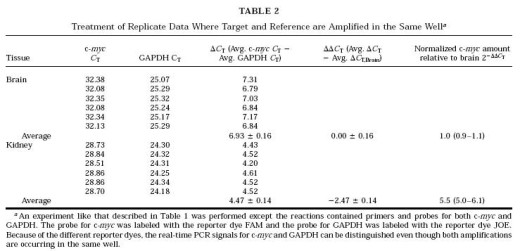

2. 2-△Cf方法
2.1. 2-△Cf方法的推导
通过内标RNA 可以对加入 RNA 的量的差异进行校正。2-△△CT方法的数据分析的一个特点就是能够利用实时PCR 实验的一部分数据来完成这种校正。在其它的方法不能定量初始 RNA 量的时候:例如,在能得到的 RNA 量非常有限的时候或者需要处理高通量的样品的时候,这一方法的优势就格外明显。当然我们也可以利用PCR 实验以外的方法来完成这种校正。最常用的一种方法就是用紫外吸收来确定用于cDNA 合成的RNA 量,然後将相同的RNA 反转录产生的cDNA 用于PCR 定量反应。这种外标法校正的一个应用例子就是研究某种实验处理是否影响内标基因的表达。在这里,目标基因和内标合二为一。在这个例子中,公式[2] 不被公式[3] 除,公式[5 ]变成:
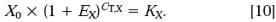
整理得: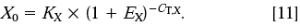 任一样品X0,q 除以参照品X0,cb 得:
任一样品X0,q 除以参照品X0,cb 得: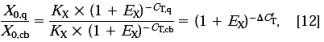
在这里△ C'T=CT,q-CT,cb 。△C’T与前面计算中用的△CT(用目标基因CT值减去参照基因CT值)相互区别。
就象在1.1部分所描述的,如果条件优化较好,效率接近于1,内标相对于参照因子为:

2.2. 2-△Cf方法的应用
2-△CT'方法的一个应用就是确定实验处理对某一候选内标基因的影响。为了显示这一过程,我们做了血清饥饿/ 诱导实验(7) 。血清饥饿/ 诱导是研究某些 mRNA 降解的常用方法(8)。然而,血清可能影响一些基因的表达包括标准的看家基因的表达。
在24-h 血清饥饿培养之後,在NIH 3T3 细胞中加入15% 血清诱导基因表达。从细胞中提取Poly(A)+ RNA ,并将之反转录成cDNA 。利用SYBR Green 通过实时定量PCR 检测GAPDH ,β 2 -microglobulin cDNA 的量。GAPDH 和β2 -microglobulin 各自的相对量通过2-△CT'公式求得。细胞处理对于GAPDH 的基因表达有明显影响,但对β2 -microglobulin 没有什么影响。因此β2 -microglobulin 很适合做血清刺激定量实验的内标,而GAPDH并不适合。这一例子向大家展示了在只研究一个基因的时候怎么用2-△CT'的方法分析基因相对表达数据。
实时PCR 最终分析的是阈值循环或CT。
CT值通过 PCR 信号的对数值和循环数来确定。因此CT值是一个指数而非线性概念。因此,在任何统计分析中都不要用原始的CT值来表示结果。正如我们在前文中所描述的一样,PCR 相对量通常和内标和参照样本一起计算而很少直接用CT值来表示,除非我们想检验重复样本之间的差别。为了向大家显示这一点,我们用SYBR Green 通过real-time PCR 来检测相同cDNA 的96 个重复反应。所有反应组分在同一管中混好後分装到 96 个管中,做实时 PCR 分析,得到了每一个样本的 C T 值。为了比较样品间变化,计算了96 个样本的平均±SD,如果通过原始CT值计算,平均 ±SD 是20.00±0.193,CV 为0.971% 。但是如果把原始CT 值用2 -CT 转化成线性形式,平均±SD是9.08 × 10-7 ±1.33 × 10-7,CV 为13.5 %。从这个简单的例子我们可以看出,通过原始CT值来反映变化是错误的,应该避免。用2-CT 将单个数据转化成线性形式来说明重复样本之间的变化和差异更准确可靠。
4. 结论
实时定量PCR 实验设计和数据分析可以采用相对定量和绝对定量两种方法,研究人员在设计实时定量PCR 实验分析基因表达的时候首先要问的一个问题就是:数据最後会以一个什么样的形式得到。如果需要知道绝对的拷贝数,就必须用绝对定量的方法,否则只需要给出基因表达相对量就足够了。相对定量可能比绝对定量要更容易一些,因为它不需要作标准曲线。
本文所给出的公式对于每个用相对定量的方法分析基因表达差异的研究人员都足够了。下面,我们总结一下实验设计和评估中的一些重要步骤:
选择一个内标基因。
确定内标的有效性,确保它不会受到实验处理的影响。
通过PCR 扩增目标基因和内标基因RNA 或cDNA 的一系列梯度稀释模板确保它们的扩增效率相同。
最後通过2-△CT 计算将统计数据转化成线性形式而不是原始CT值。
参考文献
1. Murphy, L. D., Herzog, C. E., Rudick, J. B., Fojo, A. T., and Bates, S. E. (1990) Biochemistry 29, 10351–10356.
2. Noonan, K. E., Beck, C., Holzmayer, T. A., Chin, J. E., Wunder,J. S., Andrulis, I. L., Gazdar, A. F., Willman, C. L., Griffith, B.,Von-Hoff, D. D., and Robinson, I. B. (1990) Proc. Natl. Acad. Sci. to a linear form using 2 2 C T more accurately depicts the USA 87, 7160–7164.
3. Horikoshi, T., et al. (1992) Cancer Res. 52, 108–116.
4. Heid, C. A., Stevens, J., Livak, K. J., and Williams, P. M. (1996) Genome Res. 6, 986–994.
5. Winer, J., Jung, C. K., Shackel, I., and Williams, P. M. (1999) Anal. Biochem. 270, 41–9.
6. Schmittgen, T. D., Zakrajsek, B. A., Mills, A. G., Gorn, V., Singer, M. J., and Reed, M. W. (2000) Anal. Biochem. 285, 194–204.
7. Schmittgen, T. D., and Zakrajsek, B. A. (2000) J. Biochem. Biophys.Methods 46, 69–81.
7. Schmittgen, T. D., and Zakrajsek, B. A. (2000) J. Biochem. Biophys.Methods 46, 69–81.
8. Chen, C. Y., and Shyu, A. B. (1994) Mol. Cell. Biol. 14, 8471–8482.
9. Iyer, V. R., et al. (1999) Science 283, 83–87.
10. Giulietti, A., Overbergh, L., Valckx, D., Decallone, B., Bouillon, R., and Mathieu, C. (2001) Methods 25, 386–401.
11. Niesters, H. G. M. (2001) Methods 25, 419–429.